285: ヒストン脱アセチル化酵素(Histone Deacetylases)
この記事は、定量生物医学ラトガース研究所(Rutgers Institute for Quantitative Biomedicine)が主催した学部生・大学院生向けの1週間集中合宿の一環として、ジェシカ・ダマンスキー(Jessica Damanski)、パロマ・ムンギア・サラザール(Paloma Munguia Salazar)、ミヒカ・シャー(Mihika Shah)、ラジヴ・スネイプ(Rajiv Snape)によって執筆・制作されたものです。またこの記事は、2023-2024年のPDB-101健康特集「がんに関する生物学と治療法」(health focus on "Cancer Biology and Therapeutics") の一部として発表されたものです。
ヒトのゲノムには、全身の細胞が機能するために必要な情報の多くが含まれている。しかし、細胞の種類によって必要な情報は異なることが多い。例えば、神経細胞は電位依存性イオンチャネル(voltage-gated ion channel)を作る必要があり、筋肉細胞はアクチン(actin)、ミオシン(myosin)、タイチン(titin)をたくさん作る必要がある。細胞は必要な遺伝子を選択的に発現させることで、固有の特性を獲得している。DNAへのアクセス制御の一部は、DNAがヌクレオソームの中にあるヒストン(histones in nucleosomes)にどれだけきつく巻きついているかによって行われている。ヒストンのDNAに対する親和性は、ヒストンの末端にあるアセチル基のように電荷を帯びた官能基を付加したり除去したりすることによって調整され、転写因子がどこに結合しどの遺伝子の発現を開始するのかを決めている。
脱アセチル化の管理人
ヒストン脱アセチル化酵素(histone deacetylase、HDAC)は通常核内に存在し、ヒストンの末端からアセチル基を除去する反応を触媒する。これらの酵素には構造的に特徴のある2種類のファミリーがある。ここに示すヒストン脱アセチル化酵素のファミリーは亜鉛の補酵素に依存しているが、一方サーチュイン(sirtuin)はNADの補酵素を使って化学反応を触媒する。HDACによってアセチル基が除去されると、ヒストンはより正に帯電し、負に帯電したDNAをより強く巻きつける。これによって転写因子がDNAによりアクセスしにくくなり、近くにある遺伝子の発現が抑制される。アセチル化とHDACは、p53腫瘍抑制因子(p53 tumor suppressor)や微小管(microtubule)のようなその他多くのタンパク質、さらにはポリアミドのような荷電分子の機能を調節するためにも使われる。
HDACを標的とする
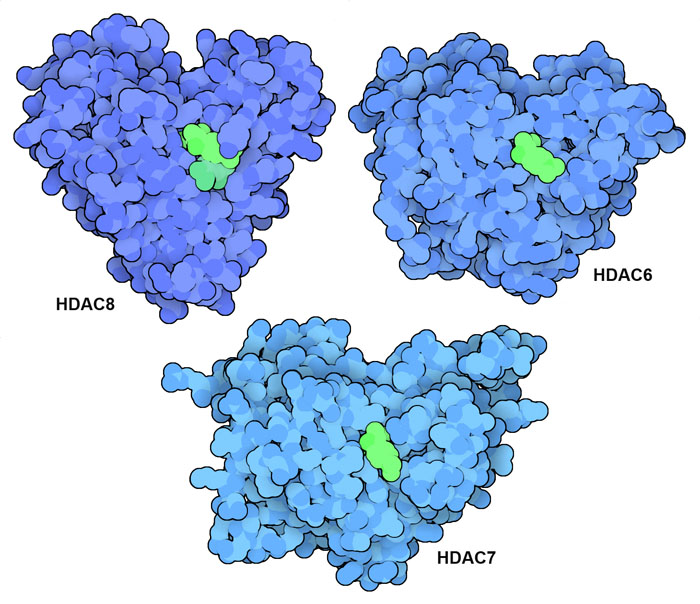
HDACは、がん遺伝子やがん抑制遺伝子の転写に影響を与えることにより、いくつかのタイプのがんにおいて発生と進行に関わっている。通常、HDACの濃度が高いこととがん患者の予後不良は相関するため、HDAC阻害剤は抗腫瘍薬の魅力的な候補となる。細菌に由来するトリコスタチンA(trichostatin A)という分子は、HDAC阻害薬開発の出発点となった。この薬剤は、HDAC8、HDAC7、HDAC6の構造(PDB ID 1t64、3c10、5edu)で示されているように、多くのHDAC活性部位に結合する。そしてその作用をより改善した抗がん剤がいくつか開発され、現在患者の治療に使われている。
チームとして働く
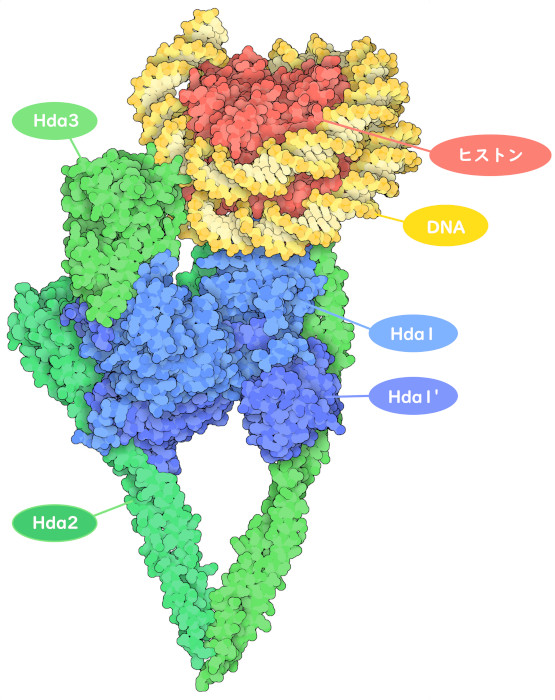
遺伝子制御に関わる多くのタンパク質と同様に、HDACも単独では働かない。細胞の種類や周囲の環境に応じて、様々な組成を持つ複数のタンパク質で構成された複合体の一部として働く。この複合体中にあるタンパク質は転写因子によって呼び出され、HDACが適切な制御部位を見つけるのを助ける。これらのタンパク質は、HDAC活性の調節、触媒活性の向上、他の酵素やタンパク質と結合するための橋渡し役としての働きなど、さまざまな役割を担っている。ここに示している酵母細胞由来の複合体(PDB ID 6z6p)には、ヒトのHDAC酵素と似たいくつかのHda酵素が含まれている。Hda2とHda3は非触媒性で、Hda1-Hda1'二量体をはさむV字型クランプをつくっている。この集合体においてHda1は活性化されており、ヒストン末端からアセチル基を除去する場所に位置している。
構造をみる
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
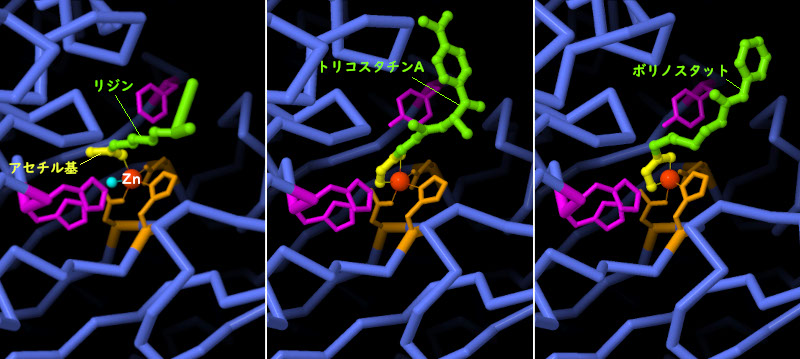
現在、様々なHDAC阻害剤が抗がん剤として使われている。それらはHDACが元々標的としているアミノ酸のアセチル化リジン(acetylated lysine)を模倣することで作用する。PDB ID 2v5wの構造は、HDAC8がどのようにリジンを認識するかを示している。亜鉛イオンはアセチル基(黄色)に配位し、脱アセチル化反応を助けるいくつかのアミノ酸(赤紫色)や水分子(水色)を分子に隣接させる。Streptomyces hygroscopicusから単離された天然物のトリコスタチンA(PDB ID 1t64)や、米国FDA(アメリカ食品医薬品局)が承認した抗がん剤ボリノスタット(Vorinostat、PDB ID 1t69)の構造で見られるように、阻害剤にはアセチル基と水の両方を模倣する化学基があって、分子を亜鉛イオンにしっかりと固定している。画像の下のボタンをクリックして対話的操作のできる画像に切り替え、これらの構造をより詳しく調べてみて欲しい。
理解を深めるためのトピックス
参考文献
- 1t64、1t69 2004 Structural snapshots of human HDAC8 provide insights into the Class I histone deacetylases. Structure 12 1325–1334
- 2v5w 2007 Substrate binding to histone deacetylases as revealed by crystal structure of Hdac8-substrate complex. EMBO Rep 8 879-884
- 3c10 2008 Crystal structure of catalytic domain of human histone deacetylase HDAC7 in complex with trichostatin A (TSA). J Biol Chem 283 11355-11363
- 5edu 2016 Crystal structure of human histone deacetylase 6 catalytic domain 2 in complex with trichostatin A. Nat Chem Biol 12 741-747
- 6z6p 2021 Structural basis for the regulation of nucleosome recognition and HDAC activity by histone deacetylase assemblies. Sci Adv 7 eabd4413
- 2012 Roles of histone deacetylases in epigenetic regulation: emerging paradigms from studies with inhibitors. Clin Epigenetics 4 5
- 2016 Histone and non-histone targets of dietary deacetylase inhibitors. Curr Top Med Chem 16 714-731
- 2016 HDACs and HDAC Inhibitors in cancer development and therapy. Cold Spring Harb Perspect Med 6 a026831
- 2017 Histone deacetylase 10 structure and molecular function as a polyamine deacetylase. Nat Comm 8 15368
- 2020 Histone deacetylases (HDACs): Evolution, specificity, role in transcriptional complexes, and pharmacological actionability. Genes (Basel) 11 556
- 2020 A short guide to histone deacetylases including recent progress on class II enzymes. Exp Mol Med 52 204–212
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









