284: ATMキナーゼとATRキナーゼ(ATM and ATR Kinases)
この記事は、定量生物医学ラトガース研究所(Rutgers Institute for Quantitative Biomedicine)が主催した学部生・大学院生向けの1週間集中合宿の一環として、カミエル・ベックリー(Kamiel Beckley)、サラ・マクギネス(Sarah McGuinness)、アシャ・ポラット(Asya Polat)、ケイラ・プエブラ(Kayla Puebla)によって執筆・制作されたものです。またこの記事は、2023-2024年のPDB-101健康特集「がんに関する生物学と治療法」(health focus on "Cancer Biology and Therapeutics") の一部として発表されたものです。
細胞が分裂するとき、DNAは完全でかつ正確に複製されるようにしなければならない。DNAは、紫外線や有毒化学物質のような環境中の危険因子によって傷つけられることがあるが、DNA複製のように正常な細胞過程においても、損傷につながる問題を起こす可能性がある。各細胞は毎日何万ものDNA損傷部位を抱えていることになる。このDNA損傷を見つけて修復することができなければ、しばしばがんにつながってしまう。私たちの細胞には、壊れたDNAを認識し、修復されるまで細胞分裂を一時停止させるチェックポイントがある。タンパク質キナーゼのATM(Ataxia-telangiectasia mutated)とATR(Ataxia telangiectasia and Rad3-related protein)は、これらのDNA修復チェックポイントにおいて重要な制御を行っている。これらのタンパク質は、免疫系の機能障害、放射線に対する感受性上昇、がんになりやすい体質といった複雑な神経変性疾患を起こす毛細血管拡張性運動失調症を研究しているときに同定された。DNA損傷が見つかると、ATMとATRは協働して細胞周期を一時的に停止させ、それを修復するタンパク質を動員する。
助けを求めるシグナル
ATMとATRはどちらもDNA損傷を細胞に知らせるが、その損傷の種類は異なっている。ATMは細胞に重大な危険をもたらす二本鎖切断に注目するが、一方ATRは一本鎖DNAを識別する。この場合、未修復の鎖が1本あるだけで細胞の死につながることがある。一本鎖領域は通常、DNAを複製するときにできるが、DNAポリメラーゼが停止すると、ヘリカーゼが二重らせんをほどき続け、危険なほど長い一本鎖区間がつくられてしまう可能性がある。一本鎖DNAは、修復の過程で切断されたDNAの末端にもできる。これは、DNAを再接続する前に末端が切り取られるからである。ATMとATRはDNAの損傷を見つけると、細胞周期の管理とDNA修復に関わる何百ものタンパク質をリン酸化し活性化する。この対象となるタンパク質には、p53腫瘍抑制因子(p53 tumor suppressor)や、相同組換え経路に関わるRAD51のようなタンパク質が含まれる。なお、相同組換え経路とは、無傷の染色体コピーを使って損傷した染色体を修復・復元する忠実度の高いDNA修復過程である。
行動への呼びかけ
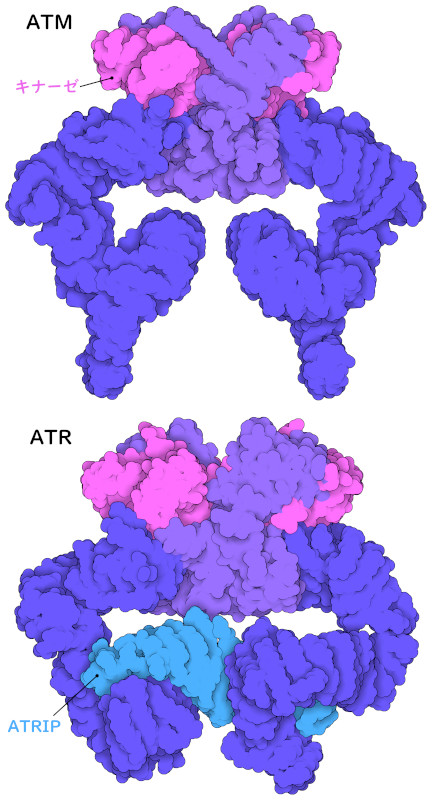
現在、ATMとATRによる信号伝達の基盤は低温電子顕微鏡によって明らかにされつつある。ヒトATMの立体構造はPDB ID 5np0で、ATRの立体構造はPDB ID 5yz0で見ることができ、一本鎖DNAに結合するタンパク質とATRの相互作用を仲介するタンパク質ATRIPと結合している。ATMとATRは同じ種類に分類されるタンパク質(同じタンパク質ファミリーのメンバー)であり、どちらもいくつかの機能部分を持つ巨大な蝶形複合体を形成している。キナーゼドメイン(赤紫色で示す部分)はリン酸化反応を行う。残りの部分にはいくつかのドメインがあり、傷ついたDNAを特異的に検知するセンサーや、リン酸化によって活性化されるさまざまなタンパク質との相互作用を仲介すると考えられている。
DNA損傷に反応するヒーローたち
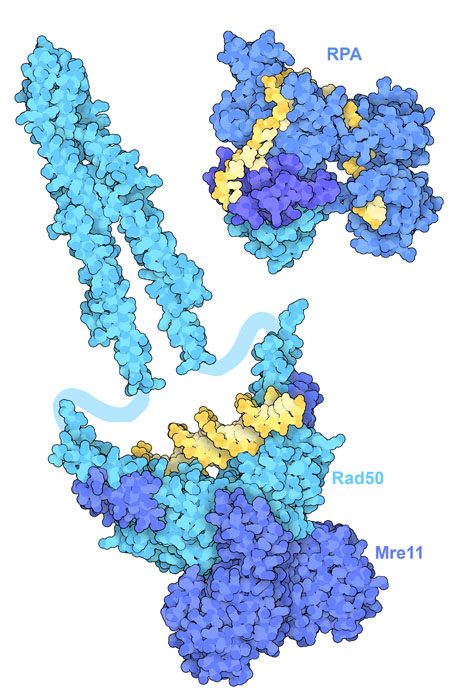
ATMとATRはDNA損傷応答における重要なメッセンジャーで、損傷したDNAを見つけ認識する上で他のタンパク質に依存している。ここでは2つの例を示す。Mre11とRad50の複合体(PDBエントリー 5dnyと5gox)は二本鎖切断に結合し、複製タンパク質A(RPA、PDBエントリー4gnx)は一本鎖DNAに結合する。これらのタンパク質は、それぞれATMとATRとの間で相互作用し、キナーゼドメインを活性化して、細胞周期と修復経路の下流にある多くのタンパク質を活性化できるようにする。
構造をみる
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
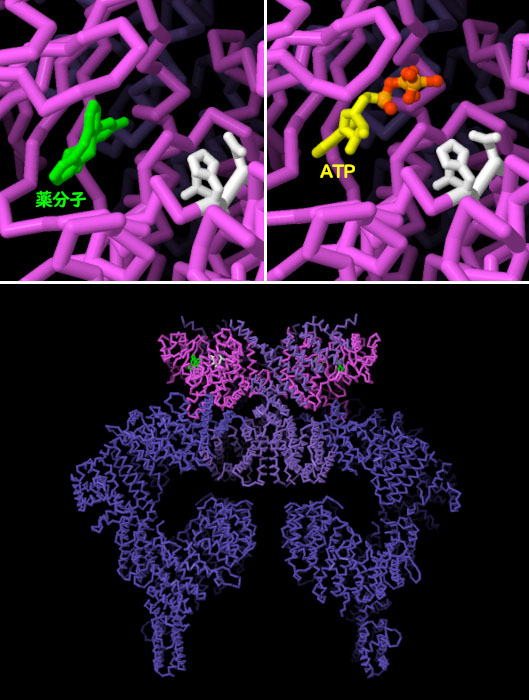
DNA修復におけるこれらの中心的役割を考えると、ATMとATRががんの発生に深く関わっていることは驚くべきことではない。がん細胞では、ゲノムが損傷した細胞の細胞分裂を止められなくなるような変異がATMやATRに生じていることが多い。研究者はこの致命的な症状を利用し、ATMを阻害する薬剤を設計して、がん治療に使えるATM阻害剤を探す試みを行っている。逆説的に聞こえるかもしれないが、これらの薬剤を腫瘍に注入することで、放射線療法が効きやすくなるかもしれない。PDBエントリー7ni5には、ATMのキナーゼドメインに結合した実験的薬剤が含まれている。薬剤分子(緑色)は、ATP(黄色、PDBエントリー 7ni6の構造)が結合するのと同じ場所、リン酸化反応を触媒する3つのアミノ酸(白色)の近くに結合する。阻害剤があると、酵素はATPと結合できず、キナーゼ活性を発揮できない。画像の下のボタンをクリックして対話的操作のできる画像に切り替え、これらの構造をより詳しく調べてみて欲しい。
理解を深めるためのトピックス
参考文献
- 2021 ATM: Main features, signaling pathways, and its diverse toles in DNA damage response, tumor suppression, and cancer development. Genes 12 845
- 7ni5、7ni6 2021 Molecular basis of human ATM kinase inhibition. Nat Struct Mol Biol 28 789–798
- 6z3a 2021 Mechanism of auto-inhibition and activation of Mec1ATR checkpoint kinase. Nat Struct Mol Biol 28 50–61
- 2020 Structures and regulations of ATM and ATR, master kinases in genome integrity. Curr Opin Struct Bio 61 98–105
- [[PDB:]] 2019 Structural basis of homologous recombination. Cell Molec Life Sci 77 3–18
- 5np0 2017 Structures of closed and open conformations of dimeric human ATM. Sci Adv 3 e1700933-e1700933
- 5gox 2017 Eukaryotic Rad50 functions as a rod-shaped dimer. Nat Struct Mol Biol 24 248-257
- 2017 Cryo-EM structure of human ATR-ATRIP complex. Cell Research 28 143–156
- 5dny 2016 ATP-dependent DNA binding, unwinding, and resection by the Mre11/Rad50 complex. EMBO J 35 743-758
- 2015 ATM and ATR as therapeutic targets in cancer. Pharmacology & therapeutics 149 124–138
- 2013 DNA damage sensing by the ATM and ATR kinases. Cold Spring Harbor Persp Biol 5 a012716
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









