285: 组蛋白脱乙酰酶(Histone Deacetylases)
这篇文章和文章中的插图是由Jessica Damanski,Paloma Munguia Salazar,Mihika Shah和Rajiv Snape撰写和制作的,作为罗格斯定量生物医学研究所(Rutgers Institute for Quantitative Biomedicine)组织的为期一周的本科生和研究生强化训练营的一部分。这也作为2023-2024年PDB-101健康重点 "癌症生物学和治疗学 "(health focus on "Cancer Biology and Therapeutics")的一部分发表。
人类基因组包含了全身细胞运作所需的大部分信息。然而,不同类型的细胞所需的信息往往不同。例如,神经细胞需要制造电压门控离子通道(voltage-gated ion channel),而肌肉细胞则需要制造大量的肌动蛋白(actin)、肌球蛋白(myosin)和肌联蛋白(titin)。细胞通过选择性地表达所需的基因来获得其独特的特性;对 DNA 存取的部分控制是基于 DNA 与核小体中的组蛋白(histones in nucleosomes)包裹的紧密程度。组蛋白与 DNA 的亲和力受带电功能基团(如组蛋白末端的乙酰基)的添加或去除的调节,而这些功能基团决定了转录因子在何处结合以及哪些基因被表达。
去乙酰化监管者
组蛋白脱乙酰酶(histone deacetylase,HDAC)通常存在于细胞核中,催化从组蛋白末端去除乙酰基的反应。这些酶有两个结构不同的家族。此处所示的组蛋白去乙酰化酶家族依赖锌辅助因子,而 长寿因子 (sirtuin)则使用 NAD 辅助因子催化化学反应。这使得转录因子更难接触到 DNA,从而抑制了附近基因的表达。乙酰化和 HDAC 还可用于调节许多其他蛋白质的功能,如 p53肿瘤抑制基因 (p53 tumor suppressor) 和 微管 (microtubule),甚至带电分子,如聚酰胺。
瞄准 HDAC
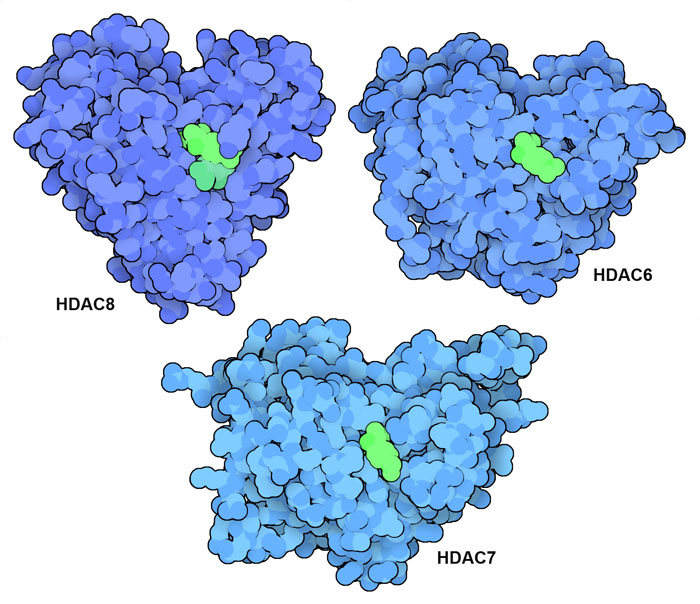
HDAC 通过影响致癌基因和抑癌基因的转录,参与多种癌症的产生和发展。高含量 HDAC 通常与癌症患者的不良预后相关,因此 HDAC 抑制剂成为抗肿瘤药物的理想候选药物。从细菌中提取的分子曲古抑菌素A(trichostatin A)是开发 HDAC 抑制剂的起点。正如 HDAC8、HDAC7 和 HDAC6(PDB ID 1t64、3c10 和 5edu)的结构所示,这种药物能与许多 HDAC 活性位点结合。几种抗癌药物可改善曲古抑菌素 A 的作用,目前已用于治疗患者。
团队合作
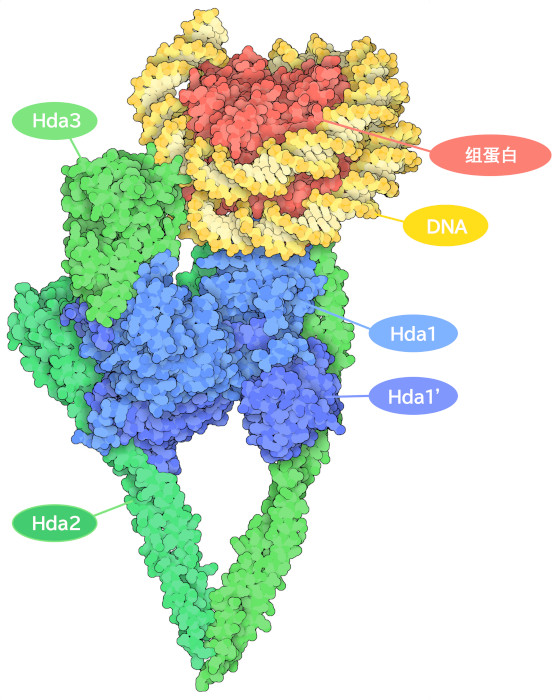
与许多参与基因调控的蛋白质一样,HDAC 也不是单独工作的。根据细胞类型和周围环境的不同,它们是由多种蛋白质组成的复合体的一部分。该复合体中的蛋白质由转录因子调用,转录因子帮助 HDAC 找到适当的调控位点。这些蛋白质发挥着多种作用,包括调节 HDAC 的活性、增强催化活性以及充当与其他酶和蛋白质结合的桥梁。图中所示的酵母细胞衍生复合物(PDB ID 6z6p)含有几种与人类 HDAC 酶类似的 Hda 酶;Hda2 和 Hda3 是非催化型,在 Hda1-Hda1' 二聚体上形成一个 V 形钳夹。在此复合体中,Hda1 被激活,并位于从组蛋白末端去除乙酰基的位置。
探索结构
要切换到有互动控制的页面,请点击图表下面的按钮。如果加载没有开始,请尝试点击图表。
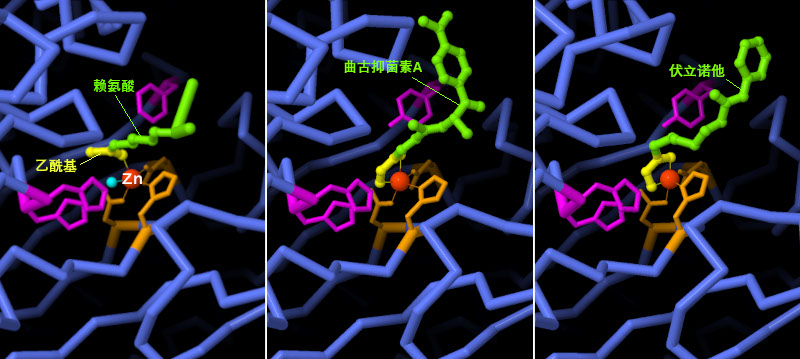
目前有多种 HDAC 抑制剂被用作抗癌药物。它们通过模拟氨基酸乙酰化赖氨酸(acetylated lysine)起作用,该氨基酸是 HDAC 的原始靶标;PDB ID 2v5w结构显示了 HDAC8 如何识别赖氨酸。锌离子与乙酰基(黄色)配位,与其相邻的一些氨基酸(红紫色)和水分子(浅蓝色)有助于去乙酰化反应,如曲古抑菌素A(PDB ID 1t64),一种从透明链霉菌(Streptomyces hygroscopicus)中分离出来的天然产物,也是美国 FDA(美国食品药品监督管理局)批准的抗癌药物伏立诺他(Vorinostat,PDB ID 1t69),这种抑制剂具有模拟乙酰基和水的化学基团,可将分子牢牢地固定在锌离子上。点击图片下方的按钮,切换到可操作的交互式图片,详细了解这些结构。
进一步的讨论议题
参考文献
- 1t64、1t69 2004 Structural snapshots of human HDAC8 provide insights into the Class I histone deacetylases. Structure 12 1325–1334
- 2v5w 2007 Substrate binding to histone deacetylases as revealed by crystal structure of Hdac8-substrate complex. EMBO Rep 8 879-884
- 3c10 2008 Crystal structure of catalytic domain of human histone deacetylase HDAC7 in complex with trichostatin A (TSA). J Biol Chem 283 11355-11363
- 5edu 2016 Crystal structure of human histone deacetylase 6 catalytic domain 2 in complex with trichostatin A. Nat Chem Biol 12 741-747
- 6z6p 2021 Structural basis for the regulation of nucleosome recognition and HDAC activity by histone deacetylase assemblies. Sci Adv 7 eabd4413
- 2012 Roles of histone deacetylases in epigenetic regulation: emerging paradigms from studies with inhibitors. Clin Epigenetics 4 5
- 2016 Histone and non-histone targets of dietary deacetylase inhibitors. Curr Top Med Chem 16 714-731
- 2016 HDACs and HDAC Inhibitors in cancer development and therapy. Cold Spring Harb Perspect Med 6 a026831
- 2017 Histone deacetylase 10 structure and molecular function as a polyamine deacetylase. Nat Comm 8 15368
- 2020 Histone deacetylases (HDACs): Evolution, specificity, role in transcriptional complexes, and pharmacological actionability. Genes (Basel) 11 556
- 2020 A short guide to histone deacetylases including recent progress on class II enzymes. Exp Mol Med 52 204–212
 生物大分子学习的教育门户
生物大分子学习的教育门户 









