283: c-Ablタンパク質キナーゼとイマチニブ(c-Abl Protein Kinase and Imatinib)
この記事は、定量生物医学ラトガース研究所(Rutgers Institute for Quantitative Biomedicine)が主催した学部生・大学院生向けの1週間集中合宿の一環として、アリソン・エイベル(Allison Abel)、ダーリーン・R・マラベ=ラモス(Darlene R. Malave-Ramos)、バヴヤ・ソーニ(Bhavya Soni)、クリストファー・タイ(Christopher Thai)、エイミー・ウーウー(Amy Wu-Wu)によって執筆・制作されたものです。またこの記事は、2023-2024年のPDB-101健康特集「がんに関する生物学と治療法」(health focus on "Cancer Biology and Therapeutics") の一部として発表されたものです。
タンパク質キナーゼ(protein kinase、タンパク質リン酸化酵素)は当初、がん化学療法の標的としては敬遠されていた。それらは細胞機能のほぼすべての側面において重要な役割を果たし、厳密に制御されていて、その制御に何らかの変化が生じると、がんやその他の疾患の発症につながるため、優れた標的となることがよく理解されていた。しかしながら、細胞内には多くの種類のキナーゼが存在し、それらはすべて構造的に類似しており、ATPとの結合部位も類似している。このような類似性から、これらのキナーゼの活性部位を標的とする薬剤は、必然的に標的とするもの以外への結合を引き起こし、その結果副作用が生じると長い間考えられてきた。
ドアを開ける構造
幸いなことに、構造生物学はこのような当初の推測が誤りであることを証明してくれた。タンパク質キナーゼの構造が解明されるにつれ、そのATP結合ポケットには十分な違いがあることがわかり、構造誘導による選択的な薬剤の発見と開発が容易になったのである。抗がん剤のイマチニブ(imatinib)は画期的な成功事例となり、それ以来、さまざまなタンパク質キナーゼに対する選択的低分子阻害剤の開発に成功した。その多くが構造誘導創薬の成果によるものである。
構造は定まらないけど機能不全ではない
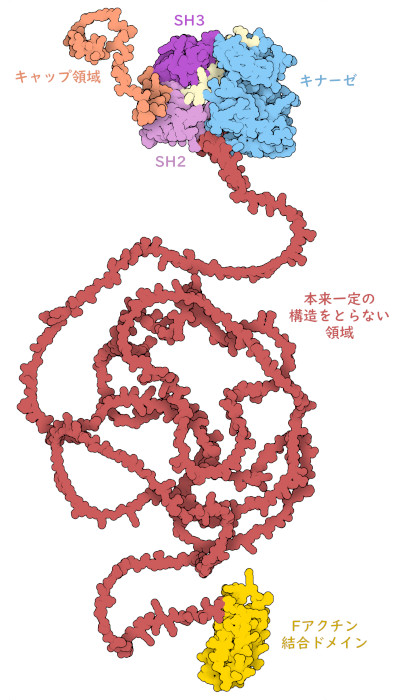
イマチニブが標的とするc-Ablは、ATPからチロシンへと選択的にリン酸基を転移するチロシンキナーゼである。計算によって求められたタンパク質全長のモデル(PDBエントリーAF_AFP00519F1)で見られるように、c-Ablはいくつかの機能部分を持った複合体である。キナーゼドメインおよび制御ドメイン(kinase and regulatory domains、PDBエントリー1opl)とF-アクチン結合ドメイン(F-actin binding domain、PDBエントリー1zzp)という2つの領域は、安定した折りたたみ構造を持っていて、X線結晶構造解析法とNMR分光法を使って詳細に研究されている。これらは長くて本来一定の構造をとらない部分を介して連結されているが、依然高い柔軟性を持っている。そして一端にはキャップ領域と呼ばれる短い柔軟な尾部があって、ミリストイル基(myristoyl group)と結合している。キャップ領域はタンパク質のロック機構として働き、そのタンパク質の機能を制御する。不活性状態にあるときは、このミリストイル基がキナーゼドメインにあるポケットに結合し、キナーゼドメインと調節ドメインを強固に結合させ、触媒活性を阻害する構造変化を引き起こす。タンパク質を活性化するには、外部からの細胞信号がc-Ablの制御ドメインと相互作用し、キナーゼドメインからミリストイル基が遊離し、活性部位を露出させて、特定の基質をリン酸化できるようにする。
がんの組み合わせ
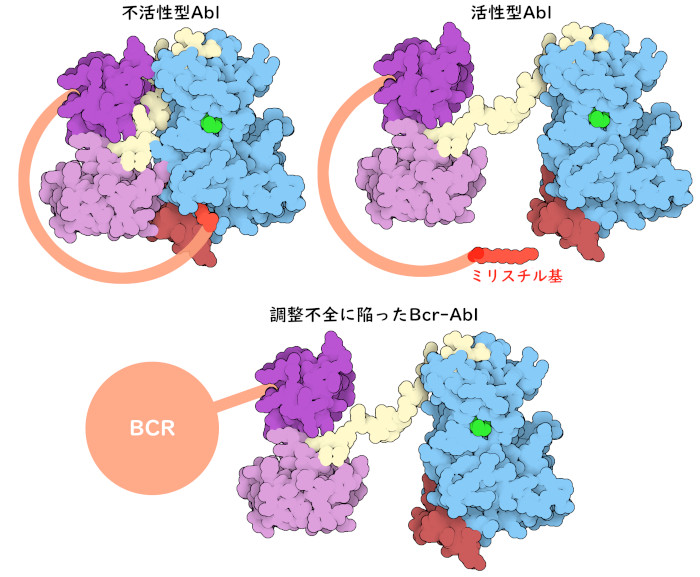
c-Ablチロシンキナーゼに影響を及ぼす遺伝子異常は、骨髄にある未熟細胞のがんである慢性骨髄性白血病(chronic myelogenous leukemia、CML)に関連している。CMLの原因はフィラデルフィア染色体(Philadelphia chromosome)として知られる遺伝子異常で、9番染色体の一部が22番染色体の一部と入れ替わることで形成される。このバランスのとれた遺伝子転座によってc-abl遺伝子とbcr遺伝子が融合し、Bcr-Ablと呼ばれる別のタンパク質がつくられる。この変異型チロシンキナーゼは自己抑制性キャップ領域とミリストイル基を持たないため、常に活性があり、通常の細胞信号による活性化を必要としない。これにより、Bcr-Ablは細胞分裂をし続けることができる。Bcr-Ablはまた、がん細胞の蓄積を防ぐために細胞が行うアポトーシス(apoptosis、自ら死を誘導する細胞に備わった過程)も阻止する。Bcr-Ablにの作用によって未成熟で部分的に機能する血液細胞が蓄積されることにより、CMLと診断された患者に壊滅的な影響を及ぼし、最終的には急性転化期(blast phase)と呼ばれる急性白血病状態(acute leukemic condition)へと進行する。イマチニブや、米国食品医薬品局(United States Food and Drug Administration)が承認したこの他のBcr-Abl阻害剤による治療により、10年生存率は約85%と劇的に改善した。現在、CML患者の多くは、健康な成人とほぼ同じ寿命を生きることができるようになった。
構造をみる
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
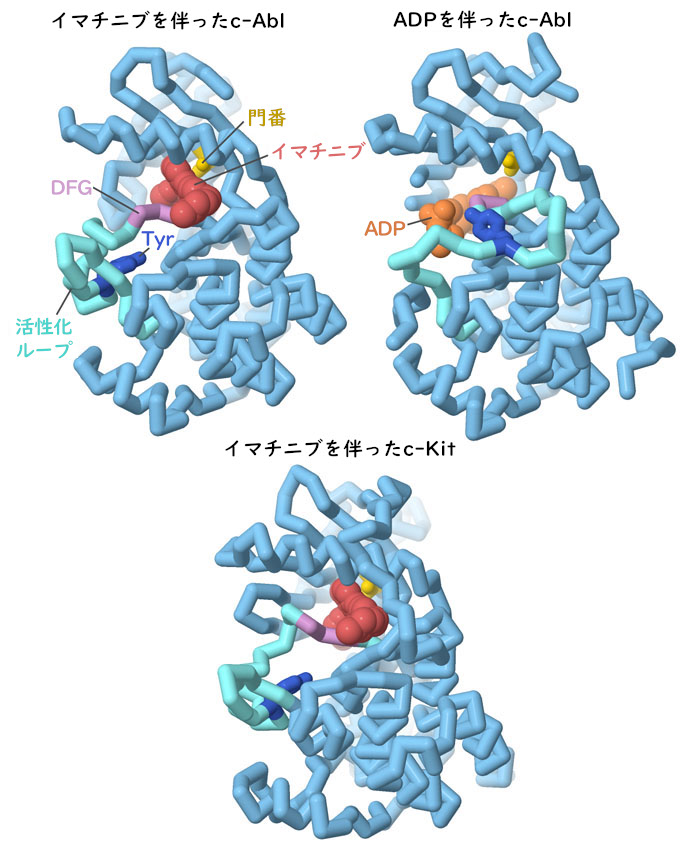
イマチニブはc-Ablが持つキナーゼドメインのATP結合部位を塞ぎ、その作用を停止させる。ADP(PDBエントリー2g2i)とイマチニブ(PDBエントリー2hyy)を含めたキナーゼの構造で見られるように、キナーゼにあるいくつかの領域が、その機能とイマチニブによる阻害において重要である。「活性化ループ」(activation loop)は活性部位を覆ってふたをし、活性部位へのアクセスを制御する。またリン酸化されるチロシンによって制御される。「門番」となるスレオニン、「DFGモチーフ」、それにいくつかの水素結合アミノ酸(ここには示していない)は、イマチニブとの特異的相互作用に関与し、薬物の選択性を高めるのに役立っている。ただ、イマチニブは完全な選択性を持っているわけではないが、幸いなことにこれを利用できる場合がある。イマチニブは消化管間質腫瘍(gastrointestinal stromal tumor、GIST)を引き起こすがんタンパク質チロシンキナーゼの一種c-Kit(PDBエントリー1t46)も阻害する。c-Kitとc-Ablは近縁のキナーゼであり、イマチニブは両方に効く。図の下のボタンをクリックして対話的操作のできる画像に切り替え、これらの構造をさらに詳しく調べてみて欲しい。
理解を深めるためのトピックス
- キナーゼドメインと制御ドメインを詳しく見るには、RCSB PDBの注釈付き視覚化サイト3D Protein Structure Viewを参照してみて欲しい。
- イマチニブは、造血細胞において免疫受容体の信号伝達に関与する脾臓チロシンキナーゼ(spleen tyrosine kinase、Syk)を含む、ある一定配位のキナーゼに対して効果を示す。イマチニブが非常にコンパクトなU字型の形状をとってSykを阻害していることがPDBエントリー1xbbの構造で確認できる。
参考文献
- 2021 Kinase drug discovery 20 years after imatinib: progress and future directions. Nat Rev Drug Discovery 20 551–569
- 2020 Impact of the Protein Data Bank on antineoplastic approvals. Drug Discovery Today 25 837–850
- 2017 Structural and functional dissection of the DH and PH domains of oncogenic Bcr-Abl tyrosine kinase. Nat Commun 8 1–14
- 2013 Conformation-selective inhibitors reveal differences in the activation and phosphate-binding loops of the tyrosine kinases Abl and Src. ACS Chem Biol 8 2734–2743
- 2HYY 2006 Structural biology contributions to the discovery of drugs to treat chronic myelogenous leukaemia. Acta Cryst D Biol Crystallog 63 80–93
- 2G2I 2006 A src-like inactive conformation in the ABL tyrosine kinase domain. PLoS Biol 4 e144
- 2006 Organization of the SH3-SH2 unit in active and inactive forms of the c-Abl tyrosine kinase. Molecular Cell 21 787–798
- 1ZZP 2005 Structural basis for the cytoskeletal association of Bcr-Abl/c-Abl. Mol Cell 19 461–473
- 1XBB 2004 A novel mode of Gleevec binding is revealed by the structure of spleen tyrosine kinase. J Biol Chem 279 55827–55832
- 1T46 2004 Structural basis for the autoinhibition and STI-571 inhibition of c-kit tyrosine kinase. J Biol Chem 279 31655–31663
- 1OPL 2003 Structural basis for the autoinhibition of c-Abl tyrosine kinase. Cell 112 859–871
- 2002 Structure of the Bcr-Abl oncoprotein oligomerization domain. Nat Struct Biol 9 117–120
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









