284: ATR和ATR激酶(ATM and ATR Kinases)
這篇文章和文章中的插圖是由Kamiel Beckley, Sarah McGuinness, Asya Polat和Kayla Puebla撰寫和製作的,作為羅格斯定量生物醫學研究所(Rutgers Institute for Quantitative Biomedicine)組織的為期一周的本科生和研究生強化訓練營的一部分。這也作為2023-2024年PDB-101健康重點"癌症生物學和治療學"(health focus on "Cancer Biology and Therapeutics")的一部分發表。
細胞分裂時,必須確保DNA 複製的完整和準確。 DNA可能會受到紫外線輻射和有毒化學物質等環境危害的破壞,但即使是正常的細胞過程,如DNA複製,也可能出現問題導致DNA受損。每個細胞每天都會有數以萬計的DNA損傷位點。未能發現並修復這種DNA 損傷通常會導致癌症。我們的細胞有檢查點,可以識別受損的DNA 並暫停細胞分裂,直到修復為止。蛋白激酶ATM(Ataxia-telangiectasia mutated,共濟失調毛細血管擴張症突變蛋白)和ATR(Ataxia telangiectasia and Rad3-related protein,共濟失調毛細血管擴張症和Rad3相關蛋白)是這些DNA修復檢查點的重要調節因子。毛細血管擴張症是一種複雜的神經退化性疾病,會導致免疫系統功能失調、對輻射敏感性增加和易患癌症。當發現DNA 損傷時,ATM 和ATR 會一起暫時停止細胞週期,並調動蛋白質來修復損傷。
求救訊號
ATM 和ATR 會根據損傷類型的不同,向細胞發出DNA 受損訊號:ATM 專注於對細胞造成嚴重危害的雙股斷裂,而ATR 則辨識單股DNA。在這種情況下,只要有一條未修復的鏈就會導致細胞死亡。單股區域通常是在DNA 複製時產生的,但如果DNA 聚合酶停止運作,螺旋酶就會繼續解開雙螺旋,產生危險的長單股部分。在修復過程中,斷裂的DNA末端也會產生單股DNA。當ATM和ATR發現DNA損傷時,它們會磷酸化並激活數百種參與細胞週期管理和DNA修復的蛋白質。這些標靶蛋白包括p53腫瘤抑制基因 (p53 tumor suppressor)和參與同源重組途徑的RAD51 等蛋白。請注意,同源重組途徑是一種高保真DNA修復過程,它使用完整的染色體拷貝來修復和恢復受損染色體。
呼籲採取行動
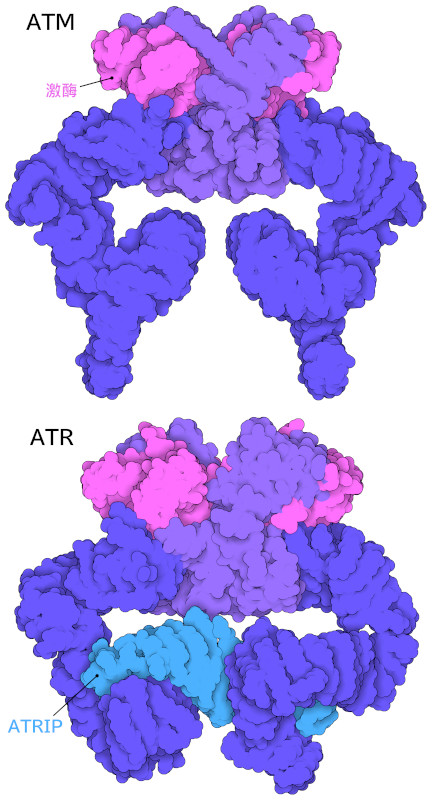
ATM和ATR訊號傳導的基礎資訊現在透過冷凍電子顯微鏡得以揭示。在PDB ID 5np0 中可以看到人類ATM 的三維結構,在PDB ID 5yz0 中可以看到ATR 的三維結構,ATR 與蛋白ATRIP 結合,ATRIP是一種有助於介導ATR與單股DNA結合的蛋白質相互作用的蛋白質。 ATM 和ATR 是同一蛋白質家族的成員,都形成具有多個功能部分的巨大蝴蝶形複合物。激酶結構域(此處以洋紅色顯示)執行磷酸化反應。該蛋白質的其餘部分有幾個結構域,這些結構域被認為介導與受損DNA的特定感測器以及透過磷酸化活化的多種蛋白質的相互作用。
對DNA損傷做出反應的英雄
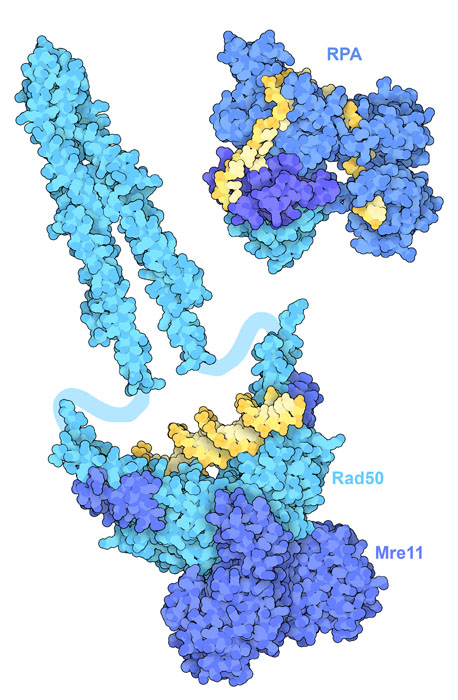
ATM 和ATR 是DNA 損傷反應中的關鍵信使。它們依靠其他蛋白質來尋找和識別受損的DNA。這裡舉兩個例子:Mre11 和Rad50(PDB 條目5dny 和5gox)的複合物與斷裂的雙股結合,而複製蛋白ARPA,PDB 條目4gnx)與單股DNA結合。這些蛋白分別與ATM 和ATR 相互作用,激活激酶結構域,從而激活細胞週期和修復途徑下游的許多蛋白。
探索結構
若要切換到有互動控制的頁面,請點擊圖表下方的按鈕。如果載入沒有開始,請嘗試點擊圖表。
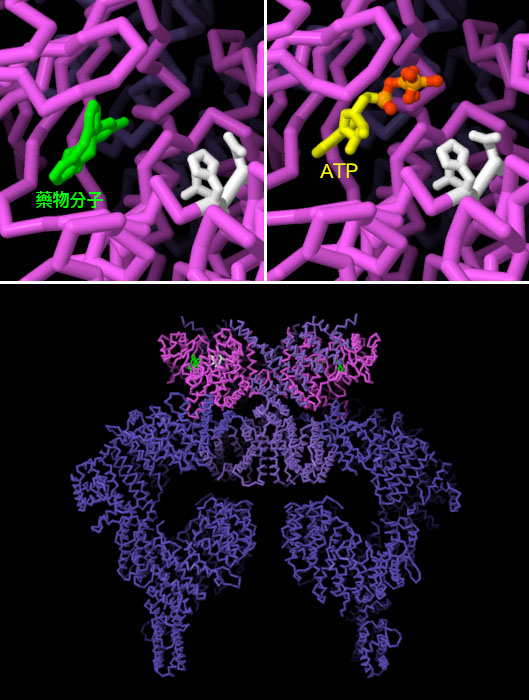
鑑於ATM 和ATR 在DNA 修復中的核心作用,它們深度參與癌症的發展也就不足為奇了。在癌細胞中,ATM 和ATR 經常發生突變,導致基因組受損的細胞無法停止細胞分裂。研究人員正試圖利用這種致命的情況來設計抑制ATM的藥物,試圖找到可用於治療癌症的ATM抑制劑。雖然聽起來有些矛盾,但將這些藥物注射到腫瘤中可能會使放射治療更有效。 PDB 條目7ni5 包含一種與ATM 激酶結構域結合的實驗性藥物。藥物分子(綠色)與ATP(黃色,PDB 條目7ni6的結構)結合的位置相同,靠近催化磷酸化反應的三個胺基酸(白色)。在有抑制劑的情況下,酵素無法與ATP 結合,也無法發揮激酶活性。點擊圖像下方的按鈕,切換到可操作的互動式圖像,詳細查看這些結構。
進一步的討論議題
參考文獻
- 2021 ATM: Main features, signaling pathways, and its diverse toles in DNA damage response, tumor suppression, and cancer development. Genes 12 845
- 7ni5、7ni6 2021 Molecular basis of human ATM kinase inhibition. Nat Struct Mol Biol 28 789–798
- 6z3a 2021 Mechanism of auto-inhibition and activation of Mec1ATR checkpoint kinase. Nat Struct Mol Biol 28 50–61
- 2020 Structures and regulations of ATM and ATR, master kinases in genome integrity. Curr Opin Struct Bio 61 98–105
- [[PDB:]] 2019 Structural basis of homologous recombination. Cell Molec Life Sci 77 3–18
- 5np0 2017 Structures of closed and open conformations of dimeric human ATM. Sci Adv 3 e1700933-e1700933
- 5gox 2017 Eukaryotic Rad50 functions as a rod-shaped dimer. Nat Struct Mol Biol 24 248-257
- 2017 Cryo-EM structure of human ATR-ATRIP complex. Cell Research 28 143–156
- 5dny 2016 ATP-dependent DNA binding, unwinding, and resection by the Mre11/Rad50 complex. EMBO J 35 743-758
- 2015 ATM and ATR as therapeutic targets in cancer. Pharmacology & therapeutics 149 124–138
- 2013 DNA damage sensing by the ATM and ATR kinases. Cold Spring Harbor Persp Biol 5 a012716
 生物大分子學習的教育門戶
生物大分子學習的教育門戶 









