284: ATM 및 ATR 키나제 (ATM and ATR Kinases)
이 기사는 정량생물의학 럿거스연구소(Rutgers Institute for Quantitative Biomedicine)가 주최한 학부생 및 대학원생을 위한 1주일 집중 합숙의 일환으로 카미엘 베클리(Kamiel Beckley), 사라 맥기네스(Sarah McGuinness), 아샤 폴랫(Asya Polat), 케일라 푸에블라(Kayla Puebla)가 집필 및 제작한 것입니다. 또한 이 기사는 2023~2024년의 PDB-101 건강특집 ‘암에 관한 생물학과 치료법’(health focus on “Cancer Biology and Therapeutics”)의 일부로 발표된 것입니다.
세포가 분열될 때 DNA는 완전하고 정확하게 복제되도록 해야 한다. DNA는 자외선이나 유독화학물질과 같은 환경 속의 위험인자에 의해 손상될 수 있지만 DNA 복제처럼 정상적인 세포 과정에서도 손상으로 이어지는 문제를 일으킬 수 있다. 각 세포들은 매일 수만 개의 DNA 손상 부위를 안고 있는 셈이다. 이 DNA 손상을 발견하여 복구하지 못하면 종종 암으로 이어져버린다. 우리 세포에는 파괴된 DNA를 인식하고 복구될 때까지 세포 분열을 일시 정지시키는 체크 포인트가 있다. 단백질 키나아제의 ATM(Ataxia-telangiectasia mutated)과 ATR(Ataxia telangiectasia and Rad3-related protein)은 이들의 DNA 복구 체크 포인트에서 중요한 제어를 수행하고 있다. 이 단백질들은 면역계 기능장애, 방사선에 대한 감수성 상승, 암에 걸리기 쉬운 체질과 같은 복잡한 신경변성질환을 일으키는 모세혈관 확장성 운동실조증을 연구할 때 동정(同定)되었다. DNA 손상이 발견되면 ATM과 ATR은 협동해 세포 주기를 일시적으로 정지시키고 이를 복구하는 단백질을 동원한다.
도움을 요청하는 신호
ATM과 ATR은 모두 DNA 손상을 세포에 알리지만 그 손상의 종류는 다르다. ATM은 세포에 중대한 위험을 초래하는 이중가닥 절단에 주목하지만 ATR은 단일가닥 DNA를 식별한다. 이 경우 미복구 사슬이 1개만 있어도 세포의 죽음으로 이어질 수 있다. 단일가닥 영역은 보통 DNA를 복제할 때 생기지만 DNA 중합효소가 정지하면 헬리카제가 이중 나선을 계속 풀어 위험할 정도로 긴 단일가닥 사슬 구간이 만들어져버릴 가능성이 있다. 단일가닥 사슬 DNA는 복구 과정에서 절단된 DNA 말단에도 생긴다. 이는 DNA를 재접속하기 전에 말단이 잘려 나가기 때문이다. ATM과 ATR은 DNA 손상을 발견하면 세포 주기 관리와 DNA 복구에 관련된 수백 개의 단백질을 인산화하고 활성화한다. 이 대상이 되는 단백질에는 p53 종양억제인자 (p53 tumor suppressor)나 상동 재조합 경로와 관련된 RAD51과 같은 단백질이 포함된다. 또한 상동 재조합 경로란 손상되지 않은 염색체 복제를 사용하여 손상된 염색체를 복구 및 복원하는 충실도 높은 DNA 복구 과정이다.
행동에 대한 호소
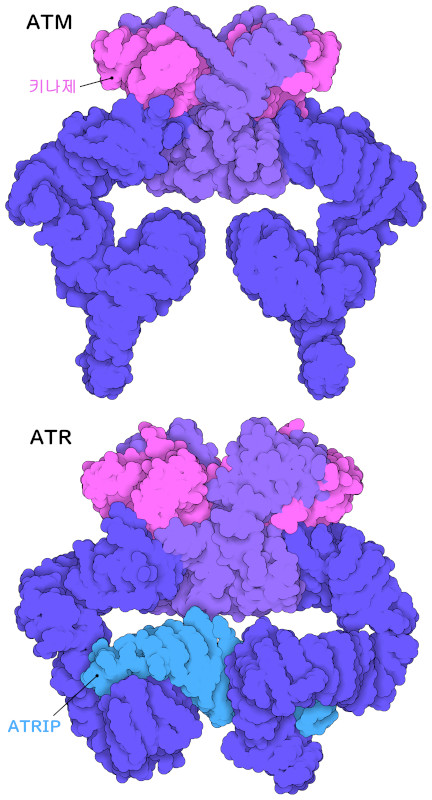
현재 ATM과 ATR에 의한 신호전달 기반은 저온 전자현미경에 의해 밝혀지고 있다. 인간 ATM의 입체구조는 PDB ID 5np0이고 ATR의 입체구조는 PDB ID 5yz0에서 볼 수 있으며 단일가닥 DNA에 결합하는 단백질과 ATR의 상호 작용을 중개하는 단백질 ATRIP와 결합되어 있다. ATM과 ATR은 같은 종류로 분류되는 단백질(같은 단백질 패밀리의 멤버)이며, 양쪽 모두 몇 가지 기능 부분을 갖는 거대한 나비형 복합체를 형성하고 있다. 키나아제 도메인(자홍색으로 나타낸 부분)은 인산화 반응을 수행한다. 나머지 부분에는 몇 개의 도메인이 있어 손상된 DNA를 특이적으로 감지하는 센서와 인산화에 의해 활성화되는 다양한 단백질과의 상호 작용을 중개하는 것으로 여겨진다.
DNA 손상에 반응하는 영웅들
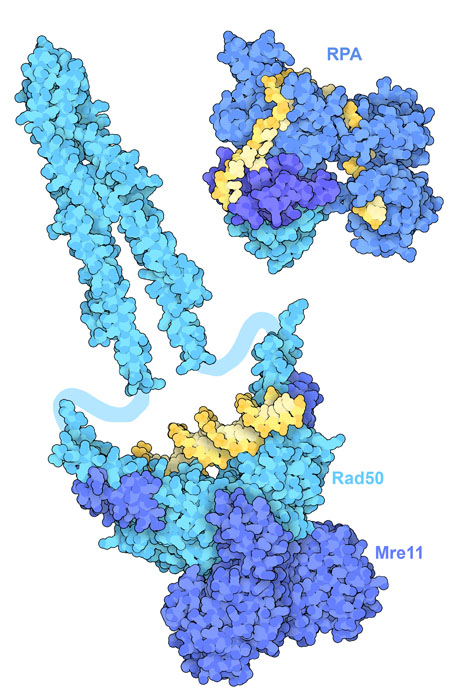
ATM과 ATR은 DNA 손상 응답의 중요한 메신저로, 손상된 DNA를 찾아 인식하는 데 다른 단백질에 의존하고 있다. 여기서는 두 가지 예를 나타낸다. Mre11과 Rad50의 복합체(PDB 엔트리 5dny와 5gox)는 이중가닥 절단에 결합하고 복제 단백질A(RPA, PDB 엔트리 4gnx)는 단일가닥 DNA에 결합한다. 이 단백질들은 각각 ATM과 ATR 사이에서 상호 작용을 하고 키나아제 도메인을 활성화하여 세포 주기와 복구 경로의 하류에 있는 많은 단백질을 활성화할 수 있도록 한다.
구조 보기
인터랙티브 조작이 가능한 페이지로 전환하려면 그림 아래의 버튼을 클릭하십시오. 읽기가 시작되지 않을 때는 그림을 클릭하십시오.
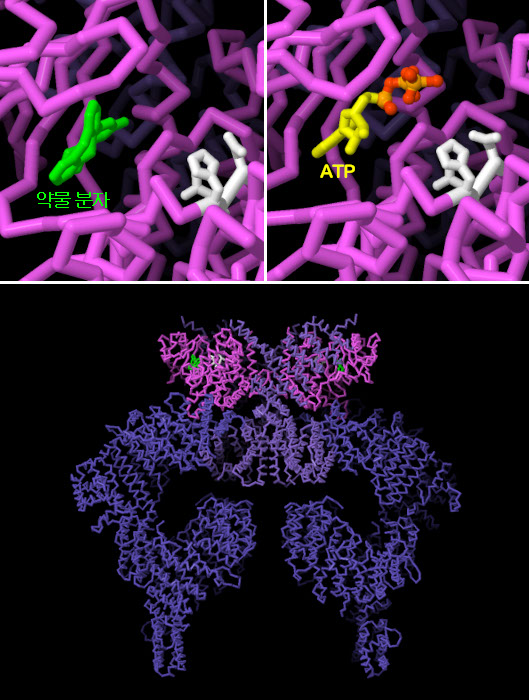
DNA 복구에 있어서 이들의 중심적 역할을 고려할 때 ATM과 ATR이 암의 발생에 깊이 관여하고 있다는 것은 놀라운 일이 아니다. 암세포에서는 게놈이 손상된 세포의 세포분열을 멈출 수 없게 되는 변이가 ATM이나 ATR에 생기는 경우가 많다. 연구자들은 이 치명적인 증상을 이용하여 ATM을 저해하는 약제를 설계하고, 암 치료에 사용할 수 있는 ATM 저해제를 찾는 시도를 하고 있다. 역설적으로 들릴지 모르지만 이들 약제를 종양에 주입함으로써 방사선 요법이 효과가 더 좋아질 수 있다. PDB 엔트리 7ni5에는 ATM의 키나아제 도메인에 결합한 실험적 약제가 포함되어 있다. 약제분자(녹색)는 ATP(노란색, PDB 엔트리 7ni6 구조)가 결합하는 것과 같은 장소, 인산화 반응을 촉매하는 3개 아미노산(흰색) 근처에 결합한다. 저해제가 있으면 효소는 ATP와 결합할 수 없어 키나아제 활성을 발휘할 수 없다. 이미지 아래의 버튼을 클릭하여 인터랙티브로 조작할 수 있는 이미지로 전환하여 이러한 구조를 보다 자세하게 조사해 보기 바란다.
이해를 높이기 위한 토픽
참고문헌
- 2021 ATM: Main features, signaling pathways, and its diverse toles in DNA damage response, tumor suppression, and cancer development. Genes 12 845
- 7ni5、7ni6 2021 Molecular basis of human ATM kinase inhibition. Nat Struct Mol Biol 28 789–798
- 6z3a 2021 Mechanism of auto-inhibition and activation of Mec1ATR checkpoint kinase. Nat Struct Mol Biol 28 50–61
- 2020 Structures and regulations of ATM and ATR, master kinases in genome integrity. Curr Opin Struct Bio 61 98–105
- [[PDB:]] 2019 Structural basis of homologous recombination. Cell Molec Life Sci 77 3–18
- 5np0 2017 Structures of closed and open conformations of dimeric human ATM. Sci Adv 3 e1700933-e1700933
- 5gox 2017 Eukaryotic Rad50 functions as a rod-shaped dimer. Nat Struct Mol Biol 24 248-257
- 2017 Cryo-EM structure of human ATR-ATRIP complex. Cell Research 28 143–156
- 5dny 2016 ATP-dependent DNA binding, unwinding, and resection by the Mre11/Rad50 complex. EMBO J 35 743-758
- 2015 ATM and ATR as therapeutic targets in cancer. Pharmacology & therapeutics 149 124–138
- 2013 DNA damage sensing by the ATM and ATR kinases. Cold Spring Harbor Persp Biol 5 a012716
 의 생체 고분자 학습 포털 사이트
의 생체 고분자 학습 포털 사이트 









