262: PDB構造への50年間のオープンアクセス(Fifty Years of Open Access to PDB Structures)
蛋白質構造データバンク(Protein Data Bank)は、自分たちの研究成果を共有したいと考えた結晶学者たちによるコミュニティ主導の取り組みとして始まった。そして、国際蛋白質構造データバンク(Worldwide PDB)のメンバー組織が協力して運営し、世界中の研究室から提供され厳選された生体分子構造に誰でも自由にアクセスできることをを保証している世界的アーカイブへと成長した。過去50年間、PDBアーカイブは、私たちの健康を改善するのに効果的な医薬品の見つけたり、タンパク質が機能する構造へとどのように折りたたまれるのかを予測するなど多くの分野で、成長と革新を促してきた。PDBにある情報はまた、生物学と進化の基本的なしくみに関する重要な原子レベルでの理解につながり、研究や教育に使われている。サービスが始まって50年という記念すべき年を祝うため、PDBアーカイブに登録されている数多くの魅力的な構造を年代ごとに選んだ。この情報を元にPDBアーカイブを探索し、各自のお気に入りを見つけて欲しい。
構造生物学の始まり
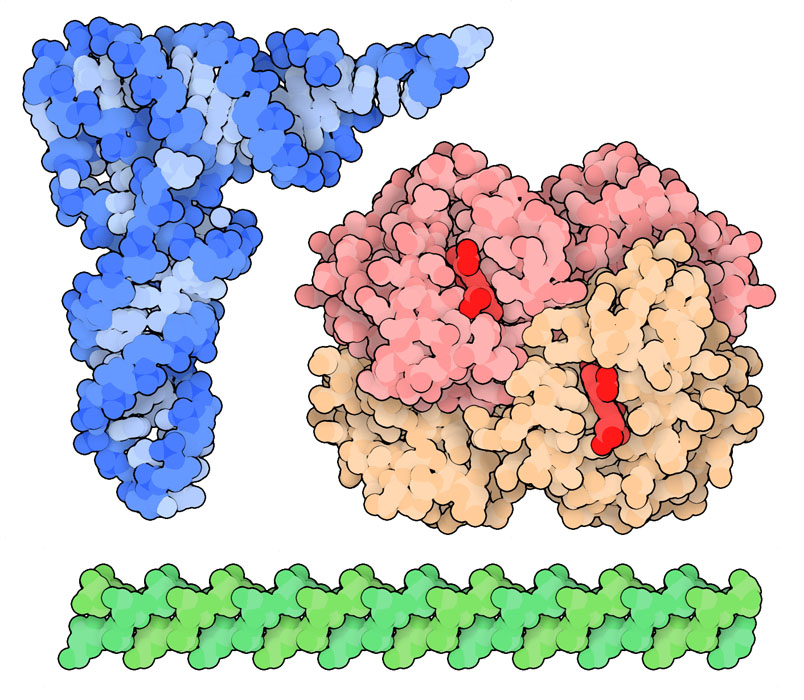
PDBの最初の10年は構造生物学(structural biology)の黄金時代だった。1960年代に最初のタンパク質構造が決定され、この記念すべき構造を解くために開発された実験技術に基づいてこの分野は急速に発展した。PDBは1971年に、先駆的なタンパク質構造から始まり、ポリペプチド鎖がどのようにしてある定まった階層的な立体的形状へと折りたたまれるのかを初めて垣間見せてくれた。最初の10年の終わりまでには、タンパク質、核酸、多糖という3種類の主な生体高分子の構造がPDBアーカイブで利用できるようになった。これらの構造を使って、科学者たちは生体高分子の構造、タンパク質の折りたたみ、酵素による触媒、そして遺伝情報の伝達に関して原子レベルで理解するための基礎を築いた。
恐ろしい美
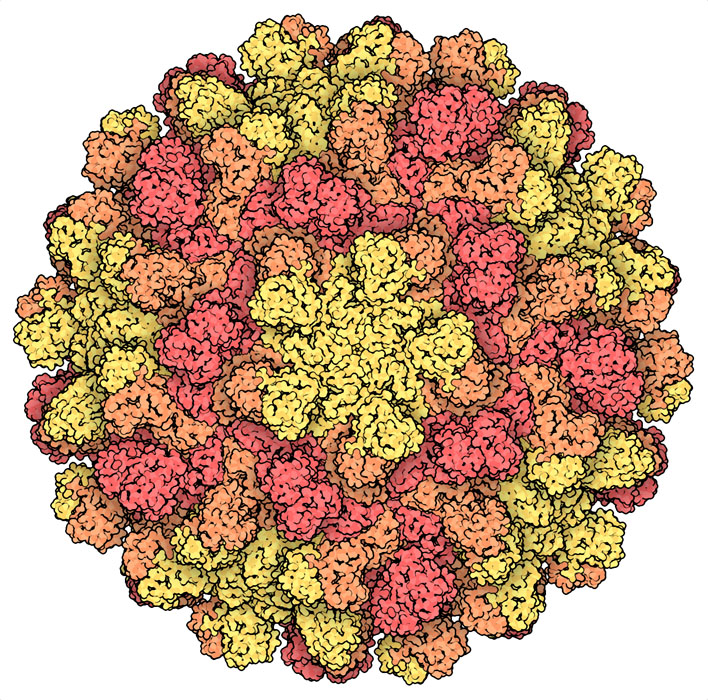
次の10年では、X線結晶学(X-ray crystallography)のツールが引き続き発展し、また溶液NMR(solution NMR)によっても構造が決定されるようになった。その結果新しい構造が次々と解明されるようになった。また、技術の進歩に加え、固有の対称性も活用することにより、初めてウイルスの原子構造が得られた。トマトブッシースタントウイルス(tomato bushy stunt virus)のような正二十面体型ウイルスのカプシド構造(ここに示す構造)と、ヒトに病気を引き起こすポリオウイルスとライノウイルス(poliovirus and rhinovirus)の構造により、準対称性(quasisymmetry)の理論に関する原子的基礎が明らかになり、ワクチン作用の分子的基礎に関する重要な知見が得られた。この10年の終わりになると、HIV-1タンパク質分解酵素(HIV-1 protease)の構造が解明され、HIV感染を致死的な危険から、扱いやすい慢性疾患へと変えるのに役立った。今日、構造生物学は既存のウイルス病原体および新たに出現するウイルス病原体と戦うための欠かせないツールとなっている。
とらえ難い膜チャネル
PDBにおける3期目の10年は、最初の膜貫通タンパク質である光駆動型プロトンポンプのバクテリオロドプシン(bacteriorhodopsin)の構造から始まった。膜タンパク質は非常に気まぐれであることで知られているが、何十年にもわたる研究の結果、膜に並んだタンパク質の詳細な構造が明らかになってきた。この成功を元にして、その後すぐに多くの膜貫通タンパク質の構造が解かれ、PDBアーカイブで利用できるようになった。今日、このような構造解析が難しいタンパク質の構造を解明するためのさまざまな手法がある。例えば、タンパク質をナノディスク(nanodisc)に単離しクライオ電子顕微鏡(cryo cryoelectron microscopy、クライオEM)を使って構造を決定することができる。
リボソームの構造が明らかになった
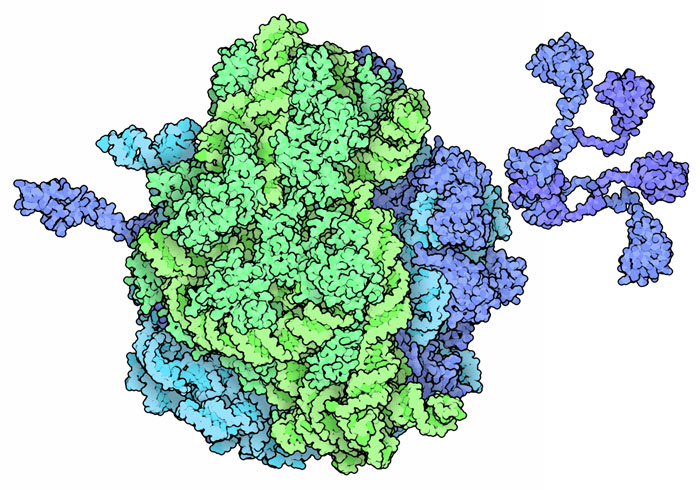
PDB 40年までの10年では、実験的な進歩が積み重なって、長い間探し求められていたリボソーム(ribosome)の構造が決定された。大サブユニットと小サブユニットの画期的な構造がきっかけとなり、その後すぐに翻訳因子(translation factor)、転移RNA(transfer RNA)、伝令RNA(messenger RNA)、およびその他の翻訳装置を伴ったリボソームの構造も解明され、タンパク質合成過程の原子レベルでの詳細が明らかになった。さらに、多くの構造によって細菌のリボソームを標的とする抗生物質(antibiotic drug)の作用が明らかになった。しかし、話はこれで終わったわけではなく、その後も次々と解明される構造によってリボソームの作用、集合、進化に関するさまざまな興味深いことが明らかになってきている。例えば、最近解かれたエクスプレソーム(expressome)の構造は、細菌が持つ一つの巨大機械が転写(transcription)と翻訳(translation)の全過程をどのようにして行っているのかを示してくれている。
分子を見る新たな手法
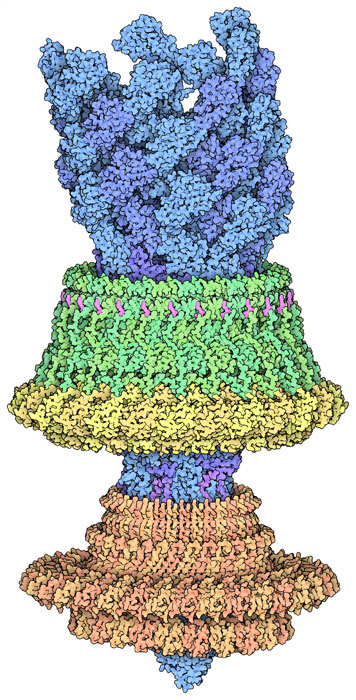
多くの新たな実験技術は、PDBの最近10年間における生体分子世界の新しい見方を提供してきた。XFEL(X-ray Free Electron Laser、X線自由電子レーザー)のような技術を使うと、分子機械を動画でとらえることができ、例えばリボスイッチ(riboswitch)のナノスケール(nm ナノメートルは1mmの100万分の1)での動きを明らかにしたり、発色団が光を吸収するところを観察したりすることができる。固体NMR(Solid state NMR)はアミロイド線維(amyloid fiber)のようなとらえにくい標的の構造を明らかにしてくれる。複数の技術を組み合わせた手法を使うと、核膜孔複合体(nuclear pore complex)のような前例のない大きさと複雑さを持つ集合体の構造を見ることができる。しかし、おそらく最も影響のあった進歩は、クライオEM(クライオ電子顕微鏡)の分解能が最近になって革命的に向上したことであり、これによりこれまで手に負えなかったあらゆる種類の分子集合体に手の届くようになった。この急成長している分野で例はたくさんあって一つの構造だけを選ぶことはできないが、私が現在気に入っているのは細菌の鞭毛モーター(flagellar motor)のローター(回転部)とブッシング(軸受筒)部分の構造である。この構造は、分子進化の展示物として興味をそそる一シーンを提供してくれる。そして、クライオEM研究の急速な発展ペースを考えると、集合体全体の原子レベルでの構造はそれほど時代遅れでもないようだ。
構造をみる
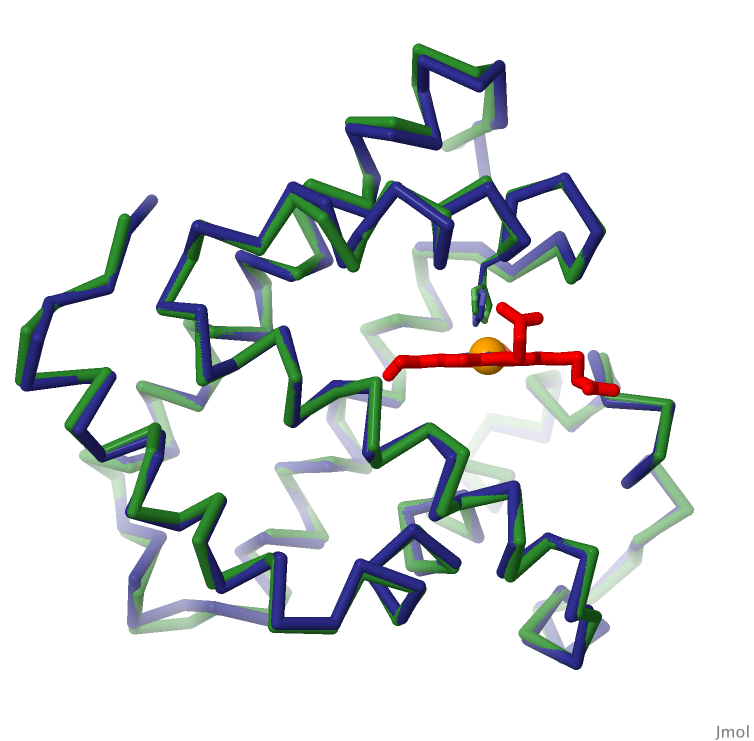
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
私がこの記事を書いているとき、構造生物学のコミュニティは、最近タンパク質構造予測の成功率を飛躍的に向上させたAlphaFold2とRoseTTAFoldによって変化しつつあるところだった。ここに示すのは簡単な一例で、AlphaFold2によって予測されたヒトのミオグロビン(myoglobin)の構造(青色)は、ケンドリュー(Kendrew)の研究室で解かれた歴史的構造でクジラ由来のミオグロビンの構造(緑色、PDBエントリー1mbn)や、その後に解明されたヒトのミオグロビンとほとんど変わらない(画像下のボタンをクリックすると対話的操作のできる画像に切り替えられる)。もちろん、この構造が予測しやすい理由はたった一つだけである。すべての予測方法は、PDBアーカイブで利用できる数十年分の構造に基づいて構築されているからである。これらの予測手法は巧妙なコンピュータ処理の成功例である。これはまた、PDBアーカイブに貢献してきた何万人もの研究者にとっても大いなる成功だと言えよう。これらはすべて、PDBにあるエントリーに含まれる広範な構造的知識と、アーカイブを自由に使えるようにしようとする彼らの意欲によって可能となったのである。
同じことは薬の発見と開発、ワクチン開発、酵素工学、バイオナノテクノロジーなど数多くの分野についても言える。これらはすべて、この構造データの宝の山に基づいて成り立っており、これによって生体分子の基本原理を理解し、それが新しい画期的な目標の達成に役立てられていく。今日、新たな構造決定技術の爆発的な増加に伴い、PDBアーカイブに登録された構造の数は急速に増加し続けている。何が可能になるか誰にもわからない。PDBの次の50年に向けて、わくわくする時代は確実にやってきているのだ!
理解を深めるためのトピックス
- PDBの歴史についてもっと知りたいなら、PDBの歴史(RCSB PDB、英語)にあるタイムラインを見てみてください。
- RCSB PDBのウェブサイトにはPDB50周年を祝うさまざまな出版物や教育的資料を掲載しています。
参考文献
- 2021 Synergies between the Protein Data Bank and the community. Nat Struct Mol Biol 28 400-401
- 7cgo 2021 Structural basis of assembly and torque transmission of the bacterial flagellar motor. Cell 184 2665-2679.e19
- 2019 Protein Data Bank: the single global archive for 3D macromolecular structure data. Nucl Acids Res 47 D520-D528
- 2013 Revealing structural views of biology. Biopolymers 99 817-824
- 4v4q 2005 Structures of the bacterial ribosome at 3.5 A resolution. Science 310 827-834
- 1rqv 2004 From structure and dynamics of protein L7/L12 to molecular switching in ribosome. J Biol Chem 279 17697-17706
- 2brd 1996 Electron-crystallographic refinement of the structure of bacteriorhodopsin. J Mol Biol 259 393-421
- 2tbv 1984 Structure of tomato bushy stunt virus. V. Coat protein sequence determination and its structural implications. J Mol Biol 177 701-713
- 6tna 1978 Crystal structure of yeast phenylalanine transfer RNA. I. Crystallographic refinement. J Mol Biol 123 607-630
- 1aga 1974 The agarose double helix and its function in agarose gel structure. J Mol Biol 90 269-284
- 1971 Nature New Biology 233 223
- 2dhb 1970 Three dimensional fourier synthesis of horse deoxyhaemoglobin at 2.8 Angstrom units resolution. Nature 228 551-552
- 1mbn 1969 The stereochemistry of the protein myoglobin. Prog Stereochem 4 299
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 










