262: PDB 구조에 대한 50년의 오픈 액세스 (Fifty Years of Open Access to PDB Structures)
단백질 정보 은행(Protein Data Bank)는 자신들의 연구 성과를 공유하고 싶다고 생각한 결정학자들에 의한 커뮤니티 주도의 활동으로서 시작되었다. 그리고 국제단백질구조 데이터뱅크(Worldwide PDB) 회원조직이 협력하여 운영하며, 전 세계의 연구실에서 제공되어 엄선된 생체분자구조에 누구나 자유롭게 접근할 수 있도록 보장하는 세계적 아카이브로 성장하였다. 지난 50년간 PDB 아카이브는 우리의 건강을 개선하는 데 효과적인 의약품을 찾거나 단백질이 기능하는 구조로 어떻게 접힐 수 있는지 예측하는 등 많은 분야에서 성장과 혁신을 촉구해왔다. 또한 PDB에 있는 정보는 생물학과 진화의 기본적인 구조에 관한 중요한 원자 수준에서의 이해로 이어져 연구나 교육에 사용되고 있다. 서비스가 시작된 지 50년이라는 기념할 만한 해를 축하하기 위해 PDB 아카이브에 등록된 수많은 매력적인 구조를 연대별로 선택했다. 이 정보를 바탕으로 PDB 아카이브를 탐색하여 각자의 마음에 드는 것을 찾아 주기 바란다.
구조생물학의 시작
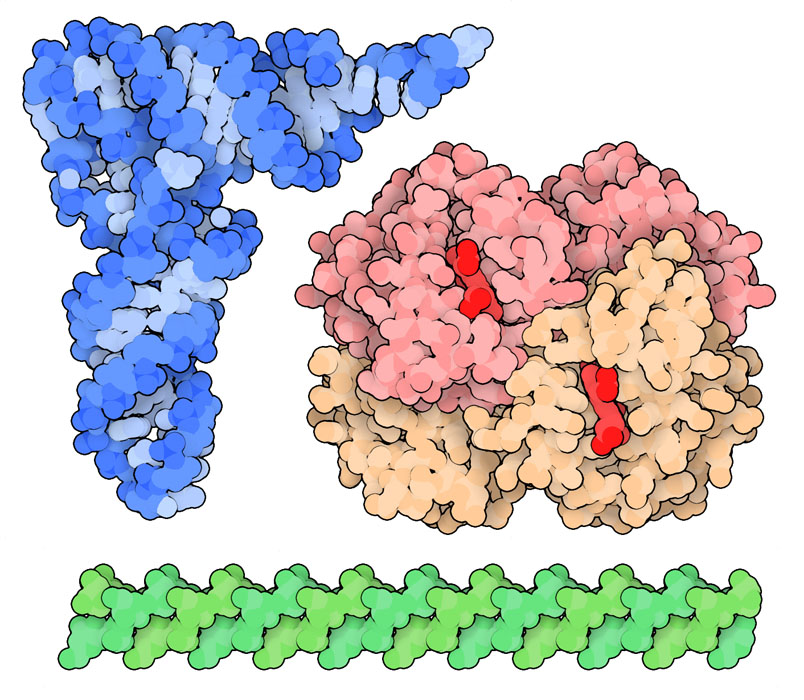
PDB의 첫 10년은 구조생물학(structural biology)의 황금기였다. 1960년대에 최초의 단백질 구조가 결정되었고, 이 기념할 만한 구조를 풀기 위해 개발된 실험 기술을 바탕으로 이 분야는 빠르게 발전했다. PDB는 1971년에 선구적인 단백질 구조에서 시작하여 폴리펩타이드 사슬이 어떻게 어떤 정해진 계층적인 입체적 형상으로 접히는지를 처음으로 엿볼 수 있었다. 처음 10년 말까지는 단백질, 핵산, 다당 등 3종류의 주요 생체 고분자 구조가 PDB 아카이브에서 이용할 수 있게 됐다. 이러한 구조를 사용하여 과학자들은 생체 고분자 구조, 단백질의 접기, 효소에 의한 촉매, 그리고 유전 정보 전달에 관해 원자 수준에서 이해하기 위한 기초를 구축했다.
무서운 미(美)
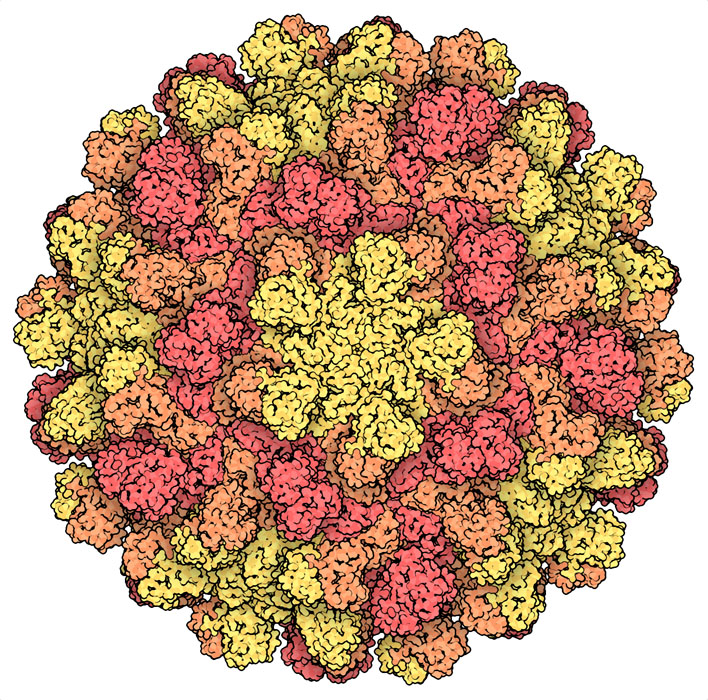
다음 10년에서는 X선 결정학(X-ray crystallography)의 툴이 계속 발전하고, 또한 용액 NMR(solution NMR)에 의해서도 구조가 결정되게 되었다. 그 결과, 새로운 구조가 속속 밝혀지게 되었다. 또한 기술의 진보에 더해 고유의 대칭성도 활용함으로써 처음으로 바이러스의 원자 구조를 얻을 수 있었다. 토마토덤불위축바이러스(tomato bushy stunt virus)와 같은 정이십면체형 바이러스의 캡시드 구조(여기에 나타낸 구조)와 사람에게 질병을 일으키는 폴리오바이러스과 리노바이러스 (poliovirus and rhinovirus)의 구조에 의해 준대칭성(quasisymmetry) 이론에 관한 원자적 기초가 밝혀져 백신 작용의 분자적 기초에 관한 중요한 식견을 얻을 수 있었다. 이 10년이 끝나자 HIV-1 단백질 분해 효소(HIV-1 protease)의 구조가 규명되어 HIV 감염을 치사적 위험에서 다루기 쉬운 만성질환으로 바꾸는 데 도움이 됐다. 오늘날 구조생물학은 기존의 바이러스 병원체 및 새롭게 출현하는 바이러스 병원체와 싸우기 위한 필수 도구가 되었다.
파악하기 어려운 멤브레인 채널

PDB의 3기째 10년은 최초의 막 관통 단백질인 광구동형 포톤 펌프인 박테리오로돕신(bacteriorhodopsin) 구조에서 시작됐다. 막 단백질은 매우 변덕스러운 것으로 알려져 있지만 수십 년에 걸친 연구 결과, 막에 나열된 단백질의 상세한 구조가 밝혀지고 있다. 이 성공을 바탕으로 그 후 곧 많은 막 관통 단백질의 구조가 풀려 PDB 아카이브에서 이용할 수 있게 되었다. 오늘날 이러한 구조 해석이 어려운 단백질의 구조를 규명하기 위한 다양한 수법이 있다. 예를 들어 단백질을 나노디스크(nanodisc)에 분리하고 저온전자현미경(cryo cryoelectron microscopy, 크라이오EM)을 사용하여 구조를 결정할 수 있다.
리보솜의 구조가 밝혀졌다
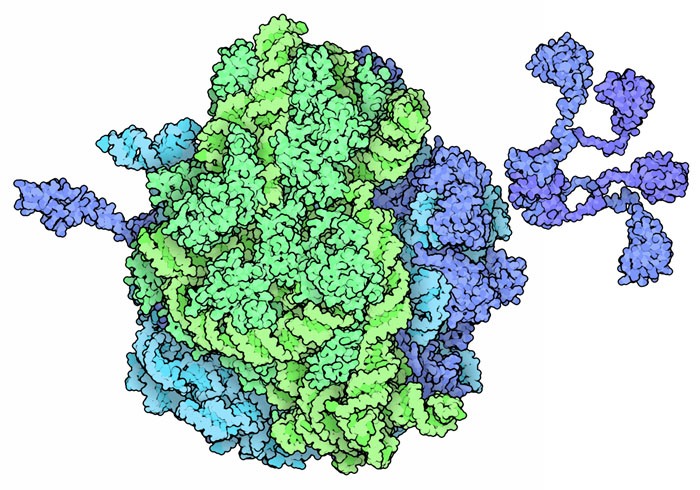
PDB 40년까지의 10년에서는 실험적 진보가 겹쳐 오랫동안 찾던 리보솜(ribosome)의 구조가 결정됐다. 대(大) 서브유닛과 소(小) 서브유닛의 획기적인 구조가 계기가 되었으며, 그 후 곧바로 번역인자(translation factor), 전이 RNA (transfer RNA), 전령 RNA(messenger RNA), 기타 번역 장치를 동반한 리보솜의 구조도 밝혀져 단백질 합성 과정의 원자 수준에서의 상세한 내용이 밝혀졌다. 게다가 많은 구조에 의해 세균의 리보솜을 표적으로 하는 항생물질(antibiotic drug)의 작용이 밝혀졌다. 그러나 이야기는 이것으로 끝난 것이 아니라 그 후에도 차례차례 규명되는 구조에 의해 리보솜의 작용, 집합, 진화에 관한 다양한 흥미로운 것들이 밝혀지고 있다. 예를 들어 최근 풀린 발현체(expressome)의 구조는 세균이 가진 하나의 거대 기계가 전사(transcription)와 번역(translation)의 전 과정을 어떻게 수행하는지 보여준다.
분자를 보는 새로운 수법
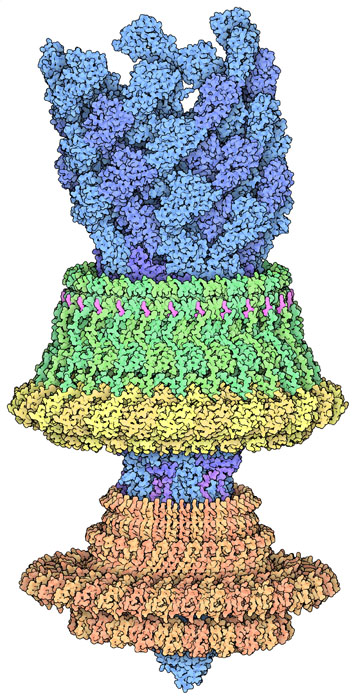
많은 새로운 실험 기술은 PDB의 최근 10년간 생체분자 세계에 대한 새로운 시각을 제공해 왔다. XFEL(X-ray Free Electron Laser, X선 자유전자레이저) 같은 기술을 사용하면 분자 기계를 동영상으로 포착할 수 있어서, 예를 들어 리보스위치(riboswitch)의 나노스케일(nm나노미터는 1mm의 100만분의 1)에서의 작용을 밝히거나 발색단이 빛을 흡수하는 것을 관찰할 수 있다. 고체 NMR(Solid state NMR)은 아밀로이드 섬유(amyloid fiber) 같은 포착하기 어려운 표적의 구조를 밝혀준다. 여러 기술을 조합한 수법을 사용하면 핵공복합체(nuclear pore complex) 같은 전례 없는 크기와 복잡성을 갖는 집합체의 구조를 볼 수 있다. 그러나 아마도 가장 영향이 있었던 진보는 크라이오 EM(저온전자현미경)의 분해능이 최근 들어 혁명적으로 향상된 것이며, 이를 통해 지금까지 감당할 수 없었던 모든 종류의 분자 집합체에 도달하게 되었다. 이 급성장하고 있는 분야에서 예가 많이 있어서 하나의 구조만 선택할 수는 없지만 내가 현재 마음에 드는 것은 세균의 편모 모터(flagellar motor)의 로터(회전부)와 부싱(베어링통) 부분의 구조이다. 이 구조는 분자 진화의 전시물로서 흥미를 유발하는 한 장면을 제공한다. 그리고 크라이오 EM 연구의 급속한 발전 속도를 생각하면 집합체 전체의 원자 수준에서의 구조는 그리 시대에 뒤떨어진 것도 아닌 것 같다.
구조 보기
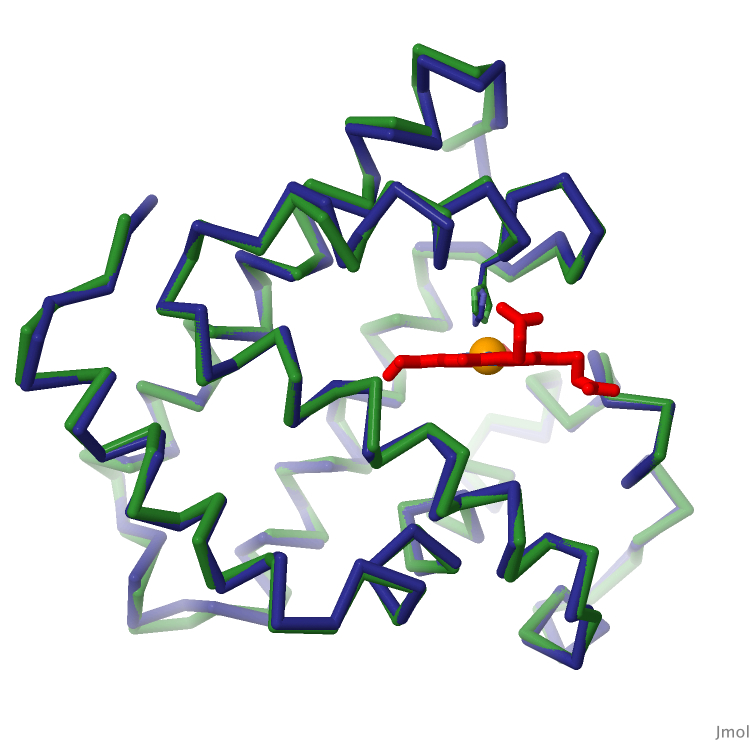
인터랙티브 조작이 가능한 페이지로 전환하려면 그림 아래의 버튼을 클릭하십시오. 읽기가 시작되지 않을 때는 그림을 클릭하십시오.
내가 이 기사를 쓰고 있을 때 구조생물학 커뮤니티는 최근 단백질 구조 예측 성공률을 비약적으로 향상시킨 AlphaFold2와 RoseTTAFold에 의해 변화하고 있는 중이었다. 여기에 나타낸 것은 간단한 예로, AlphaFold2에 의해 예측된 인간의 미오글로빈(myoglobin)의 구조(파란색)는 켄드류(Kendrew)의 연구실에서 규명된 역사적 구조로 고래에서 유래한 미오글로빈의 구조(녹색, PDB 엔트리 1mbn)나 그 후에 규명된 인간의 미오글로빈과 거의 다르지 않다(이미지 아래의 버튼을 클릭하면 인터랙티브 조작이 가능한 이미지로 전환됨). 물론 이 구조가 예측하기 쉬운 이유는 단 하나뿐이다. 모든 예측 방법은 PDB 아카이브에서 이용할 수 있는 수십 년분의 구조를 바탕으로 구축되어 있기 때문이다. 이러한 예측 수법은 정교한 컴퓨터 처리의 성공 사례이다. 이것은 또한 PDB 아카이브에 기여해 온 수만 명의 연구자에게 있어서도 큰 성공이라고 말할 수 있을 것이다. 이것들은 모두 PDB에 있는 엔트리에 포함되는 광범위한 구조적 지식과 아카이브를 자유롭게 사용할 수 있도록 하려는 그들의 의욕에 의해서 가능해진 것이다.
이와 같은 일은 약의 발견과 개발, 백신 개발, 효소 공학, 바이오나노기술 등 수많은 분야에 대해서도 말할 수 있다. 이들은 모두 이 구조 데이터의 보물섬을 바탕으로 이루어지고 있으며, 이를 통해 생체분자의 기본 원리를 이해하고 그것이 새로운 획기적인 목표 달성에 도움이 되어 간다. 오늘날 새로운 구조 결정 기술이 폭발적으로 증가함에 따라 PDB 아카이브에 등록된 구조의 수는 계속해서 빠르게 증가하고 있다. 무엇이 가능할지 아무도 모른다. PDB의 다음 50년을 향해 설레는 시대가 확실히 다가오고 있는 것이다!
이해를 높이기 위한 토픽
- PDB의 역사에 대해 더 알고 싶다면 PDB의 역사(RCSB PDB, 영어)에 있는 타임라인을 살펴보십시오.
- RCSB PDB의 웹사이트에는 PDB 50주년을 축하하는 다양한 출판물과 교육적 자료를 게재하고 있습니다.
참고문헌
- 2021 Synergies between the Protein Data Bank and the community. Nat Struct Mol Biol 28 400-401
- 7cgo 2021 Structural basis of assembly and torque transmission of the bacterial flagellar motor. Cell 184 2665-2679.e19
- 2019 Protein Data Bank: the single global archive for 3D macromolecular structure data. Nucl Acids Res 47 D520-D528
- 2013 Revealing structural views of biology. Biopolymers 99 817-824
- 4v4q 2005 Structures of the bacterial ribosome at 3.5 A resolution. Science 310 827-834
- 1rqv 2004 From structure and dynamics of protein L7/L12 to molecular switching in ribosome. J Biol Chem 279 17697-17706
- 2brd 1996 Electron-crystallographic refinement of the structure of bacteriorhodopsin. J Mol Biol 259 393-421
- 2tbv 1984 Structure of tomato bushy stunt virus. V. Coat protein sequence determination and its structural implications. J Mol Biol 177 701-713
- 6tna 1978 Crystal structure of yeast phenylalanine transfer RNA. I. Crystallographic refinement. J Mol Biol 123 607-630
- 1aga 1974 The agarose double helix and its function in agarose gel structure. J Mol Biol 90 269-284
- 1971 Nature New Biology 233 223
- 2dhb 1970 Three dimensional fourier synthesis of horse deoxyhaemoglobin at 2.8 Angstrom units resolution. Nature 228 551-552
- 1mbn 1969 The stereochemistry of the protein myoglobin. Prog Stereochem 4 299
 의 생체 고분자 학습 포털 사이트
의 생체 고분자 학습 포털 사이트 









