261: DNA配列ナノポア(DNA-Sequencing Nanopores)
この記事は、2021年1月に定量生物医学ラトガース研究所(Rutgers Institute for Quantitative Biomedicine)の主催で開催された1週間の集中合宿「生物医学における科学コミュニケーション」(Science Communication in Biology and Medicine)の一環で、ジェニファー・ジャン(Jennifer Jiang)、キャサリン・H・パーク(Katherine H. Park)、キランマイ・ヴェミュリ(Kiranmayi Vemuri)により執筆され、描かれたものです。
DNA塩基配列決定法(DNA sequencing)は生物学の研究に革命をもたらし、生命の過程をコードしている情報を明らかにした。ヒトゲノム計画(Human Genome Project)では従来のサンガー鎖停止法(Sanger chain-termination method)が使われ、全部で13年の歳月と約30億ドルの費用がかかった。ゲノミクスへの関心が高まるにつれ、より速く高性能で費用対効果の高い配列決定技術を求める声もますます高まっている。ナノポア配列決定法(nanopore sequencing)はこのような要求を満たすため急速に発展しているツールであり、デバイスの価格が1,000ドルを下回ることも珍しくない。今日、この技術は南極から国際宇宙ステーションまでどこででも使うことができる!
ポケットサイズの配列読み取り装置
ナノポア配列決定では、膜に埋め込まれた小さな穴をDNA鎖が通り抜けるときに配列を解読する。従来のDNA配列決定法とは異なり、DNAの合成や増幅に頼らずヌクレオチドを直接読み取っている。この技術は長いDNA断片の配列解読にも使えるので、生物の全ゲノムを素早く確実に断片化し読み取ることができる。一つのデバイス(配列読み取り装置、シーケンサー)には何千ものナノポア(nm ナノメートル=10億分の1メートル、程度のサイズを持つ穴)があるが、それでもシンプルかつ安価でポケットに入れることさえできる。現在、エラー率の低減や1回あたりの収量向上といった問題は最小限に抑えられている。
ナノポアが働く様子
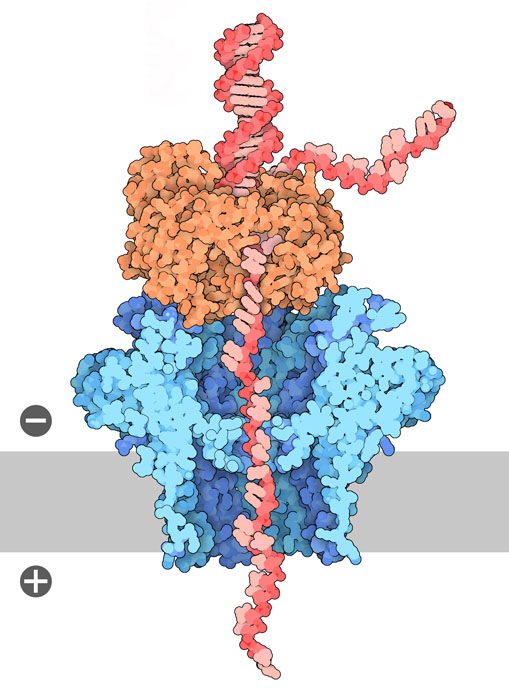
ナノポアシーケンサーには、塩溶液を2つの区画に区切る膜があり、その膜にナノポアが埋め込まれている。膜に電圧をかけるとイオンの流れが発生し、それを測定する。DNA鎖が穴を通るとき、イオンの流れが部分的に妨げられ、測定される電流値が下がる。4種類の核酸塩基に対応するイオン流の変化量はそれぞれ違うので、識別することができる。商業的に入手可能なシーケンサーは現在、細菌のタンパク質CsgGをナノポアに相当するものとして用いた派生品を使っている。ここに示すCsgG(PDBエントリー4uv3)は、直径約1nmで開閉しない非選択的な外膜タンパク質で、1本鎖DNAが容易に通り抜けられる。DNA読み取り性能を向上させるため、CsgGには改変が加えられていて、以下に示すようにナノポアの形が変わっている。その結果、収率と配列読み取りの正確性が向上している。
DNAの穴通しを制御する
DNA鎖がナノポアを通るときに配列を効率的に読み取れる速度を維持するには、DNA鎖を正確に制御する必要がある。遊離したDNA鎖がナノポアを通過する速度が速すぎると、各塩基をはっきりと読み取ることができなくなる。研究者は、ポリメラーゼ(polymerase)やヘリカーゼ(helicase)のようなDNA結合性酵素をナノポアのしくみに付加することにより速度を調節できることを発見した。ここに示すのはバクテリオファージ
天然の穴
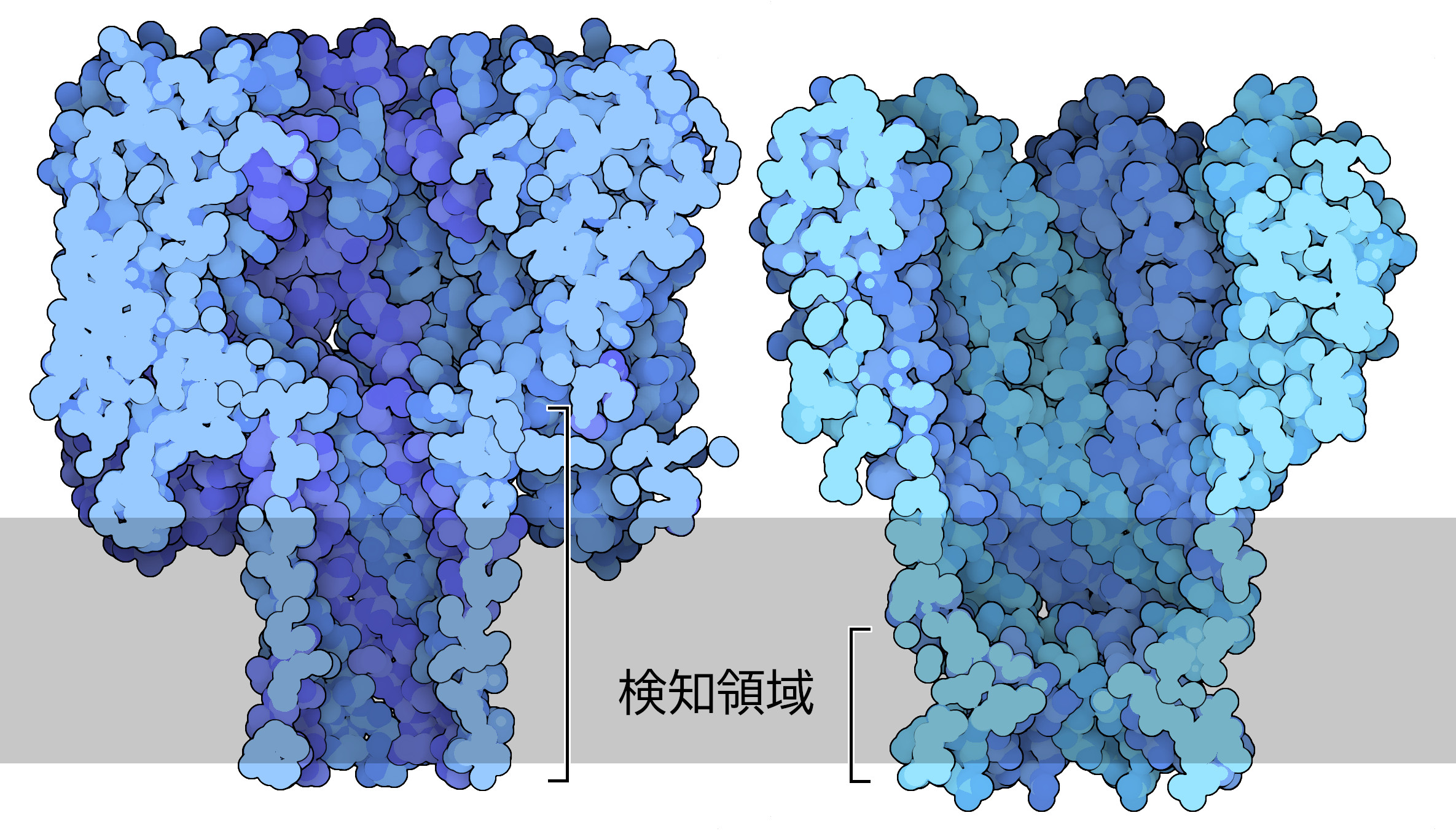
穴をつくる天然の膜タンパク質は他にもたくさん存在する。α溶血素(α-hemolysin、PDBエントリー7ahl)は細菌がつくる毒素で、DNA配列決定に用いられた最初の生物由来ナノポアでもある。穴は1.5nmから2.5nmの直径を持つ長いチャネルであり、個々の核酸塩基を識別する上で最適とは言えない。マイコバクテリア由来のタンパク質MspA(PDBエントリー1uun)は、これより短い検知領域を持つ天然のナノポアである。この特徴は利点となる。なぜなら、検知領域が短いナノポアでは記録される電流の特性に影響を与えるヌクレオチドが少なくなり、その結果より正確に配列を読み取ることができるからである。MspAは直径約1.2nmでより短い検知領域を持つので、α溶血素に比べ良いシーケンサーと言える。
構造をみる
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
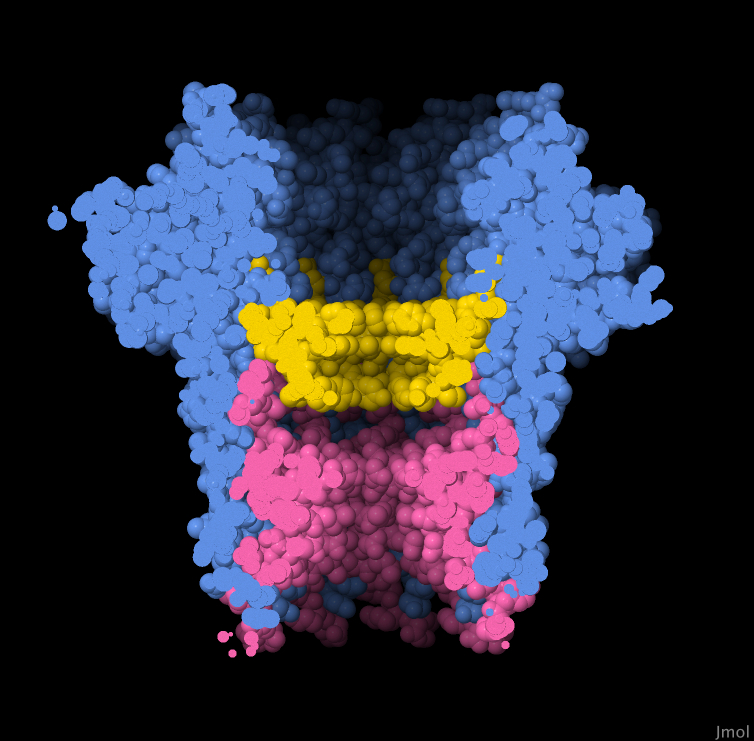
穴の安定性を向上させ、最適な機能を持つよう改変したナノポアがつくられている。例えば、二重狭窄ナノポア(dual constriction nanopor)ではCsgGに補助タンパク質を付加し、DNAを検知する2つ目の狭くなった部分を導入している。ここに示すCsgG-CsgF複合体(PDBエントリー6si7)では、CsgG分子を青色で、穴を構成している領域は黄色で、CsgF分子はピンク色で強調している。DNAの通る経路上において、CsgGが最初の狭くなった箇所を、CsgFが2番目の狭くなった箇所をつくっている。2つの狭くなった箇所からの信号を統合すると、塩基の読み取り精度が向上する。特に、同じ塩基が連続する箇所が複数存在するDNA領域ではそうである。図の下のボタンをクリックし対話的操作のできる画像に切り替えて、より詳しくこの構造を見てみて欲しい。
理解を深めるためのトピックス
参考文献
- 6si7 2020 A dual-constriction biological nanopore resolves homonucleotide sequences with high fidelity. Nat Biotechnol 38 1415-1420
- 2017 Robust long-read native DNA sequencing using the ONT CsgG Nanopore system. Wellcome Open Res 2 23
- 2017 Nanopore DNA sequencing and genome assembly on the International Space Station. Sci Rep 7 18022
- 2017 Real-time DNA sequencing in the Antarctic dry valleys using the Oxford Nanopore sequencer. J Biomol Tech 28 2-7
- 2016 Three decades of nanopore sequencing. Nat Biotechnol 34 518-524
- 4uv3 2014 Structural and mechanistic insights into the bacterial amyloid secretion channel CsgG. Nature 516 250-253
- 2010 Processive replication of single DNA molecules in a nanopore catalyzed by phi29 DNA polymerase. J Am Chem Soc 132 17961–17972
- 1uun 2004 The structure of mycobacterial outer-membrane channel. Science 303 1189-1192
- 1xhz 2004 Insights into strand displacement and processivity from the crystal structure of the protein-primed DNA polymerase of bacteriophage phi29. Mol Cell 16 609-618.
- 7ahl 1996 Structure of staphylococcal alpha-hemolysin, a heptameric transmembrane pore. Science 274 1859-1866
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









