241: 20年の分子を振り返って(Twenty Years of Molecules)
これまで毎月、今月の分子の記事を書くとき、私は科学者であること(そして科学アーティストでもあること)を幸運だと感じてきた。構造生物学は変革の途上にあり、生きた細胞の原子レベルでの働きを解き明かしつつある。すべてはミオグロビン(myoglobin)から始まり、今ではアクチン(actin)からジカウイルス(zika virus)に至る構造がすべてPDBに登録されている。20年に渡ってPDBアーカイブを調べ、見つけたものを今月の分子の記事で紹介してきて今月で20年となったご報告ができるのをうれしく思う。ではここで20年間の分子を振り返り考えたことを述べようと思う。
構造生物学の変革
構造生物学(structural biology)はケンドリュー(Kendrew)、ペルーツ(Perutz)、ワトソン(Watson)、クリック(Crick)の先駆的な研究以来、長い道のりを歩んできた。解析手法はより幅広い系においてより効率的でうまくいくようになっているため、細胞生活のあらゆる側面をさらによく調べることができる。構造ゲノミクスにおける努力の成果によって結晶構造の決定がより効率的となり、またX線自由電子レーザー(XFEL)のような手法により時間の次元での解析も新たにできるようになった。NMR分光学(NMR spectroscopy)は、より大きくより複雑なもの扱えるよう着実に進歩してきた。そして、他の手法では解析できない生物のしくみを見る他とは違った視点を提供している。クライオ電子顕微鏡(cryo electron microscopy, cryoEM)は、これまでであれば全く手に追えなかった巨大な集合体の構造を解明しつつある。今後数十年の構造生物学において間違いなく頼りになる手法となるだろう。
分子の物語
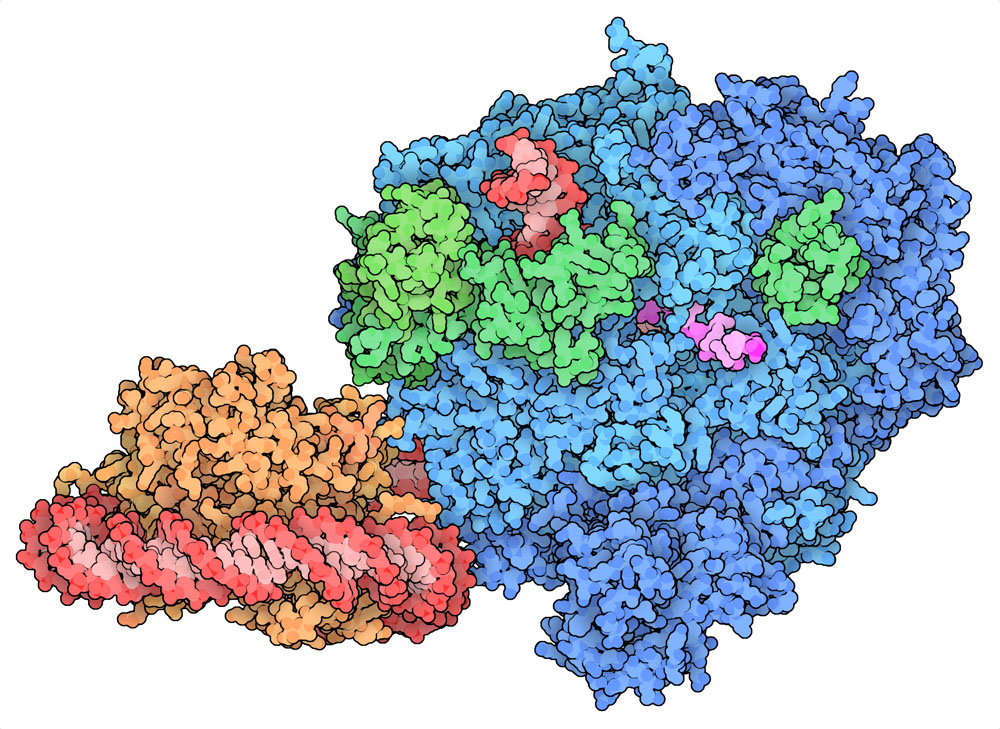
すべての構造にはなんらかの物語があって、細胞内における働きについて新たな見識を与えてくれる。ある構造はパズルにあらたなピースを埋め込むような意味合いを持つ。例えば、解糖系(glycolytic pathway)に関わるすべての酵素を解明するような場合に。またそれ自体が物語となっている構造もある。ここに示す構造(PDBエントリー6j4y)はRNAポリメラーゼ(RNA polymerase)がRNAを転写する瞬間をとらえ、ヌクレオソーム(nucleosome)に作用する場面をとどめている。伸長因子(elongation factor)は、RNAポリメラーゼがヌクレオソームからDNAをほどくのを助け、転写が続けられるようにする。この構造は、ヌクレオソームがほどかれる最中の複合体を示している。
最大のものと最良のもの
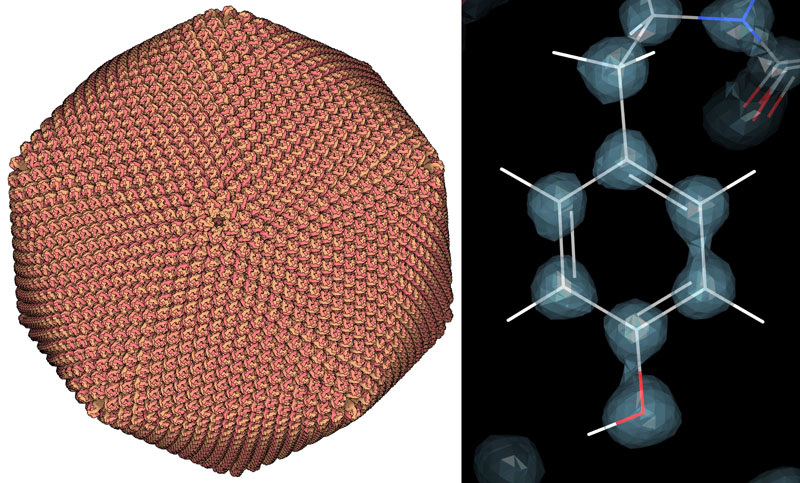
このコラムを書き始めたとき、PDBには既に13,500件の構造が登録されていた。そして20年経った今、その件数は10倍以上になっている。このPDBアーカイブについて知りたいことの一つは「最も大きい構造と最も良い構造を見つけること」かもしれないが、それは難しいことである。なぜなら「最も良い」が意味する内容を判断する尺度を見つける必要があるからである。例えば、ノーベル賞に注目すれば科学界に大きな影響を与えたテーマを見出すことはできる。ここに示すのは簡単に定量化できる2つの尺度に基づいて私が選んだものである。現在、最も結晶学的解像度が高い構造はクランビン(crambin、3nir)、最も鎖の数が多い集合体はファウストウイルス(faustovirus、5j7v)でこの構造は8280鎖で構成されている。この他の興味深い尺度でPDBアーカイブを見るには、PDB統計ページを見てみて欲しい。
分子の絵を描く
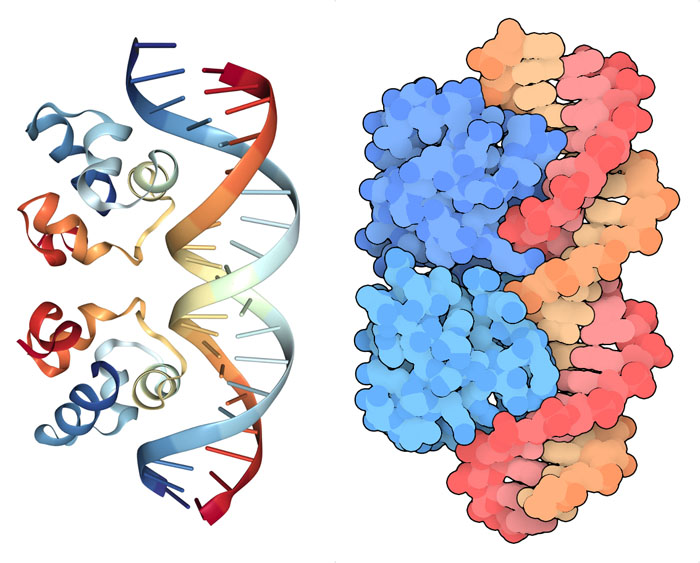
私にとって、楽しみは実際のところ分子の絵を描くところから始まった。その際、表現したい機能を強調する表現方法を選び、最も良い見栄えとなる見せ方を見つけ、魅力的で有益な配色を選ぶ、といった非常に多くの選択肢がある。幸いなことに、分子を視覚化する効果的なプログラムがたくさんあったので、私は素晴らしい画像を得ることに注力できた。この絵は、よくあるDNAとタンパク質の複合体を2種類の表現方法で示したもので、私の好きな取り組み方である。左は、NGLを使って素早く簡単に分子を見るためにつくった図で、どこにどんな構造があるのかが分かる。PDBエントリー2or1の概要ページにアクセスし、「構造」パネルをクリックすればこれと似たようなものを見ることができる。リボン表現(cartoon representation)で見ると、特徴的なαらせんがDNAの溝に入り込んでいるのがよく示されているところに注目して欲しい。次に、全原子を表示した図をつくる私の開発した描画プログラムで、タンパク質がDNAの表面をどのように包み込んでいるのかをわかりやすくした図を描いた。
世界の健康
恐らく、構造生物学にとっての主たる励みになっているのは、私たちの健康を改善する方法を見つけることである。生体分子の構造は、私たちの体について原子レベルでの基礎を理解し、何か不具合が生じたとき標的をしぼった介入を行う上での他にない情報源となる。構造の情報を活用した創薬が最も直接的な取り組みとなる。HIV感染に対抗する新たな薬を見つけるのにHIVタンパク分解酵素(HIV protease)の構造を使う、糖尿病(diabetes)を治療するインスリン(insulin)の新たな型をつくる知見を得るのに構造情報を使うなどの事例がこれにあたる。また生体分子による過程を使って命令を下すことも行われている。病気になった細胞で特定の遺伝子を編集する改変型CRISPR分子がこの例として挙げられる。ワクチンも現代医学における奇跡の一つと言えよう。これは、世界からポリオ(polio)と天然痘(smallpox)による危機を取り除くのに欠かせないものとなっている。現在、対象となるウイルスと結合した抗体(antibody)の構造を使って成功したこれまでの事例を、HIVやエボラ(ebola、ここに示すのはPDBエントリー6mamの構造)などより困難な対象にも広げようという試みが行われている。
生物学の利用
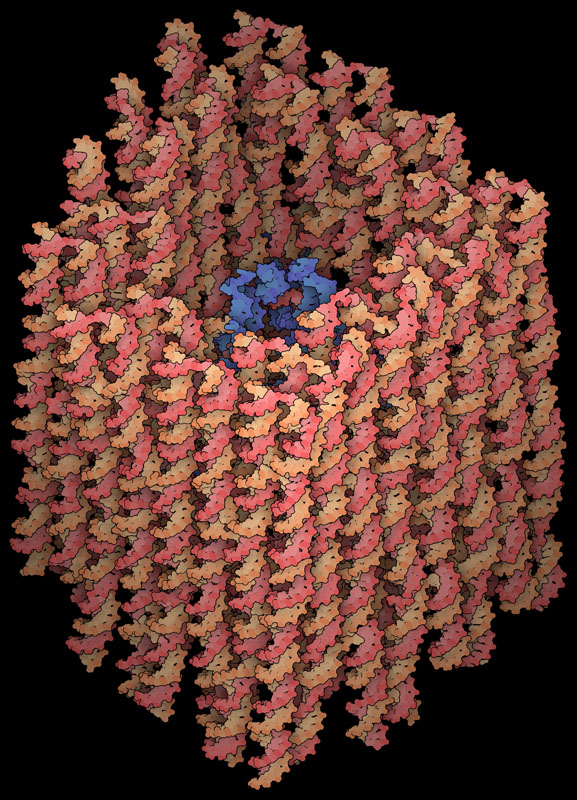
生体分子は実際に動作するナノサイズ機械の実例であるため、当然ながら研究者たちは自分たちでナノマシンをつくることに関心を持っている。そして多くの巧妙な取り組みが成功している。指向性進化(directed evolution)を利用し、既存の酵素が持つ機能を調整して希望する機能を生み出した事例がある。また切り取り&貼り付けのやり方も取り入れた事例もある。例えば、既存のタンパク質をつなげて自律集合型のナノサイズ容器がつくられている。DNA折り紙では、DNAの塩基対の精巧な特異性を利用し、任意の形状を持つナノサイズの構造、例えばここに示す小さな箱型(PDBエントリー6by7)がつくられている。これには一本鎖領域があって、機能部位をつなぎとめるのに使われる。
機能的な対称性
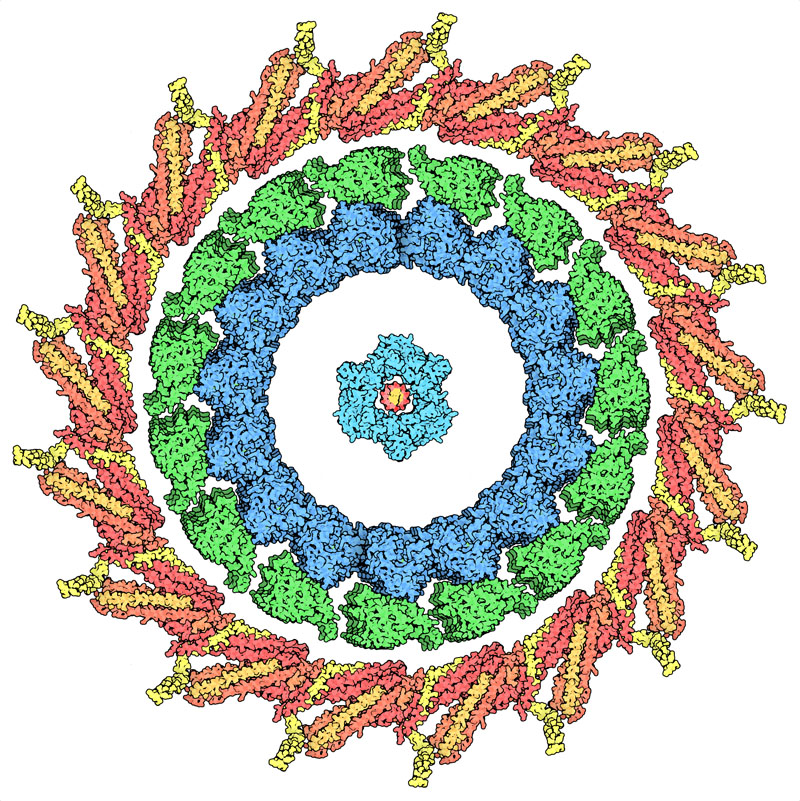
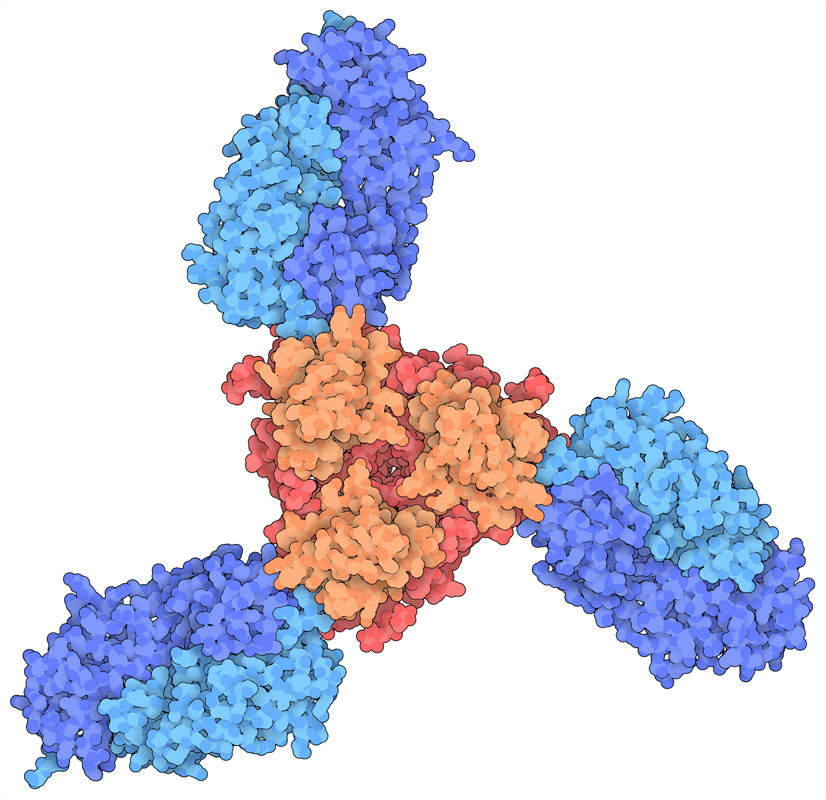
生体分子には独特な美しさがあって、対称性のある生体分子の場合、その美しさは目的を伴っている。私は大学院にいた頃から、生物学的対称性に魅せられてきた(そうなるのは当然だろうと確信している、私は結晶学者としてやってきたのだから)。部品となる分子サブユニットが対称性をもって多数集まり機能を持つ集合体となるが、機能がこの対称性に依存してる興味深い事例が生体分子にはたくさんある。例えば、ウイルスカプシドは完全な対称性をもってつくられるが、より大きな入れ物が必要なときはそれに近い準対称性(quasisymmetry)が使われる。ここでは環状の対称性を用いてつくられたリング状分子が構築された事例を2つ紹介する。これらはらせん対称を持つ線維状生体分子を取り囲む機能を持っている。ここには動原体(kinetochore、PDBエントリー6cfz)があってこれが微小管(microtubule)とキネシン(kinesin、PDBエントリー3j2u)を取り囲んでいる。またプロセシビリティー因子PCNA(processivity factor PCNA、PDBエントリー1plq)がDNAを取り囲んでいる。
構造をみる
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
テオドシウス・ドブジャンスキー(Theodosius Dobzhansky)は「進化の観点から見れば生物学に意味のあるものなどない」(Nothing in biology makes sense except in the light of evolution)と言ったことで有名だが、PDBアーカイブでは直接これにつながるものを見ることができる。千年以上かかって起こる進化もあり、例えばグロビン(globin)などのタンパク質の配列を比較することにより、関係する生物の詳細な系統を見つけることができる。細菌の薬剤耐性(bacterial drug resistance)では、既存の生物機械を使い変異と選択を通じて薬剤を回避する新たな手法を進化させるので、数年のうちに起こる進化が見られる。
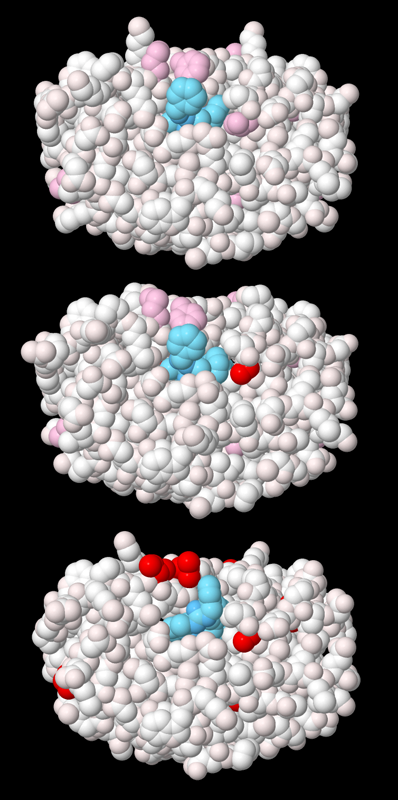
ここに示す構造は、進化が数日のうちに起こるのを明らかにしている。培養したHIVをHIVプロテアーゼにさらすと、変異と選択によって抵抗性がさらに増すのが観察された。上に示す野生型酵素(PDBエントリー2az8)は阻害剤によって強く阻害される。ロイシンをより小さなアラニンに取り替えると、中央に示す酵素(PDBエントリー2az9)は望ましい相互作用ができなくなり、阻害剤に対する抵抗性が4倍になる。下に示す構造(PDBエントリー2azc)はさらに酵素活性を調整したもので、さらに5ヶ所に変異を加え、抵抗性を30倍に高めている。図の下のボタンをクリックして対話的操作のできる画像に切り替え、より詳しく見てみて欲しい。
理解を深めるためのトピックス
- あなたの好きな分子はどれだろうか?
- 今月の分子の記事の目標と手法について興味があるなら、このRCSBニュースレターの記事を読んでみて欲しい。
参考文献
- 6j4y 2019 Structural insight into nucleosome transcription by RNA polymerase II with elongation factors. Science 363 744-747
- 6mam 2019 Structural basis of broad ebolavirus neutralization by a human survivor antibody. Nat. Struct. Mol. Biol. 26 204-212
- 6by7 2018 Folding DNA into a Lipid-Conjugated Nanobarrel for Controlled Reconstitution of Membrane Proteins. Angew. Chem. Int. Ed. Engl. 57 2072-2076
- 6cfz 2018 Structure of the DASH/Dam1 complex shows its role at the yeast kinetochore-microtubule interface. Science 360 552-558
- 5j7v 2016 Structure of faustovirus, a large dsDNA virus. Proc.Natl.Acad.Sci.USA 113 6206-6211
- 3j2u 2013 Structural model for tubulin recognition and deformation by Kinesin-13 microtubule depolymerases. Cell Rep 3 759-768
- 3nir 2011 Crystal structure of small protein crambin at 0.48 A resolution. Acta Crystallogr.,Sect.F 67 424-429
- 2az8、2az9、2acz 2006 Structural Insights into the Mechanisms of Drug Resistance in HIV-1 Protease NL4-3. J.Mol.Biol. 356 967-981
- 1plq 1994 Crystal structure of the eukaryotic DNA polymerase processivity factor PCNA. Cell 79 1233-1243
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









