280: 主要組織適合性複合体クラスIローディング複合体(MHC I Peptide Loading Complex)
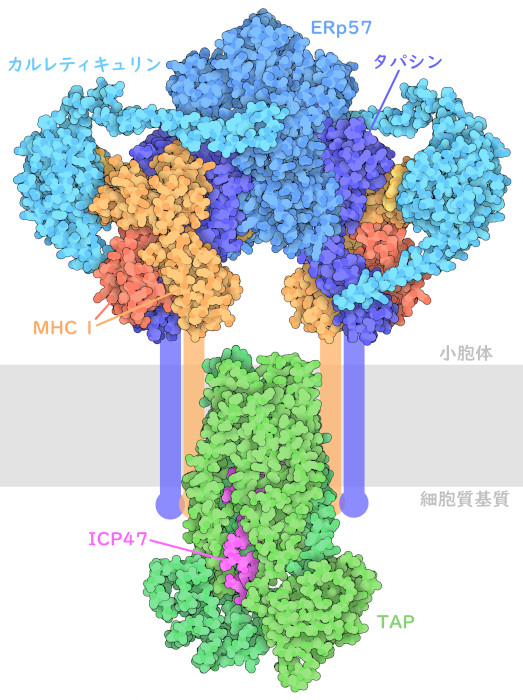
私たちの免疫システムは常に意思決定を行っている。感染症やがんを探し出して破壊すると同時に、健康な生活に必要となる多くの正常細胞が行う過程は無視するという難しい仕事を課せられている。そのため、免疫系が感染した細胞や腫瘍を発見したときは、本当に問題があるのかを確実に確認しておく必要がある。もし誤った決定を下して健康な細胞を攻撃すると、生命を脅かす炎症や自己免疫疾患につながる可能性がある。主要組織適合性複合体 I(Major Histocompatibility Complex I、MHC I)は、免疫系がこれらの決定を下すのに役立つ。私たちの細胞のほとんどは、細胞内にあるタンパク質をいくつか切り刻み、MHC Iを使ってその小さなペプチド片を細胞表面に提示する。そうすることで、免疫系は細胞内で何が起こっているかを監視できる。ここに示すペプチドローディング複合体(peptide loading complex、PLC)は、細胞が最も関心を持っているペプチドだけをMHC Iに認識させるのに役立つ。
複合体へのペプチド取り込み
ペプチドローディング複合体にはさまざまな機能部位があり、空のMHC Iを見つける部分、ペプチドを取り込ませる部分、ペプチド-MHC I複合体の安定性を確認する部分、そして複合体を細胞表面に送り出す部分で構成されている。その作用のほとんどは小胞体(endoplasmic reticulum)で起こる。タンパク質シャペロンのカルレティキュリン(calreticulin)は、MHC Iの表面にある特徴的な糖鎖(glycan)を認識して複合体の構築を始める。次に、シャペロンのタパシン(tapasin)、ジスルフィド異性化酵素(disulfide isomerase)のERp57、そして輸送体のTAP(「抗原処理に関する輸送体」、transporter associated with antigen processing)を呼び集める。全体が組み上がると、ペプチドが取り込まれる。最後に、複合体が分解され、MHC Iが持つ糖鎖の末端からブドウ糖(glucose)が切り取られる。これがペプチド-MHC I複合体の輸送準備ができたという合図になる。
PLCが活動する様子
この図には、実験的に決定された、ペプチドローディング複合体の異なる2つの部分に関する原子構造を示している。PDBエントリー7qpdの構造は、小胞体の中で何が起こっているかを示している。カルレティキュリン、タパシン、そしてERp57がMHC Iを取り囲んでいる。タパシンはペプチドが結合する場所となる溝の形状を変化させて一端を少し広くし、また溝の一部に小さな蓋をつくる。こうすることにより、結合力の弱いペプチドが入り込みにくくなる。その結果、複合体はペプチドの結合を促進し、結合力が強い最適なペプチドを優先するようになる。PDBエントリー5u1dの構造には、ペプチドを小胞体に輸送する役割を担うTAPが含まれている。これは、P-糖タンパク質(P-glycoprotein)やその他の多剤輸送体(multidrug transporter)と同様に、2つの似たタンパク質サブユニットで構成されたATP駆動型輸送体で、これらのサブユニットは小胞体膜を通る通路を作っている。この構造において、輸送体は、単純ヘルペスウイルスの小さなタンパク質であるICP47を使って動かないようにしてある。ICP47は、ウイルスタンパク質の輸送を妨げ、ウイルスが免疫系から隠れられるようにする。
二重チェック
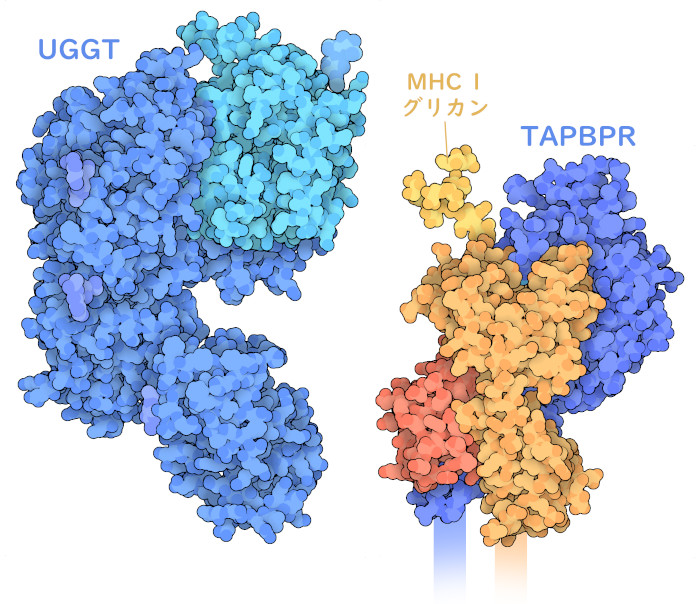
驚くべきことに、最も関連性の高いペプチドだけが細胞表面に提示されるようにするため、さらにもう1段階のしくみを使って品質管理が行われている。ペプチドとMHC Iとの複合体が輸送過程を経て移動すると、別のタンパク質群によって検査される。TAPBPR(「TAP関連結合タンパク質」、TAP-binding protein-related、ここに示すのはPDBエントリー5opiの構造)は、タパシンと同様の方法でMHC Iに結合し、ペプチドがしっかり結合しているのかをチェックする。もしペプチドがテストに合格しなければ、UGGTタンパク質(「UDP-グルコース:糖タンパク質グリコシル基転移酵素」、UDP-glucose:glycoprotein glucosyltransferase、ここに示すのはPDBエントリー5mzoの構造)がMHC Iの糖鎖にブドウ糖を戻す。これは空になったMHC Iを小胞体に戻して別の仕事ができるようリサイクルするための細胞に対する信号となる。
ペプチドの端を切り落とす
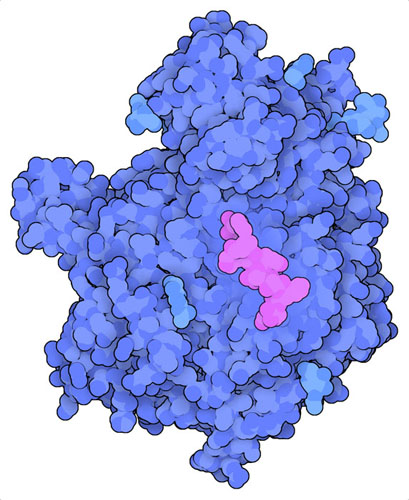
TAP輸送体は、長さが8から40アミノ酸程度の広範囲に渡るペプチドを小胞体に送り届ける。しかし、MHC Iは、より短い8から10アミノ酸程度のペプチドを好む。そのため、似た2つの酵素、ERAP1とERAP2(「小胞体アミノペプチダーゼ」、endoplasmic reticulum aminopeptidase、ここに示すのはPDBエントリー5ab0の構造)がペプチドの端を切り落として適切なサイズにする。ヒトDNAに関する最近の歴史的研究により、この過程がいかに重要であるかが明らかになった。中世に発生した黒死病(Black Death)は、記録に残る歴史上で最もたくさんの人が亡くなった出来事であり、人類の3分の1から2分の1が亡くなったと言われている。当時の人々から抽出された数百のDNA抽出物に関する最近の研究により、ERAP2活性が強くなった人は生存する可能性が40%高くなることが明らかになった。これは、記録に残る歴史の時代に発生した人類の進化的自然淘汰の一例であり、後に腺ペスト(bubonic plague)のパンデミックが再発した際、ある集団は何世紀にもわたって低い死亡率を示した。
構造をみる
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
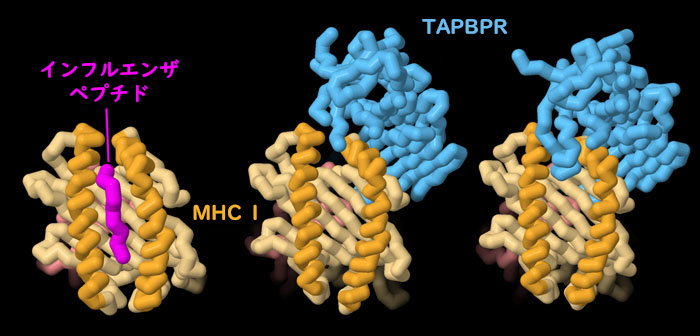
ペプチドに結合したMHC IとTAPBPRに結合したMHC Iの構造を比較することで、TAPBPRはどのようにして複合体を校正しているかを確認できる。ペプチド複合体(PDBエントリー1hoc)では、MHC Iの中にある2つのαらせんがペプチドの両側にしっかりと隣接し、全長にわたってペプチドを抱え込んでいる。一方TAPBPR複合体(PDBエントリー5wer)では、αらせんがわずかに離れている。最も適したペプチドには、MHC Iをより窮屈な狭い溝へと変形させられる十分強力な結合力が必要である。画像の下のボタンをクリックして対話的操作のできる画像に切り替え、これらの構造をより詳しく見てみて欲しい。
理解を深めるためのトピックス
- ここに示す構造の中には一部タンパク質の膜貫通部分が含まれていません。欠けている部分を理解するには、RCSB PDBのサイトでタパシンのページなど各タンパク質のGroup Sequenceページを参照してください。
- 細菌の輸送体でTAPに似ているTmrABが持つ一連の構造を見ると、輸送に必要となるさまざまな配置を確認することができます。例えば、内向きに開いた配置を取るPDBエントリー6ranの構造と、外向きに開いた配置を取るPDBエントリー6rahの構造を見比べてみてください。
- 今月の分子の記事T細胞受容体にアクセスし、これらのペプチドがどのようにして免疫系に認識されるのかを確認してみてください。
参考文献
- 7qpd 2022 Molecular basis of MHC I quality control in the peptide loading complex. Nat Commun 13 4701-4701
- 2022 Evolution of immune genes is associated with the Black Death. Nature 611 312-319
- 2020 Multifunctional chaperone and quality control complexes in adaptive immunity. Annu Rev Biophys 49 135-161
- 2019 MHC I chaperone complexes shaping immunity. Curr Op Immunol 58 9-15
- 2017 Structure of the human MHC-I peptide-loading complex. Nature 551 525-528
- 5mzo 2017 Interdomain conformational flexibility underpins the activity of UGGT, the eukaryotic glycoprotein secretion checkpoint. Proc Natl Acad Sci U S A 114 8544-8549
- 5opi 2017 Structure of the TAPBPR-MHC I complex defines the mechanism of peptide loading and editing. Science 358 1060-1064 DOI:10.1126/science.aao6001 PMID:29025996
- 5u1d 2016 Structure of the transporter associated with antigen processing trapped by herpes simplex virus. Elife 5 e21829
- 5ab0 2015 Structural basis for antigenic peptide recognition and processing by Endoplasmic Reticulum (ER) Aminopeptidase 2. J Biol Chem 290 26021-26032
- 1hoc 1994 The three-dimensional structure of H-2Db at 2.4 A resolution: implications for antigen-determinant selection Cell 76 39-50
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









