270: ピルビン酸キナーゼM2(Pyruvate Kinase M2)
この記事は、定量生物医学ラトガース研究所(Rutgers Institute for Quantitative Biomedicine)の主催で開催された1週間の大学生・大学院生向け集中合宿の一環として、ファイザ・アハメッド(Faiza Ahmed)、ジョナサン・アッシュ(Jonathan Ash)、サース・パテル(Thirth Patel)、オーリル・サンダース(Auriel Sanders)により執筆され、描かれたもので、2022-2023年 PDB-101の健康フォーカス「がん生物学と治療法」(Cancer Biology and Therapeutics)の一部としても掲載されています。
がん細胞は、自由に増殖できるようにするため、根本的な部分で多くの変化を起こしている。驚くべきことに、エネルギーをつくり出す経路の再プログラミングもそのような変化の一つである。正常な細胞の場合、ブドウ糖(glucose)をエネルギーに変換する過程は、細胞質における解糖系(glycolysis)から始まる。解糖系の最後に生成されるピルビン酸(pyruvate)は、通常ミトコンドリアに入ってクエン酸回路(citric acid cycle)に進む。酸素があると、この回路は電子伝達系(electron transfer chain、複合体I、III、IV)とATP合成酵素(ATP synthase)の働きによる好気性なエネルギー産生を促進する。がん細胞は大量のブドウ糖を取り込むが、解糖の最終段階はブロックしている。これらの細胞で作られたピルビン酸は、ミトコンドリアに入いっていくのではなく、乳酸へ変換される。
ピルビン酸キナーゼM2: 門番
解糖の最終段階は、ピルビン酸キナーゼ(pyruvate kinase、PDB ID 4fxf)という酵素によって触媒される。この酵素は、ホスホエノールピルビン酸(phosphoenolpyruvate)からリン酸基を転移させ、ATP(細胞のエネルギー通貨)とピルビン酸をつくる反応を触媒している。細胞の生存に不可欠であるため、ピルビン酸キナーゼの活性は厳重に制御されている。この酵素は、高活性型4量体と低活性型2量体の2種類の集合体をつくる。より多くのエネルギーが必要なときは、フルクトース2リン酸(fructose bisphosphate、解糖系のより前の段階で生成される分子)が4量体形成を促進する。一方、他の様々な制御分子が、特定のアロステリック部位(活性部位とは別の場所だが活性に影響を与える部位)に結合して2量体形成を促し酵素活性を抑えることができる。体内では4種類の組織特異的なピルビン酸キナーゼが作られる。PKM1は多くのエネルギーを必要とする細胞で、PKRは赤血球で、PKLは肝細胞で、PKM2は通常胚細胞やその他増殖中の細胞で作られる。PKM1酵素は一般に4量体の形で存在し、常に活性のある状態になっている。しかし、多くのアロステリック制御分子がPKM2酵素と結合し、酵素が4量体から2量体への移行に影響を与えることでその作用を制御している。がん細胞はこの制御を利用し、自らの代謝のしくみを作り直している。
エネルギー産生のパラドックス
多くのがん細胞は、酵素のピルビン酸キナーゼM2(PKM2)を大量につくり、エネルギー産生経路を解糖系に移行させている。この代謝の作り直しは、2つの理由から逆説的である。第一に、これらの細胞は酸素が豊富にあっても好気性エネルギー産生を行わない。第二に、これらの急速に分裂する細胞は正常細胞より多くのエネルギーを必要とするのに、エネルギー効率の著しく悪い解糖を主なエネルギー源として使用している。この相反する挙動に対する答えは、PKM2の速度を低下させたることによって生じる別の代謝産物にある。すなわち、解糖系における前段階でできる代謝中間体が蓄積されることにある。これらの中間体は多くの生合成経路に転用され、がん細胞は急速な成長に必要なタンパク質や核酸の構築部材をつくることができる。このように、がん細胞はエネルギー産生よりも成長を優先させる。このようなブドウ糖利用の再プログラミングは、ワールブルグ効果(Warburg Effect)と呼ばれている。この名称は、これを発見した研究者オットー・ハインリッヒ・ワールブルグ(Otto Heinrich Warburg)の名にちなんで名づけられたものである。
ワールブルグ効果とがん治療
ワールブルグ効果は、腫瘍形成における共通の特徴である。その発見以来、がん細胞ではPKM2がリン酸化およびアセチル化されていることが見いだされてきた(PDBエントリー6wp3)。これらの修飾は、フルクトース2リン酸などの活性化因子が結合する部位に影響を与える。これらの活性化因子がなければ、PKM2は効率よく高活性な4量体を形成することができない。この酵素の2量体は、解糖において活性が低いだけでなく、核内に移動して他のがんを引き起こす遺伝子の転写を促進することがある。このような変化は、乳酸の蓄積を引き起こし、腫瘍の進行やがん治療への耐性を助長することになる。PKM2はがん細胞の再プログラミングに重要な役割を果たすため、現在、PKM2を阻害する薬剤や治療法、あるいは好気性代謝を活性化する別の方法を模索する研究が行われている。
構造をみる
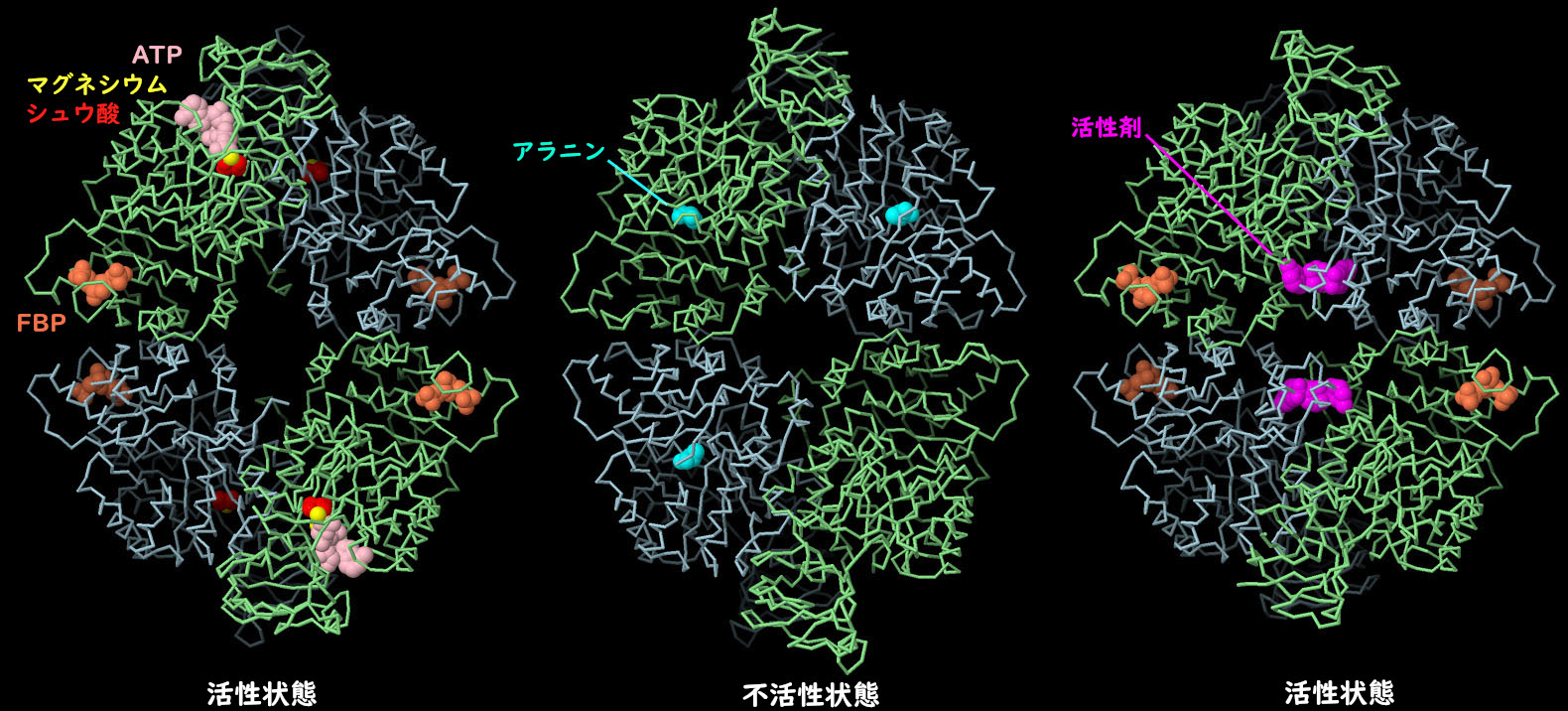
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
PKM2 4量体の構造から、さまざまな低分子がアロステリックな形状変化を引き起こし、タンパク質の作用を制御していることが明らかになった。左に示すのが活性型(PDB ID 4fxf)で、ATP、シュウ酸(oxalate)、マグネシウム(magnesium)が活性部位に、フルクトース2リン酸(fructose bisphosphate、FBP)が制御部位にある。中央に示すのが不活性型(PDB ID 6gg3)で、制御部位にはアラニンがある。形状が変化し、中央部が細くなっていることに注目してほしい。がん治療に役立てるため、PKM2に結合する薬物分子を探す研究が積極的に行われている。例えば、右に示す構造(PDB ID 3me3)では、人工の活性化分子(赤紫色)がFBPとともに結合しているが、これにより分子を活性型に変化させ、この分子で処理した細胞でウォーバーグ効果を逆転させることができると期待されている。
理解を深めるためのトピックス
- 乳酸脱水素酵素(lactate dehydrogenase)を阻害することでも細胞増殖を遅らせることができ、がんの進行度や治療効果のモニタリングに利用されています。PDBエントリー4ajpで、抗がん剤の試作品と結合した乳酸脱水素酵素の構造を見てみてください。
- PDBアーカイブには、ヒト由来PKM2に様々な低分子が結合した構造が多数登録されています。PDBエントリー4fxfを含むそのような構造を見つけるには、PDBjのPDB検索(PDBj Mine)にアクセスし、「外部データベース」で「UniProt」を選択し、入力ボックスに「P14618」と入力して検索した結果を見てみてください。
参考文献
- 2021 Revisiting the Warburg effect: Historical dogma versus current understanding. The Journal of Physiology 599 1745–1757
- 6wp3 2020 Structural basis for allosteric regulation of pyruvate kinase M2 by phosphorylation and acetylation. Journal of Biological Chemistry 295 17425–17440
- 2019 Pyruvate kinase M2: A simple molecule with complex functions. Free Radical Biology and Medicine 143 176–192
- 2019 Functional cross-talk between allosteric effects of activating and inhibiting ligands underlies PKM2 regulation. ELife 8 e45068 DOI:10.7554/eLife.45068
- 6gg3 2018 An allostatic mechanism for M2 pyruvate kinase as an amino-acid sensor. Biochemical Journal 475 1821-1837
- 2015 Pyruvate kinase: Function, regulation and role in cancer. Seminars in Cell & Developmental Biology 43 43–51
- 3me3 2012 Pyruvate kinase M2 activators promote tetramer formation and suppress tumorigenesis. Nature Chemical Biology 8 839-847
- 2009 Understanding the Warburg effect: The metabolic requirements of cell proliferation. Science 324 1029–1033
- 4fxf 2001 Five coplanar anion binding sites on one face of phospholipase A2: relationship to interface binding. Biochemistry 40 609-617
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









