270: 피루브산 키나아제M2 (Pyruvate Kinase M2)
이 기사는 정량생물의학 럿거스연구소(Rutgers Institute for Quantitative Biomedicine) 주최로 개최된1주일간의 대학생 및 대학원생을 위한 집중 합숙의 일환으로서 파이자 아흐메드(Faiza Ahmed), 조나단 애쉬(Jonathan Ash), 서스 패텔(Thirth Patel), 오릴 샌더스(Auriel Sanders)에 의해 집필되고 그려진 것으로, 2022-2023년 PDB-101의 건강 포커스 ‘암 생물학과 치료법’(Cancer Biology and Therapeutics)의 일부로도 게재되었습니다.
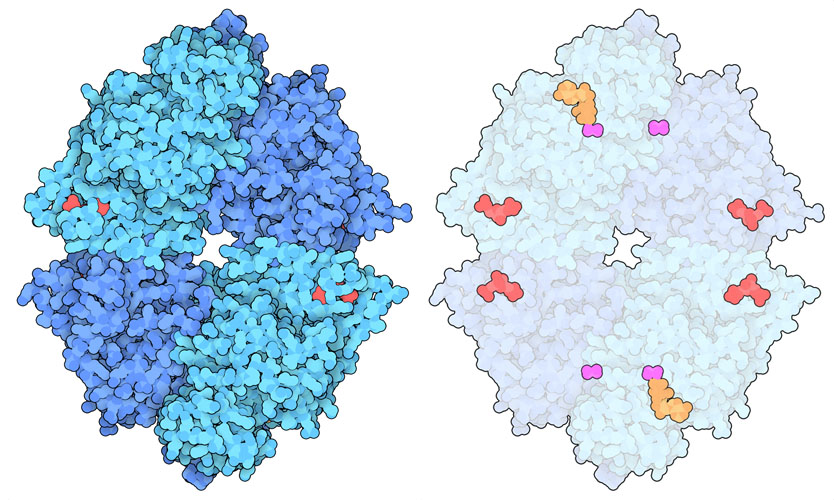
암세포는 자유롭게 증식할 수 있도록 하기 위해 근본적인 부분에서 많은 변화를 일으키고 있다. 놀랍게도 에너지를 만들어내는 경로의 재프로그래밍도 그러한 변화 중 하나이다. 정상 세포의 경우 포도당(glucose)을 에너지로 변환하는 과정은 세포질의 해당계(glycolysis)에서 시작된다. 해당계의 마지막에 생성되는 피루브산(pyruvate)은 일반적으로 미토콘드리아에 들어가 구연산 회로(citric acid cycle)로 진행된다. 산소가 있으면 이 회로는 전자전달계(electron transfer chain, 복합체 I, III, IV)와 ATP 합성효소(ATP synthase)의 작용에 의한 호기성 에너지 생산을 촉진한다. 암세포는 대량의 포도당을 흡수하지만 해당(解糖)의 마지막 단계는 차단하고 있다. 이들 세포로 만들어진 피루브산은 미토콘드리아로 들어가는 것이 아니라 젖산으로 변환된다.
피루브산 키나아제M2: 문지기
해당의 최종 단계는 피루브산 키나아제(pyruvate kinase, PDB ID 4fxf)라는 효소에 의해 촉매된다. 이 효소는 포스포에놀피루브산(phosphoenolpyruvate)에서 인산기를 전이시켜 ATP(세포의 에너지 통화)와 피루브산을 만드는 반응을 촉매하고 있다. 세포의 생존에 필수적이기 때문에 피루브산 키나아제의 활성은 엄격하게 제어되고 있다. 이 효소는 고활성형 4량체와 저활성형 2량체의 2종류의 집합체를 만든다. 보다 많은 에너지가 필요할 때는 플룩토오스 2인산(fructose bisphosphate, 해당계의 보다 이전 단계에서 생성되는 분자)이 4량체 형성을 촉진한다. 한편 다른 여러 제어 분자들이 특정 알로스테릭 부위(활성 부위와는 다른 곳이지만 활성에 영향을 주는 부위)에 결합해 2량체 형성을 촉진해 효소 활성을 억제할 수 있다. 체내에서는 4종류의 조직 특이적인 피루브산 키나아제가 만들어진다. PKM1은 많은 에너지를 필요로 하는 세포이고, PKR은 적혈구에서, PKL은 간세포에서, PKM2는 보통 배아세포나 기타 증식 중인 세포에서 만들어진다. PKM1 효소는 일반적으로 4량체 형태로 존재하며 항상 활성 상태로 되어 있다. 그러나 많은 알로스테릭 제어 분자가 PKM2 효소와 결합하고 효소가 4량체에서 2량체로의의 이행에 영향을 줌으로써 그 작용을 제어하고 있다. 암세포는 이 제어를 이용해 스스로 대사 구조를 다시 만들고 있다.
에너지 생산의 역설
많은 암세포는 효소인 피루브산 키나아제M2(PKM2)를 대량으로 만들어 에너지 생산 경로를 해당계로 이행시키고 있다. 이 대사를 다시 만드는 것은 2가지 이유에서 역설적이다. 첫째, 이 세포들은 산소가 풍부해도 호기성 에너지 생산을 하지 않는다. 둘째, 급속히 분열하는 이 세포들는 정상 세포보다 더 많은 에너지를 필요로 하는데 에너지 효율이 현저히 나쁜 해당을 주 에너지원으로 사용하고 있다. 이 상반된 거동에 대한 답은 PKM2의 속도를 저하시킴으로써 생기는 다른 대사 산물에 있다. 즉, 해당계에서의 전 단계에서 생기는 대사 중간체가 축적되는 데 있다. 이들 중간체는 많은 생합성 경로로 전용되며 암세포는 급속한 성장에 필요한 단백질이나 핵산의 구축 부재를 만들 수 있다. 이처럼 암세포는 에너지 생산보다 성장을 우선시한다. 이 같은 포도당 이용 재프로그래밍은 월버그 효과(Warburg Effect)라고 불린다. 이 명칭은 이를 발견한 연구자 오토 하인리히 월버그(Otto Heinrich Warburg)의 이름을 따서 지어졌다.
월버그 효과와 암 치료
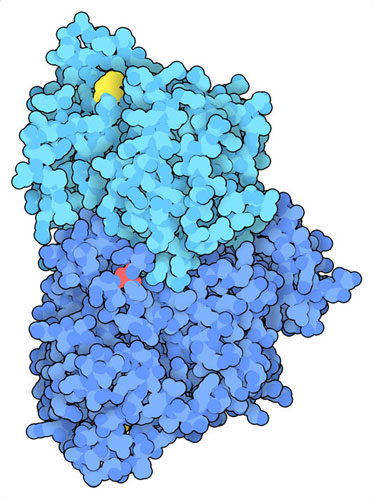
월버그 효과는 종양 형성에 있어서 공통적인 특징이다. 그 발견 이래, 암세포에서는 PKM2가 인산화 및 아세틸화되어 있는 것이 발견되고 있다(PDB 엔트리 6wp3). 이러한 수식은 플룩토오스 2인산 등의 활성화 인자가 결합하는 부위에 영향을 준다. 이러한 활성화 인자가 없으면 PKM2는 효율적으로 불활성인 4량체를 형성할 수 없다. 이 효소의 2량체는 해당(解糖)에서 활성이 낮을 뿐만 아니라 핵 내로 이동하여 다른 암을 일으키는 유전자 전사를 촉진할 수 있다. 이런 변화는 젖산 축적을 일으켜 종양의 진행과 암 치료에 대한 내성을 조장하게 된다. PKM2는 암세포의 재프로그래밍에 중요한 역할을 하기 때문에 현재 PKM2를 저해하는 약제나 치료법, 혹은 호기성 대사를 활성화하는 다른 방법을 모색하는 연구가 진행 중이다.
구조 보기
인터랙티브 조작이 가능한 페이지로 전환하려면 그림 아래의 버튼을 클릭하십시오. 읽기가 시작되지 않을 때는 그림을 클릭하십시오.
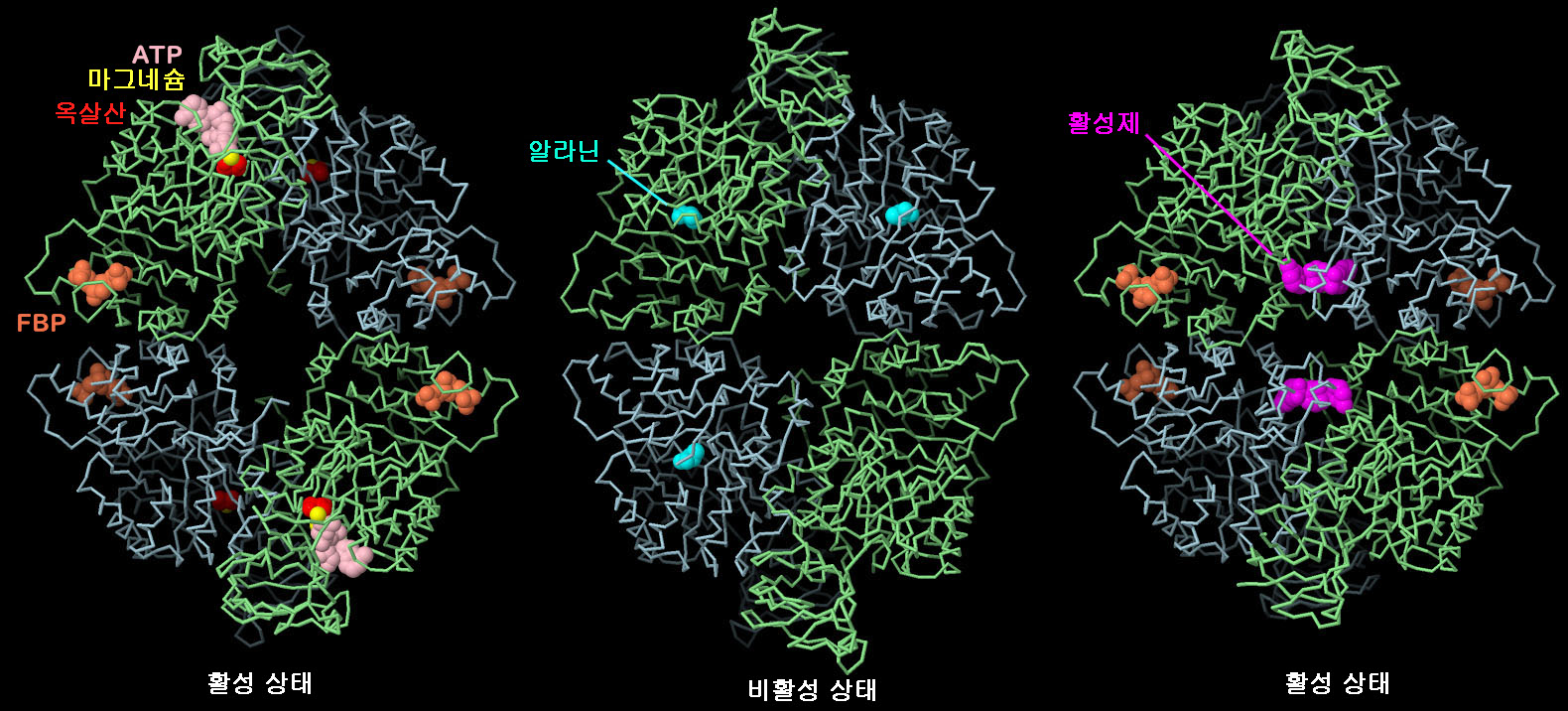
PKM2 4량체 구조에서 다양한 저분자가 알로스테릭 형상 변화를 일으켜 단백질 작용을 제어하는 것으로 나타났다. 왼쪽에 나타낸 것이 활성형(PDB ID 4fxf)으로 ATP, 수산(oxalate), 마그네슘(magnesium)이 활성 부위에, 플룩토오스 2인산(fructose bisphosphate FBP)이 제어 부위에 있다. 중앙에 나타낸 것이 불활성형(PDB ID 6gg3)으로 제어 부위에는 알라닌이 있다. 형상이 변화하고 중앙부가 가늘어진 것에 주목해 주기 바란다. 암 치료에 도움을 주기 위해 PKM2에 결합하는 약물 분자를 찾는 연구가 적극적으로 진행되고 있다. 예를 들어 오른쪽에 나타낸 구조(PDB ID 3me3)에서는 인공의 활성화 분자(자홍색)가 FBP와 함께 결합되어 있는데 이를 통해 분자를 활성형으로 변화시키고 이 분자로 처리한 세포에서 월버그 효과를 역전시킬 수 있을 것으로 기대되고 있다.
이해를 높이기 위한 토픽
- 유산 탈수소 효소(lactate dehydrogenase)를 저해함으로써 세포 증식을 지연시킬 수 있으며 암의 진행도 및 치료 효과 모니터링에 이용되고 있습니다. PDB 엔트리 4ajp에서 항암제의 시제품과 결합한 유산 탈수소 효소의 구조를 살펴보십시오.
- PDB 아카이브에는 인간에서 유래한 PKM2에 다양한 저분자가 결합된 구조가 다수 등록되어 있습니다. PDB 엔트리 4fxf를 포함하는 그러한 구조를 찾으려면 PDBj의 PDB 검색(PDBj Mine)에 액세스하여 "외부 데이터베이스"에서 "UniProt"를 선택하고 입력 상자에 "P14618"을 입력하여 검색한 결과를 보십시오.
참고문헌
- 2021 Revisiting the Warburg effect: Historical dogma versus current understanding. The Journal of Physiology 599 1745–1757
- 6wp3 2020 Structural basis for allosteric regulation of pyruvate kinase M2 by phosphorylation and acetylation. Journal of Biological Chemistry 295 17425–17440
- 2019 Pyruvate kinase M2: A simple molecule with complex functions. Free Radical Biology and Medicine 143 176–192
- 2019 Functional cross-talk between allosteric effects of activating and inhibiting ligands underlies PKM2 regulation. ELife 8 e45068 DOI:10.7554/eLife.45068
- 6gg3 2018 An allostatic mechanism for M2 pyruvate kinase as an amino-acid sensor. Biochemical Journal 475 1821-1837
- 2015 Pyruvate kinase: Function, regulation and role in cancer. Seminars in Cell & Developmental Biology 43 43–51
- 3me3 2012 Pyruvate kinase M2 activators promote tetramer formation and suppress tumorigenesis. Nature Chemical Biology 8 839-847
- 2009 Understanding the Warburg effect: The metabolic requirements of cell proliferation. Science 324 1029–1033
- 4fxf 2001 Five coplanar anion binding sites on one face of phospholipase A2: relationship to interface binding. Biochemistry 40 609-617
 의 생체 고분자 학습 포털 사이트
의 생체 고분자 학습 포털 사이트 









