102: 乳酸脱水素酵素(Lactate Dehydrogenase)
通常のペースで運動をする時、私たちの細胞は酸素を豊富に取り込んで糖を素早く効率的に分解する。ところが、全力疾走など激しい運動をすると、酸素が十分に行き渡らなくなる。そういう時、私たちの細胞はエネルギー源として解糖系を使う。解糖系の過程の中で、グルコース(ぶどう糖)から得られた水素はNAD+へと渡されて、NADHができる。通常の酸素呼吸の場合、水素はその後酸素に受け渡されて水になる。一方酸素が使えない時は、NADHが溜まってNAD+が足らなくなり、ATPを作るために解糖系を使い続けることはできなくなる。そこで乳酸脱水素酵素の出番である。この酵素はピルビン酸とNADHをくっつけて、乳酸(lactic acid)とNAD+を作り出す。この働きによってNAD+をリサイクルし、再び解糖系で再利用することで、全力疾走に必要な追加エネルギーを素早く作り出すことができるようになる。ただ、乳酸が溜まり数分もすると止まって身体を回復させないといけなくなる。この場合一息つけば、乳酸はピルビン酸に戻され、通常の有酸素的なエネルギー生産過程に入って行くことができる。
混合と適合
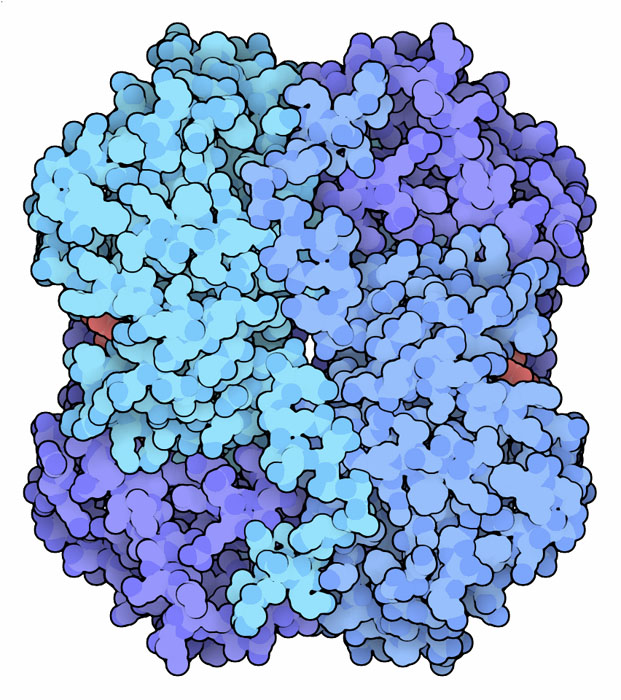
私たちの細胞は主に2種類の乳酸脱水素酵素〜M型とH型〜を作る(精子のみで見られる3番目の型もある)。両者は大きさも形も非常によく似ているが、異なった触媒の性質を持っている。M型は大きな骨格筋でよく見られる型で、ピルビン酸を乳酸に変換するのが得意である。この型は、筋肉が無酸素運動を行う必要があった時でも働けるよう待機している。一方H型は、M型とは逆に、乳酸をピルビン酸に変換する方が得意である。こちらは心臓でよく見られる型で、ここでは酸素供給が常にあり酸素呼吸によるエネルギー源として簡単に乳酸を利用できる。この2つの型は大変似た構造を持っているので、両方の型が混ざった複合体〜例えば2つのH鎖と2つのM鎖でできた複合体〜を形成する。このようにして、細胞個々時々に異なる要求事項に乳酸脱水素酵素は合わせられるのである。ここに示した分子はPDBエントリー 3ldhのもので、4つのM鎖が集まっている。
発酵
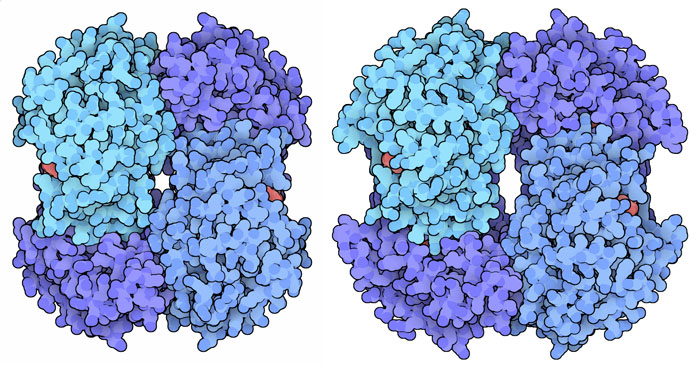
細菌の中にはエネルギーのほとんどをグルコースから乳酸への変換で得ているものがいる。この反応過程は発酵(fermentation)と呼ばれ、産物は夕食の食卓で目にしていることだろう。発酵を行う細菌は牛乳をヨーグルトに変えるのに使われ、発酵でできた乳酸はザウアークラウト(sauerkraut、塩漬けキャベツ)やサワードゥブレッド(sourdough bread、発酵パン)の鋭いにおいの主成分となっている。ここに示した細菌の乳酸脱水素酵素はアロステリック酵素(他の分子によって活性が変わる酵素)である。フルクトース1,6-2リン酸が結合すると、一方の分子は解糖系の初期段階を形成し、その結果酵素を活性型に変化させる。ここに示したPDBエントリー 1lthは、珍しく2つ別々に酵素分子の構造を含んでおり、左は活性状態、右は不活性状態の構造を示している。
構造をみる
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
マラリアを引き起こす寄生原虫は、感染サイクルのある期間、必要なエネルギーの大半を解糖系に依存して得ていると考えられている。現在研究者は、このマラリア原虫を攻撃し感染を治療する方法の1つとして乳酸脱水素酵素の活性を阻害する薬を探している。ここに示したPDBエントリー 1cetは、マラリア原虫の乳酸脱水素酵素の活性部位に4分子のクロロキン(chloroquine)が結合したものである。クロロキンは主なマラリア治療薬の1つだが、活動の場所はおそらくこの酵素にはない。寄生虫が血液を食べるのに必ず使っている通常みられない方法を阻害していると考えられている。研究者は乳酸脱水素酵素を対象とする抗マラリア薬を他にも多く調査しており、そのような分子として他にPDBエントリー 1t24、1t25およびこれらに似た構造分子がある。
キーワード「乳酸脱水素酵素」(lactate dehydrogenase)でPDBエントリーを検索した結果はこちら。
参考文献
当記事を作成するに当たって参照した文献は以下の通りです。
- D. L. Nelson and M. M. Cox 2000 Lehninger Principles of Biochemistry. Worth Publishers.
- M. T. Madigan, J. M. Martinko and J. Parker 2000 Brok Biology of Microorganisms. Prentice Hall.
- J. A. Read, K. W. Wilkinson, R. Tranter, R. B. Sessions and R. L. Brady 1999 Chloroquine binds in the cofactor binding site of Plasmodium falciparum lactate dehydrogenase. Journal of Biological Chemistry 274 10213-10218
- S. Iwata, K. Kamata, S. Yoshida, T. Minowa and T. Ohta 1994 T and R states in the crystals of bacterial L-lactate dehydrogenase reveal the mechanism for allosteric control. Structure 1 176-185
- J. J. Holbrook, A. Liljas, S. J. Steindel and M. G. Rossman 1975 Lactate Dehydrogenase. The Enzymes. P. D. Boyer, editor. Academic Press. Volume XI 191-292
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









