283: c-Abl蛋白激酶和伊馬替尼(c-Abl Protein Kinase and Imatinib)
這篇文章和文章中的插圖是由Allison Abel,Darlene R. Malave-Ramos,Bhavya Soni,AChristopher Thai和Amy Wu-Wu撰寫和製作的,作為羅格斯定量生物醫學研究所(Rutgers Institute for Quantitative Biomedicine )組織的為期一周的本科生和研究生強化訓練營的一部分。這也作為2023-2024年PDB-101健康重點"癌症生物學和治療學"(health focus on "Cancer Biology and Therapeutics")的一部分發表。
蛋白激酶(protein kinase)最初被避開作為癌症化療的標靶。眾所周知,蛋白激酶是絕佳的靶點,因為它們在細胞功能的各個方面都發揮著重要作用,並受到嚴格調控,任何調控的改變都可能導致癌症和其他疾病的發生。然而,細胞中有許多不同類型的激酶,它們在結構上相似,與ATP的結合位點也相似。長期以來,人們認為這些相似性將不可避免地導致脫靶結合,從而導致任何針對這些激酶活性位點的藥物產生副作用。
結構打開大門
幸運的是,結構生物學證明這些最初的假設是錯誤的。隨著蛋白激酶結構的闡明,人們發現它們的ATP結合口袋有足夠的差異,有助於結構引導選擇性藥物的發現和開發。抗癌藥物伊馬替尼(imatinib)是一個具有里程碑意義的成功案例,自此之後,各種蛋白激酶的選擇性小分子抑制劑被成功開發出來。其中許多都是結構引導藥物發現的結果。
紊亂但不失調
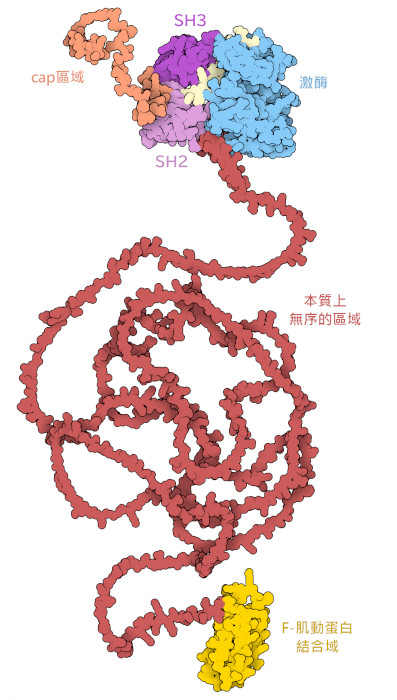
伊馬替尼以c-Abl為靶點,c-Abl是一種酪胺酸激酶,可選擇性地將磷酸基從ATP轉移至酪胺酸。從計算出的全長蛋白質模型(PDB條目AF_AFP00519F1)中可以看出,c-Abl是一個由多個功能部分組成的複合物。兩個區域,激酶和調節結構域(kinase and regulatory domains,PDB條目1opl)和F-肌動蛋白結合結構域(F-actin binding domain,PDB條目1zzp),是穩定的折疊結構。使用X射線晶體學和核磁共振光譜進行詳細研究表明,它們通過一個長的、固有的非恆定構象連接在一起,但保持高度柔性。一端是短的柔性尾部,稱為帽區,與肉荳蔻酰基(myristoyl group)相連。帽區具有蛋白質的鎖定機制,控制蛋白質的功能。在非活性狀態下,肉荳蔻酰基與激酶結構域中的一個口袋結合,導致構象變化,使激酶結構域與調節結構域緊密結合,阻止催化活性。為了活化蛋白,外部細胞訊號與c-Abl的調控結構域相互作用,從激酶結構域釋放肉荳蔻酰基,暴露出活性位點,使特定底物磷酸化。
癌症組合
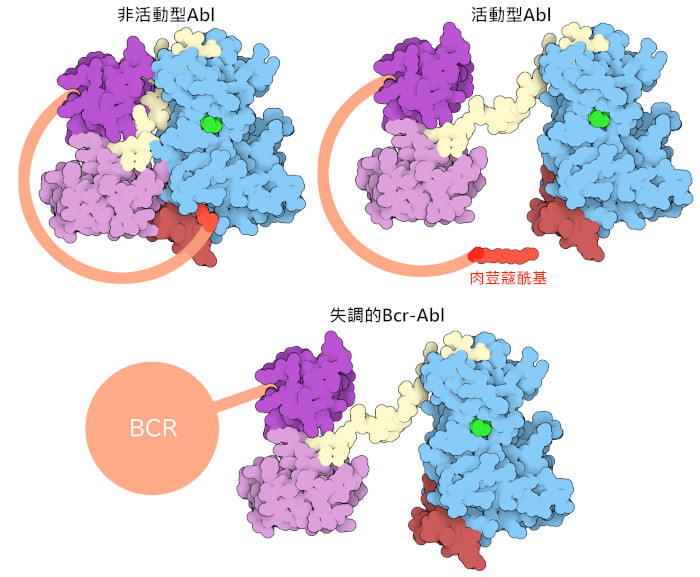
c-Abl酪胺酸激酶的基因異常與慢性骨髓性白血病(chronic myelogenous leukemia,CML)有關,這是一種骨髓中未成熟細胞的癌症。費城染色體(Philadelphia chromosome),當9號染色體的一部分被22號染色體的一部分取代時就形成了費城染色體。這種平衡的基因轉位融合了c-abl和bcr基因,從而產生了一種名為Bcr-Abl的獨立蛋白質。這種突變的酪胺酸激酶有一個自身抑製帽區,沒有肉荳蔻酰基,因此它始終處於活性狀態,不需要正常細胞訊號的活化。這使得Bcr-Abl能夠繼續分裂;Bcr-Abl還能阻止細胞凋亡(apoptosis,細胞誘導自身死亡的過程),細胞透過凋亡來防止癌細胞的累積;Bcr-Abl的作用會導致不成熟的、部分功能的血液凋亡。由於Bcr-Abl的作用,未成熟的、部分功能的血細胞的累積會對被診斷為CML的患者產生破壞性影響,最終導致急性白血病(acute leukemic condition),稱為急性轉化期(blast phase,爆發期)。美國食品藥物管理局(United States Food and Drug Administration)批准的伊馬替尼和其他Bcr-Abl抑制劑的治療已將10年存活率大幅提高至約85%。許多CML患者現在的預期壽命與健康成人大致相同。
探索結構
若要切換到有互動控制的頁面,請點擊圖表下方的按鈕。如果載入沒有開始,請嘗試點擊圖表。
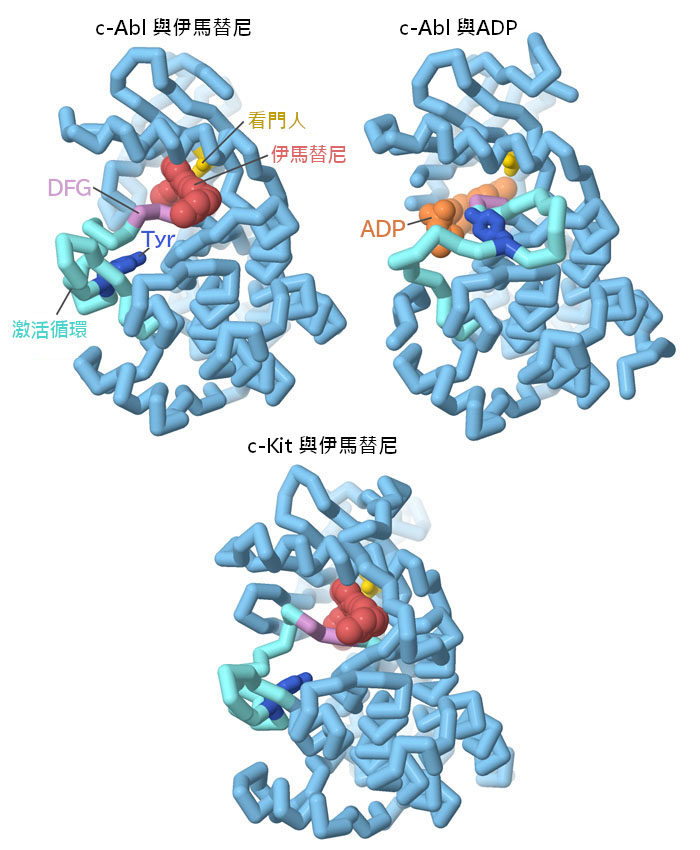
伊馬替尼阻斷c-Abl激酶結構域中的ATP結合位點並停止其作用,從激酶結構中可以看出,包括ADP(PDB條目2g2i)和伊馬替尼(PDB條目2hyy),激酶中的幾個區域對其功能和伊馬替尼的抑製作用非常重要。 "活化環"(activation loop)覆蓋活性位點並控制活性位點的進入。它也受酪胺酸的調控,酪胺酸被磷酸化。 "守門員"蘇氨酸、"DFG基序"和幾個氫鍵氨基酸(此處未顯示)參與了與伊馬替尼的特異性相互作用,有助於提高藥物選擇性。然而,伊馬替尼並非完全具有選擇性,但幸運的是,在某些情況下可以利用這一點。伊馬替尼也可以抑制c-Kit(PDB條目1t46),這是一種導致胃腸道間質瘤(gastrointestinal stromal tumor,GIST)的癌症蛋白酪胺酸激酶。伊馬替尼對這兩種激酶都起作用。點擊圖表下方的按鈕,切換到互動圖片,更詳細地了解這些結構。
進一步的討論議題
References
- 2021 Kinase drug discovery 20 years after imatinib: progress and future directions. Nat Rev Drug Discovery 20 551–569
- 2020 Impact of the Protein Data Bank on antineoplastic approvals. Drug Discovery Today 25 837–850
- 2017 Structural and functional dissection of the DH and PH domains of oncogenic Bcr-Abl tyrosine kinase. Nat Commun 8 1–14
- 2013 Conformation-selective inhibitors reveal differences in the activation and phosphate-binding loops of the tyrosine kinases Abl and Src. ACS Chem Biol 8 2734–2743
- 2HYY 2006 Structural biology contributions to the discovery of drugs to treat chronic myelogenous leukaemia. Acta Cryst D Biol Crystallog 63 80–93
- 2G2I 2006 A src-like inactive conformation in the ABL tyrosine kinase domain. PLoS Biol 4 e144
- 2006 Organization of the SH3-SH2 unit in active and inactive forms of the c-Abl tyrosine kinase. Molecular Cell 21 787–798
- 1ZZP 2005 Structural basis for the cytoskeletal association of Bcr-Abl/c-Abl. Mol Cell 19 461–473
- 1XBB 2004 A novel mode of Gleevec binding is revealed by the structure of spleen tyrosine kinase. J Biol Chem 279 55827–55832
- 1T46 2004 Structural basis for the autoinhibition and STI-571 inhibition of c-kit tyrosine kinase. J Biol Chem 279 31655–31663
- 1OPL 2003 Structural basis for the autoinhibition of c-Abl tyrosine kinase. Cell 112 859–871
- 2002 Structure of the Bcr-Abl oncoprotein oligomerization domain. Nat Struct Biol 9 117–120
 生物大分子學習的教育門戶
生物大分子學習的教育門戶 









