271: 비상동말단연결 슈퍼 복합체 (Non-Homologous End Joining Supercomplexes)
이 기사는 정량생물의학 럿거스연구소(Rutgers Institute for Quantitative Biomedicine) 주최로 개최된1주일간의 대학생 및 대학원생을 위한 집중 합숙의 일환으로서 가브리엘라 디아즈 피게로아(Gabriela Diaz-Figueroa), 마이클 에고지(Michael Egozi), 사이다 자네스(Syeda Jannath), 재스민 마디(Jasmine Maddy)에 의해 집필되고 그려진 것으로, 2022-2023년 PDB-101의 건강 포커스 ‘암 생물학과 치료법’(Cancer Biology and Therapeutics)의 일부로도 게재되었습니다.
꼭 필요한 DNA 복구
DNA는 우리 유전정보의 담당자다. 매우 중요하기 때문에 우리 DNA는 그 사슬이 끊어져도 자기 복원을 하는 지령을 내릴 수 있다. DNA의 절단은 복제 시의 오류나 방사선, 산화 스트레스, 반응성 화학물질 등 외적 요인에 의한 손상 등 다양한 형태로 일어난다. 끊어진 DNA가 하나뿐일 때는 핵 내의 복구 시스템이 상보사슬을 가이드로 삼아 어렵지 않게 틈새를 메워간다. 하지만 이중가닥이 파괴되면 복구가 어렵고 그대로 두면 세포는 죽게 된다. 비상동말단연결(Non-Homologous End Joining, NHEJ)은 끊어진 말단을 다시 연결해 이처럼 유해한 오류를 수정하는 주요 방법 중 하나이다. 우리 일상생활에서 하루에 10여 개의 이중사슬 절단이 각 세포에서 일어나기 때문에 이 과정은 필수적이다.
비상동말단연결의 처리 흐름
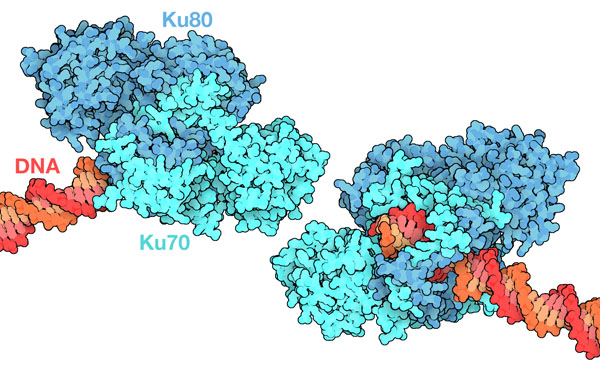
비상동말단연결은 다양한 종류의 복구 단백질에 의해 이루어진다. 이 단백질들은 파괴된 DNA 쪽으로 단계적으로 모여 초복합체를 형성하고 이것이 DNA를 다시 연결한다. 이 초복합체의 형성은 Ku70과 Ku80이라는 2개의 단백질의 헤테로 2량체에서 시작된다. 이것이 끊어진 DNA 끝을 특정해 다른 NHEJ 단백질을 불러들인다. 여기에 나타낸 구조는 Ku70과 Ku80이 절단된 말단에 결합한 초복합체 형성 과정의 초기 단계를 포착한 것이다(PDB 엔트리 1jey). 그 다음 DNA-PKcs(PDB 엔트리 6zhe 참조, 여기에는 도시하지 않음)가 결합하여 다른 많은 효소들을 불러 모아 2개의 끊어진 끝을 가지런히 잘라 연결할 준비를 한다. 2개의 말단은 XRCC4와 XLF에 의해 맞대어져 마지막으로 DNA리가아제IV(DNA ligase IV)가 파괴된 DNA 사슬을 접착해 복구된 DNA가 방출된다.
끊어진 이중가닥 DNS를 복구하는 구조
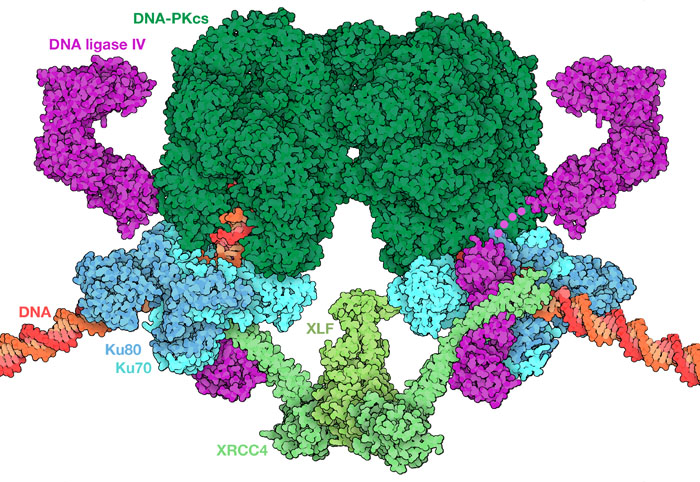
이중가닥 절단은 양 말단의 공간적인 분리, 염기의 수식, 절단된 말단에서의 단일가닥 돌출 등 다양한 문제를 야기한다. 이에 대처하기 위해 DNA-PKcs는 초복합체(여기에 나타낸 것은 PDB 엔트리 7nfc와 3w1b)에 결합하여 다양한 종류의 손상을 복구하고 DNA의 말단끼리를 맞대는 처리를 하는 다른 효소군을 불러 모은다. 여기서 모아지는 효소에는 빠진 뉴클레오타이드를 채우는 여러 종류의 작은 중합효소(polymerase)나 아르테미스(Artemis) 등 돌출된 사슬을 잘라내는 작용을 하는 뉴클레아제(nuclease)가 포함된다. 손상된 말단에서 이러한 여러 가지 복구를 수행하기 때문에 NHEJ가 수행하는 처리는 정확하지 않고 접합 부위에서 작은 실수를 일으킬 수 있다.
결합을 만들다
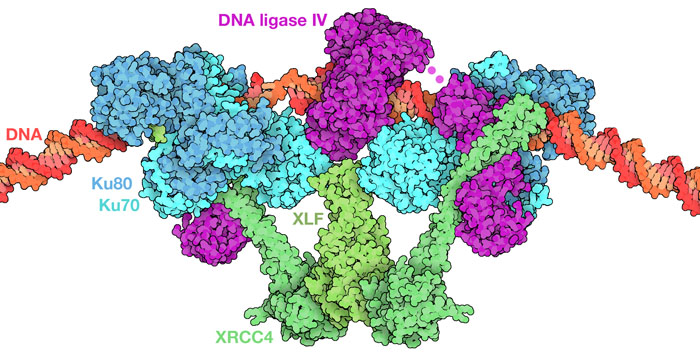
마지막으로 말단의 준비가 정돈되면 집합체는 재배치되고 DNA리가아제 IV가 DNA를 재접속한다(PDB 엔트리 7lsy). 이중가닥 절단은 생물에게 유해함에도 불구하고 유익한 경우도 있다. 예를 들어 우리 자신의 면역계에서는 항체 유전자가 절단되고 NHEJ에 의해 재조합되어 다양성이 생성되고 있다. 이를 통해 새로운 병원체를 인식하는 기존에 없던 조합이 발견된다. 이중가닥 절단은 암의 방사선요법과 화학요법에서도 중요한 역할을 담당하고 있다. 이들 치료법은 표적으로 삼는 종양세포의 DNA를 절단해 효과적으로 종양세포를 사멸시키거나 성장을 지연시킨다. 그러나 안타깝게도 오류를 일으키기 쉽다는 NHEJ의 성질은 세포의 성장과 제어에 관련된 중요한 유전자에 발암성 오류를 초래함으로써 암의 원인이 될 수 있다.
구조 보기
인터랙티브 조작이 가능한 페이지로 전환하려면 그림 아래의 버튼을 클릭하십시오. 읽기가 시작되지 않을 때는 그림을 클릭하십시오.
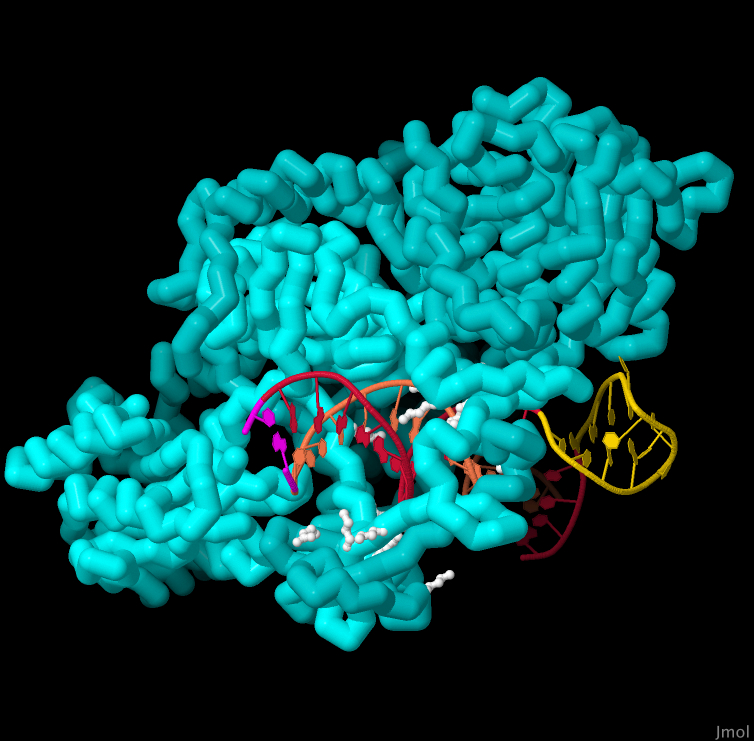
Ku 단백질은 파괴된 DNA 사슬을 어떻게 인식하느냐는 분자의 성가신 문제를 해결한다. 그것은 형상을 이용하여 수행되고 있다. DNA는 긴 사슬이며, 일반적으로 단백질은 그 주위를 둘러싸야 한다. 한편 Ku는 닫힌 링처럼 생겼기 때문에 DNA는 가운데 구멍을 통과해야 한다. 이것은 사슬의 끝에서만 할 수 있는 일이다. 여기에 나타낸 구조(PDB ID 1jey)는 Ku70과 Ku80의 헤테로 2량체로 안에 있는 공동(空洞)에 DNA의 짧은 단편이 지나간 상태를 보여준다. 양전하를 가진 리딘이나 아르기닌의 아미노산(흰색)이 몇 개 공동에 배열되어 DNA의 주사슬과 상호 작용을 한다. 끝이 모인 DNA의 말단은 자홍색으로 나타낸다. 짧은 머리핀(노란색)은 구조를 결정하기 쉽게 하기 위해 DNA에 부가된 것으로 DNA를 한 곳에 고정하는 역할을 한다.
이해를 높이기 위한 토픽
참고문헌
- 7nfc 2021 Cryo-EM of NHEJ supercomplexes provides insights into DNA repair. Mol Cell 81 3400
- 7lsy 2021 Structural basis of long-range to short-range synaptic transition in NHEJ. Nature 593 294-298
- 2020 The molecular basis and disease relevance of non-homologous DNA end joining. Nat Rev Mol Cell Biol 21 765–781
- 2017 Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nat Rev Mol Cell Biol 18 495-506
- 3w1b 2013 Structure of the catalytic region of DNA ligase IV in complex with an artemis fragment sheds light on double-strand break repair. Structure 21 672-679
- 1jey 2001 Structure of the Ku heterodimer bound to DNA and its implications for double-strand break repair. Nature 412 607-614
 의 생체 고분자 학습 포털 사이트
의 생체 고분자 학습 포털 사이트 









