287: ZAR1抗病小體(ZAR1 Resistosome)
與包括人類在內的所有其他生物一樣,植物也會受到病原體的感染,並患上疾病。這是農業和糧食生產面臨的嚴重問題,因為病害會使整個農作物絕收。幸運的是,植物已經有了自己的免疫防禦系統來保護自己免受這些外部威脅,但與人類不同的是,人類同時擁有適應性免疫和先天性免疫(adaptive and innate immunities),而植物只有先天性免疫。先天性免疫系統依賴細胞表面和細胞內部的受體分別檢測與病原體相關的特定分子模式和效應分子。一旦檢測到,就會觸發一系列事件,最終導致免疫反應,以擊退病原體並維持生存。擬南芥( Arabidopsis thaliana )是一種常見的雜草植物,被研究人員廣泛當作研究模型,在這裡,我們將探討擬南芥形成巨型分子機器抗病小體(resistosome)的途徑之一。
尋找病原體
與其他免疫反應一樣,抗病小體的形成途徑始於感染本身。具體來說,致病細菌野油菜黃單胞菌( Xanthomonas campestris )是葉片"黑腐病"的主要病原菌,它通過用尿苷酸轉移酶(uridylyltransferase) AvrAC感染植物細胞來發起進攻。尿苷酰轉移酶是一組酶,顧名思義,將「尿苷酰」核苷酸「轉移」到目標蛋白或核酸鏈上,通常是將其連接到具有可用羥基的氨基酸(例如蘇氨酸或絲氨酸)上。一旦進入植物細胞,AvrAC 就會透過在調節激酶上添加尿苷基的方式來開始攻擊,從而抑制免疫訊號通路,進而導致毒力增加。然而,巧妙的是,植物細胞會以一種名為PBL2 的誘餌蛋白進行反擊;當PBL2 被尿苷酰化時,它不會關閉免疫反應,而是開始建構抗性體的過程。如下面透過互動式JSmol 可視化的「探索結構」部分所示。
阻力片
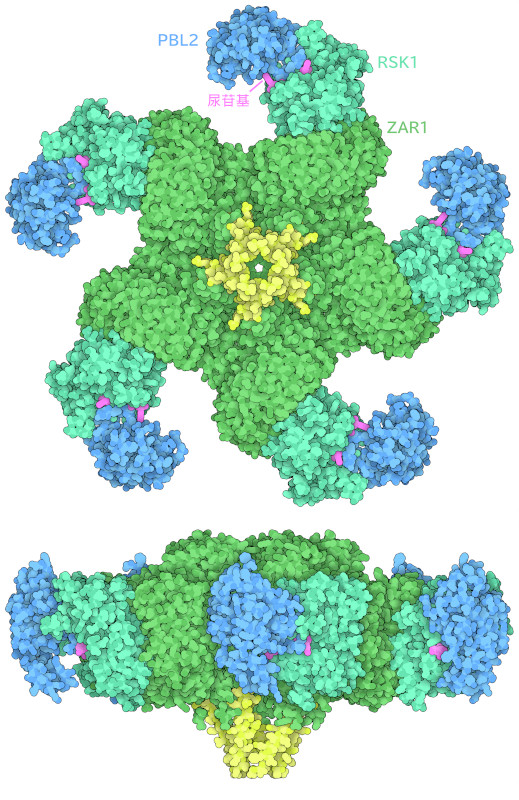
觀察抗原酶體的俯視圖(此處顯示的PDB 條目6j5t 的結構)可以發現,抗原酶體具有環狀結構,由三種不同的蛋白質各五個組成。環的外緣是一個尿苷化的PBL2 亞基(深藍色),每個亞基都朝向中心與另一個稱為RSK1 的亞基(假激酶;碧藍色)結合。環內有一個名為ZAR1 的免疫受體亞基(綠色),它是五個複合物結合的連接點。值得注意的是,在抗原基因組的最中心,ZAR1 亞基(黃色)的兩端在聚合體的一側,形成了一個漏斗狀的突起,如側視圖所示。研究發現,這個漏斗能發揮ZAR1 抗性體的重要保護功能,它能插入質膜形成一個孔,導致局部程序性細胞的死亡,即所謂的"超敏反應"。之所以假定這種插入機制,是因為漏斗外側相對疏水,與質膜疏水性的內部相互作用,而漏斗內側中空且親水,使其適合細胞質環境。
尿苷酸化調節
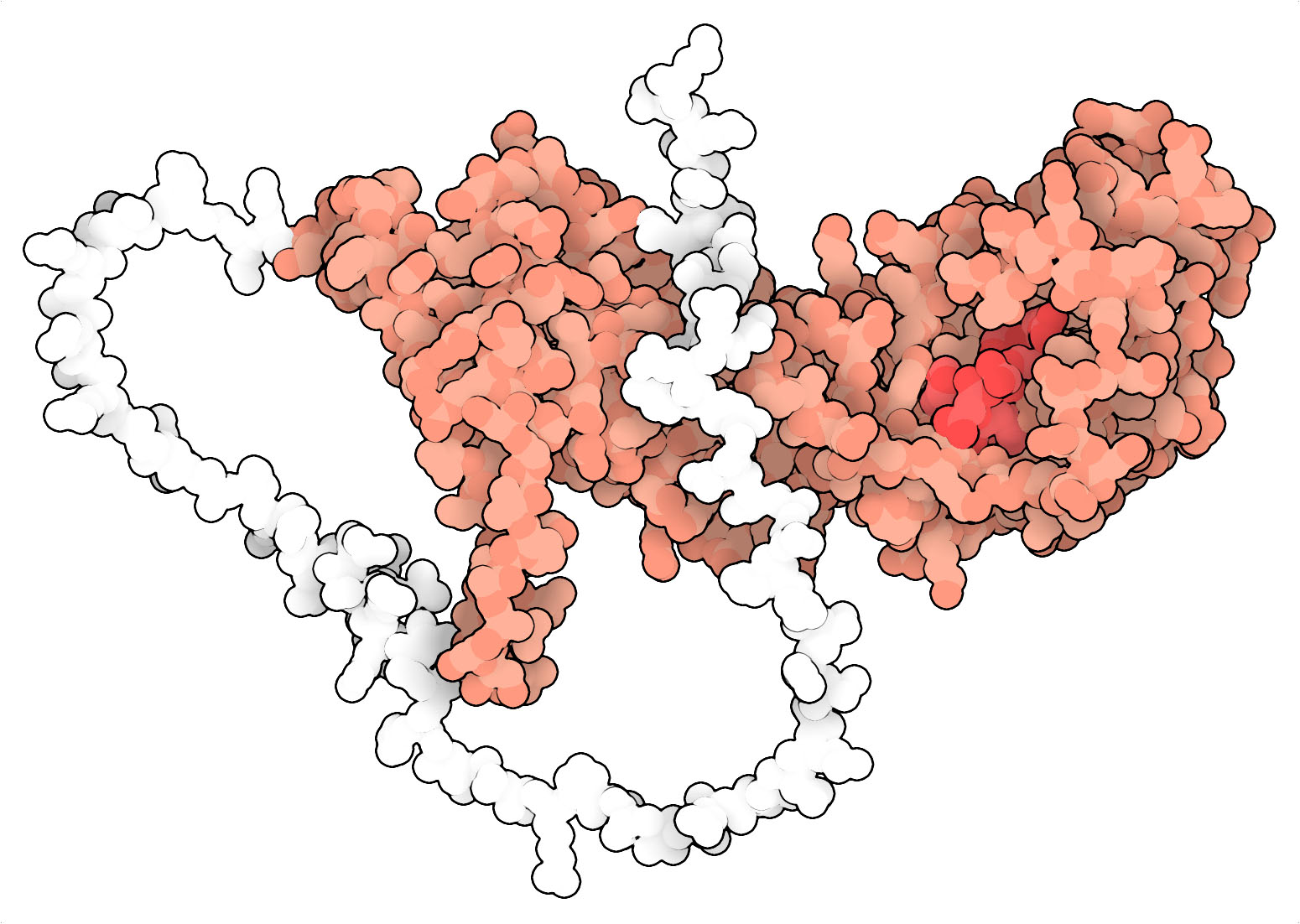
此處顯示的AF-Q4UWF4-F1的AvrAC 計算結構模型由兩個基本部分組成:激酶結合域(紅色)和無序長尾(白色)。激酶結合域不僅有助於AvrAC 與PBL2(誘餌蛋白)結合,也負責AvrAC 的尿苷酸化活性。這種活性是由一個稱為"Fic "結構域(暗紅色突出顯示)的小基團賦予的,它可以介導核苷酸的結合和轉移。重要的是,AvrAC 必須對PBL2 上的兩個位點(一個絲氨酸和一個蘇氨酸)進行尿苷酸化,才能觸發抗原體形成途徑級聯。
探索結構
若要切換到有互動控制的頁面,請點擊圖表下方的按鈕。如果載入沒有開始,請嘗試點擊圖表。
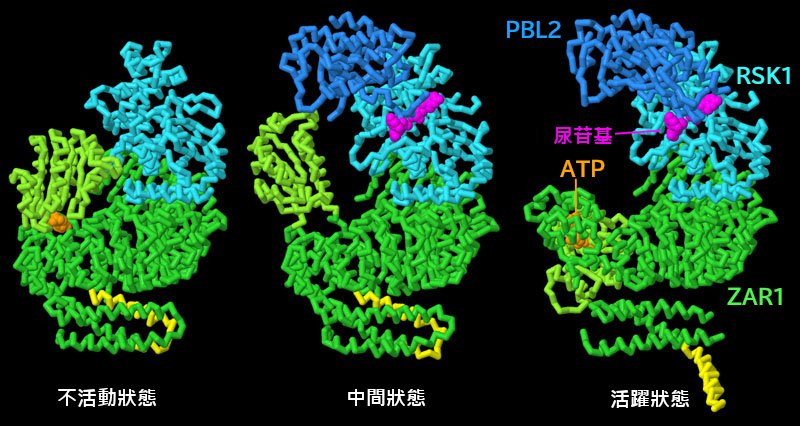
這三種結構揭示了建構活性抗原體過程中的各個步驟。首先,由與ADP(橘色)結合的免疫感受器ZAR1(綠色)和偽激酶RSK1(綠松石色)組成的複合物以非活性狀態存在於植物細胞中(PDB ID 6j5w),如左圖所示;當PBL2(深藍色)被雙尿酰化時,它會與RSK1 結合,形成中間所示的較大的中間複合物(PDB ID 6j5v),該複合物打開並允許ADP 釋放。這種開放的中間狀態是由RSK1 的兩個尿苷基團(洋紅色)和環狀區域之間的穩定相互作用誘導的,最終打開ATP/ADP 結合域(淺綠色),並導致ZAR1 結構域(綠色)發生構象變化。最後,ZAR1 亞基 結合ATP(橘色),引起其他異構構象變化,如釋放短α 螺旋尾部(黃色),最終形成右圖所示的活化複合物(PDB ID 6j5t)。當五個這樣的活化複合物聚集在一起完成電阻體時,黃色的尾部就形成了一個膜間相互作用的漏斗。
進一步的討論議題
- 探討其他生物如何利用Toll樣受體(toll-like receptor)和凋亡體聚合体(apoptosome assembly)等大分子超級複合物來控制免疫和程序性細胞死亡。
- 有關計算結構模型(CSM) 以及如何在RCSB.org 上進行探索的更多信息,請參閱文檔頁面。
进一步的讨论议题
- 2012 A Xanthomonas uridine 5′-monophosphate transferase inhibits plant immune kinases. Nature 485 114–118
- 2015 The decoy substrate of a pathogen effector and a pseudokinase specify pathogen-induced modified-self recognition and immunity in plants. Cell Host & Microbe 18 285–295
- 2019 The plant “resistosome”: structural insights into immune signaling. Cell Host & Microbe 26 193–201
- 2021 The ZAR1 resistosome is a calcium-permeable channel triggering plant immune signaling. Cell 184 3528–3541.e12
- 6j5t 2019 Reconstitution and structure of a plant NLR resistosome conferring immunity. Science 364 eaav5870
- 6j5v, 6j5w 2019 Ligand-triggered allosteric ADP release primes a plant NLR complex. Science 364 eaav5868
- AF-Q4UWF4-F1 2022 AlphaFold Protein Structure Database: massively expanding the structural coverage of protein-sequence space with high-accuracy models. Nucleic Acids Research 50 D439–D444
 生物大分子學習的教育門戶
生物大分子學習的教育門戶 









