287: ZAR1抗病小体(ZAR1 Resistosome)
与包括人类在内的所有其他生物一样,植物也会受到病原体的感染,患上疾病。这是农业和粮食生产面临的一个严重问题,因为病害会使整个农作物绝收。幸运的是,植物已经有了自己的免疫防御系统来保护自己免受这些外部威胁,但与人类不同的是,人类同时拥有适应性免疫和先天性免疫(adaptive and innate immunities),而植物只有先天性免疫。先天性免疫系统依靠细胞表面和细胞内部的受体分别检测与病原体相关的特定分子模式和效应分子。一旦检测到,就会触发一系列事件,最终导致免疫反应,以击退病原体并维持生存。拟南芥(Arabidopsis thaliana)是一种常见的杂草植物,被研究人员广泛当作研究模型,在这里,我们将探讨拟南芥形成巨型分子机器抗病小体(resistosome)的途径之一。
寻找病原体
与其他免疫反应一样,抗病小体的形成途径始于感染本身。具体来说,致病细菌野油菜黄单胞菌(Xanthomonas campestris)是叶片 "黑腐病"的主要病原菌,它通过用尿苷酸转移酶(uridylyltransferase) AvrAC感染植物细胞来发起进攻。尿苷酰转移酶是一组酶,顾名思义,将"尿苷酰"核苷酸"转移"到靶蛋白或核酸链上,通常是将其连接到具有可用羟基的氨基酸(例如苏氨酸或丝氨酸)上。 一旦进入植物细胞,AvrAC 就会通过在调节激酶上添加尿苷基的方式来开始攻击,从而抑制免疫信号通路,进而导致毒力增加。 然而,巧妙的是,植物细胞会以一种名为 PBL2 的诱饵蛋白进行反击;当 PBL2 被尿苷酰化时,它不会关闭免疫反应,而是开始构建抗性体的过程。如下面通过交互式 JSmol 可视化的"探索结构"部分所示。
阻力片
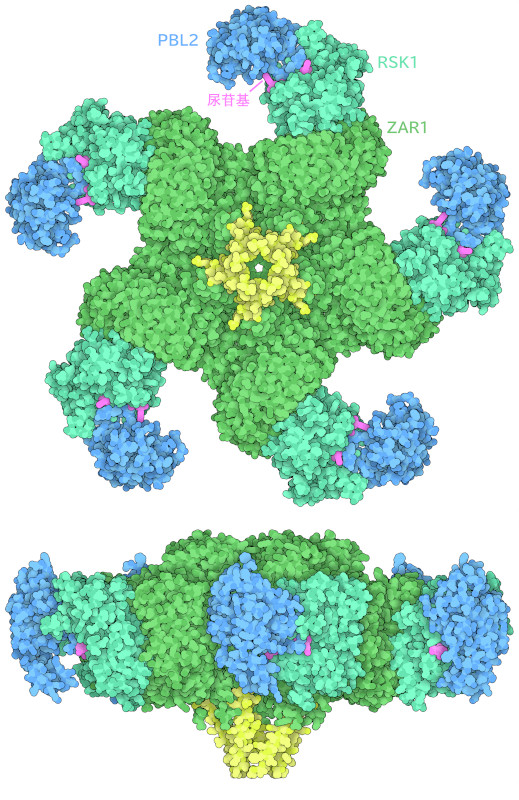
观察抗原酶体的俯视图(此处显示的 PDB 条目 6j5t 的结构)可以发现,抗原酶体具有环状结构,由三种不同的蛋白质各五个组成。环的外缘是一个尿苷化的 PBL2 亚基(深蓝色),每个亚基都朝向中心与另一个称为 RSK1 的 亚基(假激酶;碧蓝色)结合。环内有一个名为 ZAR1 的免疫受体 亚基(绿色),它是五个复合物结合的连接点。值得注意的是,在抗原基因组的最中心,ZAR1 亚基(黄色)的两端在聚合体的一侧,形成了一个漏斗状的突起,如侧视图所示。研究发现,这个漏斗能发挥 ZAR1 抗性体的重要保护功能,它能插入质膜形成一个孔,导致局部程序性细胞的死亡,即所谓的 "超敏反应"。之所以假定这种插入机制,是因为漏斗外侧相对疏水,与质膜疏水性的内部相互作用,而漏斗内侧中空且亲水,使其适合细胞质环境。
尿苷酸化调节
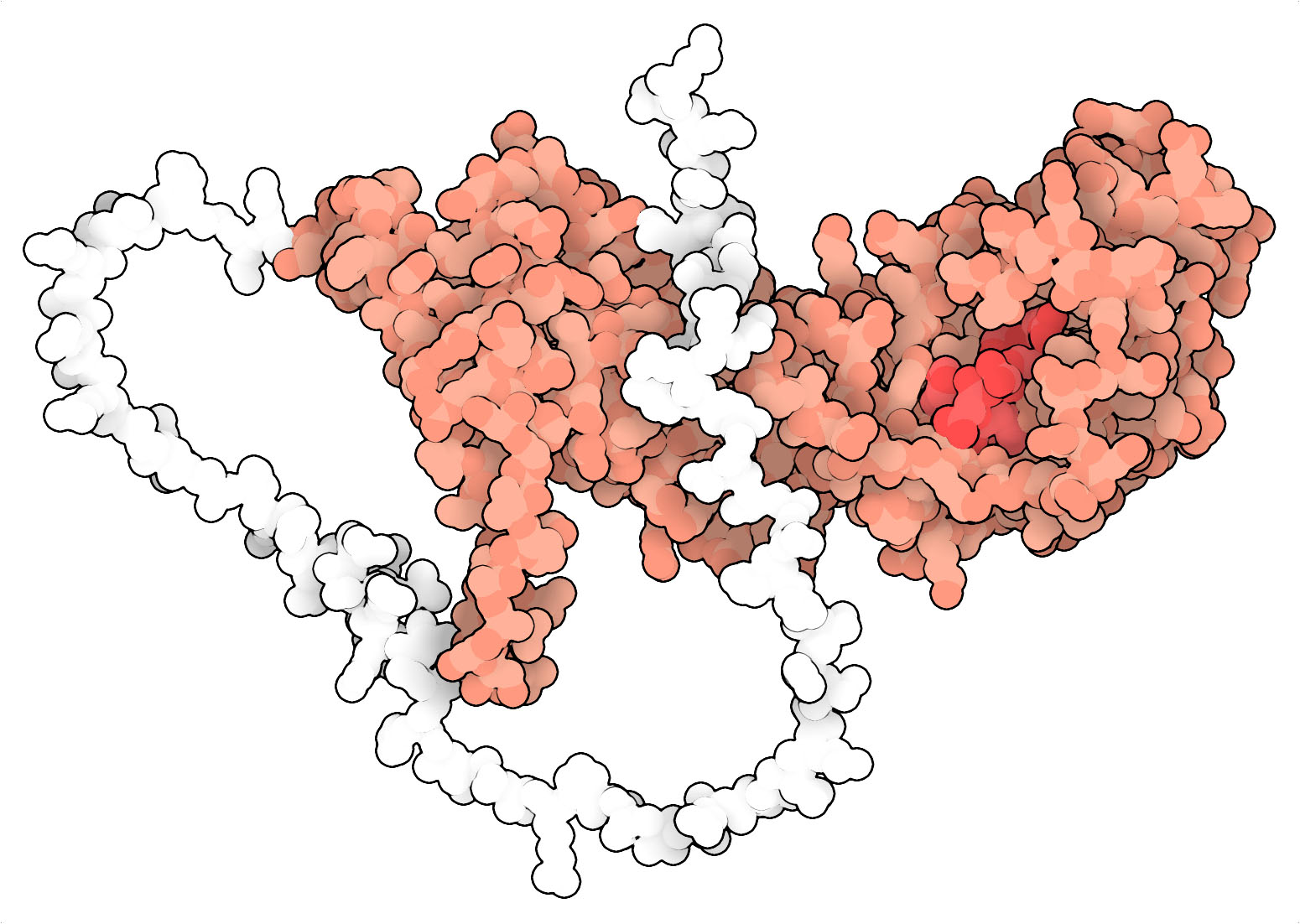
此处显示的 AF-Q4UWF4-F1 的AvrAC计算结构模型由两个基本部分组成:激酶结合域(红色)和无序长尾(白色)。激酶结合域不仅有助于 AvrAC 与 PBL2(诱饵蛋白)结合,而且还负责 AvrAC 的尿苷酸化活性。这种活性是由一个称为 "Fic "结构域(暗红色突出显示)的小基团赋予的,它可以介导核苷酸的结合和转移。重要的是,AvrAC 必须对 PBL2 上的两个位点(一个丝氨酸和一个苏氨酸)进行尿苷酸化,才能触发抗原体形成途径级联。
探索结构
要切换到有互动控制的页面,请点击图表下面的按钮。如果加载没有开始,请尝试点击图表。
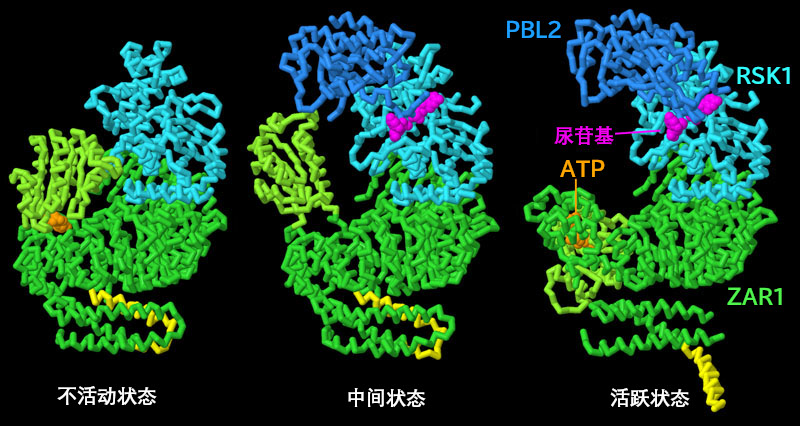
这三种结构揭示了构建活性抗原体过程中的各个步骤。首先,由与 ADP(橙色)结合的免疫感受器 ZAR1(绿色)和伪激酶 RSK1(绿松石色)组成的复合物以非活性状态存在于植物细胞中(PDB ID 6j5w),如左图所示;当 PBL2(深蓝色)被双尿酰化时,它会与 RSK1 结合,形成中间所示的较大的中间复合物(PDB ID 6j5v),该复合物打开并允许 ADP 释放。这种开放的中间状态是由 RSK1 的两个尿苷基团(洋红色)和环状区域之间的稳定相互作用诱导的,最终打开 ATP/ADP 结合域(浅绿色),并导致 ZAR1 结构域(绿色)发生构象变化。最后,ZAR1 亚基 结合 ATP(橙色),引起其他异构构象变化,如释放短 α 螺旋尾部(黄色),最终形成右图所示的激活复合物(PDB ID 6j5t)。当五个这样的活化复合物聚集在一起完成电阻体时,黄色的尾部就形成了一个膜间相互作用的漏斗。
进一步的讨论议题
- 探索其他生物如何利用 Toll样受体(toll-like receptor)和凋亡体聚合体(apoptosome assembly)等大分子超级复合物来控制免疫和程序性细胞死亡。
- 有关计算结构模型 (CSM) 以及如何在 RCSB.org 上进行探索的更多信息,请参阅文档页面。
进一步的讨论议题
- 2012 A Xanthomonas uridine 5′-monophosphate transferase inhibits plant immune kinases. Nature 485 114–118
- 2015 The decoy substrate of a pathogen effector and a pseudokinase specify pathogen-induced modified-self recognition and immunity in plants. Cell Host & Microbe 18 285–295
- 2019 The plant “resistosome”: structural insights into immune signaling. Cell Host & Microbe 26 193–201
- 2021 The ZAR1 resistosome is a calcium-permeable channel triggering plant immune signaling. Cell 184 3528–3541.e12
- 6j5t 2019 Reconstitution and structure of a plant NLR resistosome conferring immunity. Science 364 eaav5870
- 6j5v, 6j5w 2019 Ligand-triggered allosteric ADP release primes a plant NLR complex. Science 364 eaav5868
- AF-Q4UWF4-F1 2022 AlphaFold Protein Structure Database: massively expanding the structural coverage of protein-sequence space with high-accuracy models. Nucleic Acids Research 50 D439–D444
 生物大分子学习的教育门户
生物大分子学习的教育门户 









