287: ZAR1レジストソーム(ZAR1 Resistosome)
ヒトを含むその他すべての生物と同じように、植物も病原菌に感染し病気にかかることがある。これは農業や食糧生産にとって深刻な問題である。なぜなら、病気は作物の全滅させる可能性があるからである。幸いなことに、植物はそのような外部からの脅威に対抗して身を守る独自の免疫防御をすでに備えているが、適応免疫と自然免疫(adaptive and innate immunities)の両方を持つヒトとは異なり、植物は自然免疫しか持たない。自然免疫システムは、細胞表面や細胞内部にある受容体に依存しており、それぞれ病原体に関連する特定の分子パターンとエフェクター分子を検出する。検出されると、最終的に病原体を撃退し生存を維持するための免疫応答につながる一連のイベントが引き起こされる。ここでは、モデル生物として研究者に広く使われている一般的な雑草植物であるシロイヌナズナ(Arabidopsis thaliana)において、レジストソーム(resistosome)として知られる巨大な分子機械の形成に至るこれらの経路の一つを探っていくことにする。
病原菌を釣る
他の免疫反応と同様に、レジストソーム形成経路も感染そのものから始まる。例えば、葉に「黒腐れ」(black rot)を引き起こす主な原因となっている病原性細菌「キサントモナス・カンペストリス」(Xanthomonas campestris)は、ウリジリルトランスフェラーゼ(uridylyltransferase)AvrACを植物細胞に感染させることにより攻撃を開始する。この酵素は、その名の通り「ウリジリル」ヌクレオチドを標的タンパク質や核酸鎖に「転移」させる酵素群で、一般的には利用可能なヒドロキシ基を持つスレオニンやセリンなどのアミノ酸に結合させる。植物細胞内に入ると、AvrACは制御キナーゼ酵素にウリジル基を付加することで攻撃を開始し、それによって免疫信号伝達経路が阻害された結果、病原性が高まる。しかし巧妙なことに、植物細胞はPBL2と呼ばれるおとりタンパク質をエサにして反撃する。PBL2がウリジル化されると、免疫反応を停止させる代わりに、レジストソームを構築する過程を始動する。後述する「構造をみる」の節では対話的操作のできる図でそれを示している。
抵抗のピース
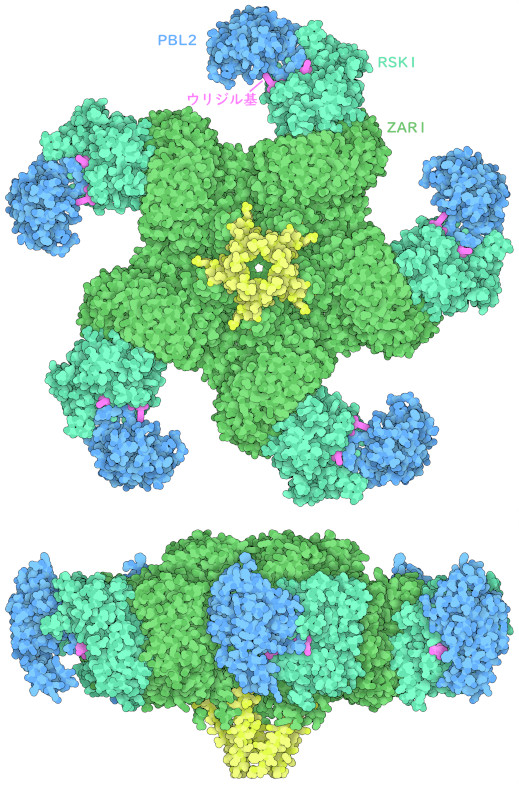
レジストソームを上から見た図(ここに示すのはPDBエントリー6j5tの構造)を観察すると、でき上がった集合体はリング状の構造をとり、3種類のタンパク質がそれぞれ5つずつ集まってできていることがわかる。リングの外縁にはウリジル化したPBL2サブユニット(濃い青色で示す部分)があり、各サブユニットの複合体中心側にはRSK1(擬似キナーゼ、青緑色で示す部分)と呼ばれる別のサブユニットが結合している。リングの内側にはZAR1(緑色で示す部分)と呼ばれる免疫受容体サブユニットがあり、5つの複合体を一つにつなげるための連結点になっている。注目すべきは、横から見た図に描かれているように、レジストソームの真ん中において、ZAR1サブユニット末端部(黄色)が、集合体の片側に漏斗状の突起を形成していることである。この漏斗形の部分は、ZAR1レジストソームの重要な保護機能を実行することが分かっている。これを細胞膜に挿入して孔をつくり、「過敏反応」(hypersensitive response)と呼ばれる局所的なプログラム細胞死を引き起こすことによって感染細胞を犠牲にし植物体全体を守るのである。漏斗の外側が比較的疎水性で、疎水性の細胞膜内部と相互作用しそうであることに加え、漏斗の内部が中空で親水性であり、細胞質環境に適していそうであることからこのような挿入機構が想定される。
ウリジル化調節
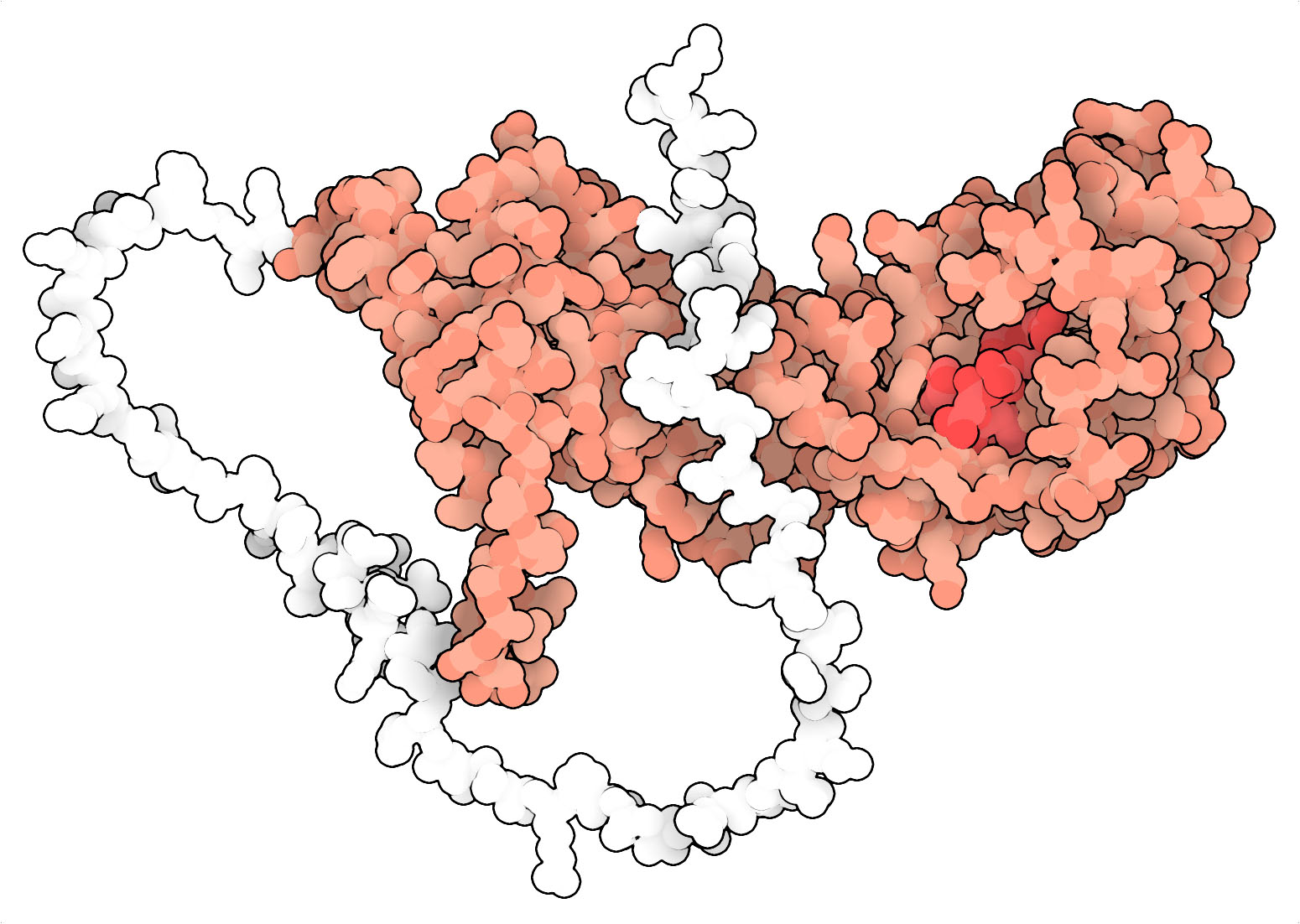
AF-Q4UWF4-F1から計算されたAvrACの構造モデルは、キナーゼ結合ドメイン(赤)と長く無秩序な尾部(白)の2つの基本部分から構成されている。キナーゼ結合領域は、AvrACとPBL2(デコイタンパク質)との結合を促進するだけでなく、AvrACのウリジル化活性を担う。この活性は、ヌクレオチドの結合と転移を仲介することができる「Fic」ドメイン(濃い赤で強調表示)と呼ばれる小さなモチーフによって付与される。重要なことは、AvrACがレジストソーム形成経路のカスケードの引き金を引くためには、PBL2上の2つの部位(1つはセリン、もう1つはスレオニン)をウリジル化しなければならないことである。
構造をみる
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
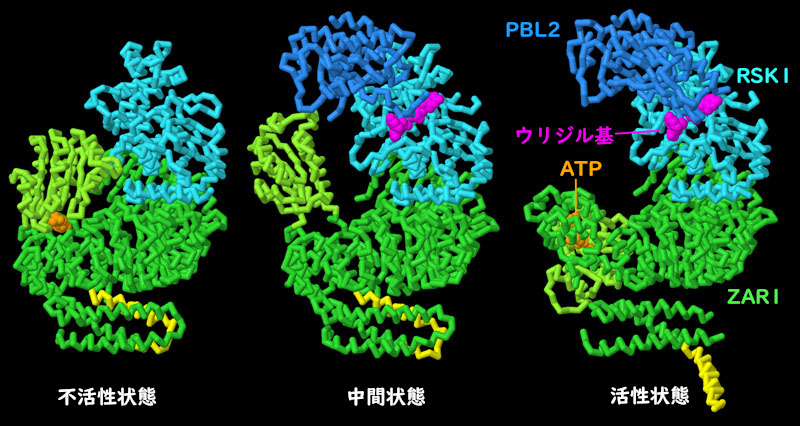
3つの構造から、活性型のレジストソームを構築する過程がどのような段階を経て進むのかが明らかになった。まず、ADP(オレンジ色)が結合した免疫受容体ZAR1(緑色)と疑似キナーゼRSK1(青緑色)とで構成された複合体は、左図に示すように、植物細胞内では不活性な状態で存在している(PDB ID 6j5w)。PBL2(濃い青色)が二重にウリジル化されると、RSK1と結合して中央に示しているより大きな中間複合体を形成し(PDB ID 6j5v)、開いてADPを放出できるようになる。この開いた中間状態は、2つのウリジル基(赤紫色)とRSK1のループ領域との安定化相互作用によって誘導され、最終的にATP/ADP結合ドメイン(黄緑色)を押し退け、ZAR1構造ドメイン(緑色)の構造変化を引き起こす。最後に、ZAR1サブユニットはATP(オレンジ色)と結合し、短いαらせんの尾部(黄色)の放出などの付加的なアロステリック構造変化を引き起こし、最終的に右図に示す活性型複合体(PDB ID 6j5t)となる。これらの活性化複合体が5つ集まってレジストソーム集合体が完成したとき、黄色の尾部は膜と相互作用する漏斗形の部分になる。
理解を深めるためのトピックス
参考文献
- 2012 A Xanthomonas uridine 5′-monophosphate transferase inhibits plant immune kinases. Nature 485 114–118
- 2015 The decoy substrate of a pathogen effector and a pseudokinase specify pathogen-induced modified-self recognition and immunity in plants. Cell Host & Microbe 18 285–295
- 2019 The plant “resistosome”: structural insights into immune signaling. Cell Host & Microbe 26 193–201
- 2021 The ZAR1 resistosome is a calcium-permeable channel triggering plant immune signaling. Cell 184 3528–3541.e12
- 6j5t 2019 Reconstitution and structure of a plant NLR resistosome conferring immunity. Science 364 eaav5870
- 6j5v, 6j5w 2019 Ligand-triggered allosteric ADP release primes a plant NLR complex. Science 364 eaav5868
- AF-Q4UWF4-F1 2022 AlphaFold Protein Structure Database: massively expanding the structural coverage of protein-sequence space with high-accuracy models. Nucleic Acids Research 50 D439–D444
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









