277: 吃塑料的酶(Plastic-eating Enzymes)
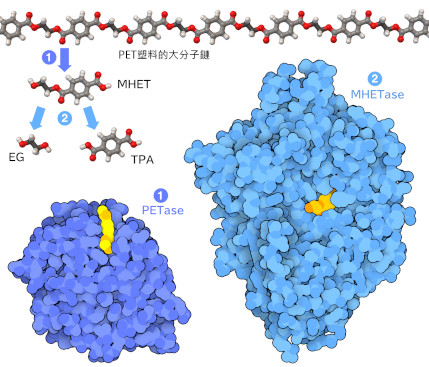
塑料(plastic)已經改變了我們生活的世界,成為製造消費產品的一種廉價和通用的材料。然而,使用塑料有著嚴重的弊端。大多數常用的塑料是高度穩定的化學聚合物,當被丟棄後,會在環境中保留幾十年甚至幾百年。塑料的廣泛使用對全球環境和人類健康構成了日益嚴重的威脅。研究人員現在正在尋找回收和分解塑料的自然方法來解決這個問題。令人驚訝的是,一些細菌比我們先一步,已經進化出可以攻擊某些種類塑料的酶。
吃塑料的酶
這裡顯示的兩種酶(PDB條目5xh3和6qga)是在回收設施的塑料瓶上生長的細菌中發現的。這兩種酶可以降解聚對苯二甲酸乙二醇酯(polyethylene terephthalate,PET)。這種塑料由兩種成分組成,通過酯鍵(ester linkage)連接形成長聚合物鏈(polymer chain)。 PET酶將聚合物鏈裂成可處理的碎片,MHET酶(monohydroxyethyl terephthalate hydrolase,單羥乙基對苯二甲酸酯水解酶)通過從片段中分離單個成分來完成任務。細菌中的另一種酶將這些成分作為生長的營養物質。
回收塑料
在工業規模上回收塑料有幾個挑戰。將塑料聚合物中的成分-通常是酯和酰胺-結合在一起的鍵具有穩定的化學性質。幸運的是,許多酶都善於裂解這種類型的鍵。這是因為這種類型的鍵在生物聚合物中也很常見。然而,塑料中許多聚合物纖維的緻密結構使酶難以進入這些化學鍵。解決這個問題的一個辦法是加熱塑料以軟化其結構。 出於這個原因,人們正在研究對這種天然酶進行人工修飾,使其更加穩定並能在更高溫度下工作。
分解尼龍
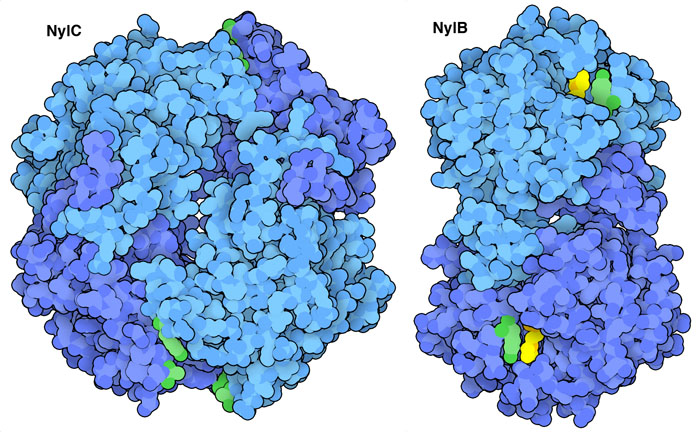
吃塑料的酶的發現引發了對新的食塑料生物的尋找。例如,在篩選(檢查和選擇那些符合標準的)許多細菌後,發現了一種能降解塑料尼龍(nylon)的酶。該酶的結構如左圖所示。尼龍是由酰胺鍵(amide bond)組成的,與連接構成蛋白質的氨基酸的鍵相同。與PET酶一樣,尼龍的降解分兩步進行,NylC將尼龍聚合物分解成更小的碎片,NylB將碎片完全降解為其組成成分。 (注:這些酶可分解尼龍6製造過程中的副產品尼龍低聚物,並有助於減少對環境的影響,固化的尼龍樹脂很難被酶直接接觸和分解)。這裡顯示的酶來自PDB ID 5y0m 和2zm0,一些氨基酸被修改以增加活性和熱穩定性。
探索結構
要切換到有互動控制的頁面,請點擊圖表下方的按鈕。如果加載沒有開始,請嘗試點擊圖表。
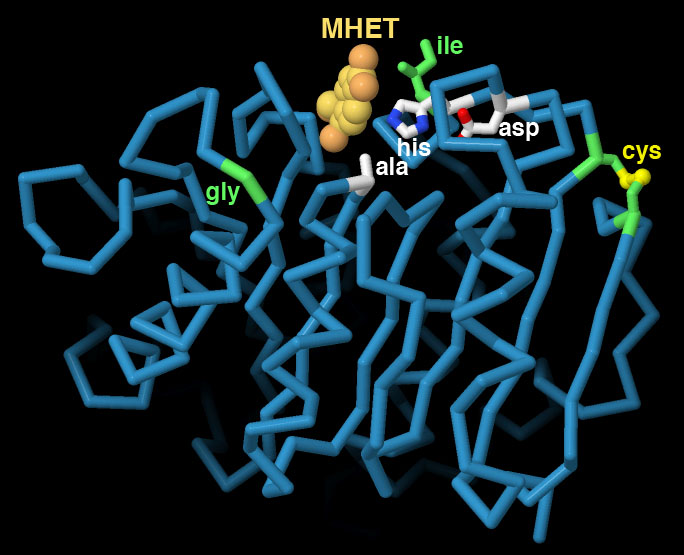
PDB ID 7vve 包含一種PET降解酶,具有增強的塑料消化能力和更高的熱穩定性。該結構包括結合在活性位點的MHET,並揭示了幾種突變如何增強酶的活性。兩個相鄰的位點被改變為半胱氨酸,它們一起形成一個二硫鍵,增加了酶的熱穩定性。另外兩個變化減小了活性位點側翼氨基酸的大小,可能使其更容易被塑料聚合物鏈接近。該酶使用經典的絲氨酸-組氨酸-天冬氨酸催化三聯體,就像在絲氨酸蛋白酶中看到的那樣。請注意,在這個結構中,絲氨酸已被改為丙氨酸,以確定與活性部位結合的塑料片的結構。為了更詳細地探索這個結構,並與野生型角蛋白酶(cutinase,PDB ID 4eb0)進行比較,請點擊圖片下面的按鈕,請單擊圖像以獲取交互式JSmol。
進一步的討論議題
參考文獻
- 2022 Microbial enzymes will offer limited solutions to the global plastic pollution crisis. Microbial Biotechnology DOI:10.1111/1751-7915.14135
- 7vve 2022 Substrate-Binding Mode of a Thermophilic PET Hydrolase and Engineering the Enzyme to Enhance the Hydrolytic Efficacy. ACS Catal 12 3033-3040
- 6qga 2019 Structure of the plastic-degrading Ideonella sakaiensis MHETase bound to a substrate. Nat Commun 10 1717
- 5y0m 2018 Structural basis of the correct subunit assembly, aggregation, and intracellular degradation of nylon hydrolase. Sci Rep 8 9725-9725
- 5xh3 2017 Structural insight into catalytic mechanism of PET hydrolase. Nat Commun 8 2106-2106
- 2zm0 2009 Molecular design of a nylon-6 byproduct-degrading enzyme from a carboxylesterase with a beta-lactamase fold. FEBS J 276 2547-2556
 生物大分子學習的教育門戶
生物大分子學習的教育門戶 









