277플라스틱을 먹는 효소 (Plastic-eating Enzymes)
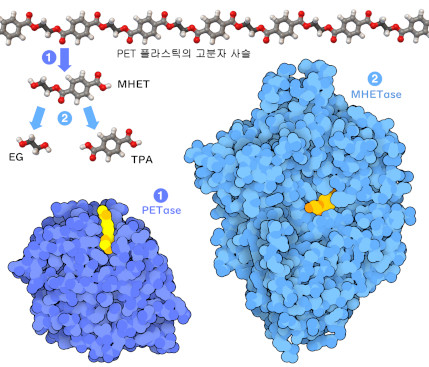
플라스틱(plastic)은 우리가 사는 세상을 크게 바꾸고 소비자용 제품을 만들기 위한 저렴하고 범용성 있는 방법이 되었다. 그러나 플라스틱에는 심각한 결점이 있다. 일반적으로 사용되는 플라스틱의 대부분은 매우 안정적인 화학 고분자이며, 폐기되면 수십 년 또는 수 세기에 걸쳐 환경 속에 잔류한다. 플라스틱 보급은 지구 환경과 사람들의 건강에 점점 더 심각한 위협을 주고 있다. 연구자들은 현재 이 문제에 맞서기 위해 플라스틱을 재활용하거나 분해하는 자연스러운 방법을 찾고 있지만 놀랍게도 일부 세균은 우리보다 먼저 모종의 플라스틱을 공격하는 효소를 이미 진화시키고 있다.
플라스틱을 먹는 것들
여기에 나타낸 2개의 효소(PDB 엔트리 5xh3, 6qga)는 재활용 시설의 페트병에서 증식하는 세균에서 발견됐다. 이 2개의 효소는 폴리에틸렌 테레프탈레이트(polyethylene terephthalate, PET)를 분해한다. 이 플라스틱은 2종류의 성분이 에스테르 결합(ester linkage)으로 이어져 긴 고분자 사슬(폴리머 사슬, polymer chain)로 된 것이다. PETase 효소가 고분자 사슬을 다루기 쉬운 크기로 절단하고 MHETase 효소(monohydroxyethyl terephthalate hydrolase)가 그 단편에서 개개의 구성 성분을 잘라 작업을 완료한다. 그리고 세균이 가진 또 다른 효소가 이 성분을 영양분으로 이용해 생육에 도움을 준다.
플라스틱을 재활용하다
플라스틱을 산업 규모로 재활용하려면 몇 가지 과제가 있다. 플라스틱 폴리머의 구성 요소인 에스테르나 아미드(amide) 등의 결합은 화학적으로 안정적이다. 다행히 효소의 대부분은 이런 종류의 결합을 절단하는 것을 특기로 하고 있다. 왜냐하면 이런 종류의 결합은 생체 고분자에서도 공통적으로 사용되는 결합이기 때문이다. 그러나 플라스틱은 많은 고분자 섬유가 밀집된 구조를 하고 있기 때문에 효소가 이 화학 결합에 접근하기 어렵다. 이 문제에 대한 한 가지 해결책은 플라스틱을 가열하여 구조를 부드럽게 하는 것이다. 이 때문에 이 천연 효소를 보다 안정시키고 고온에서도 작용하도록 인공적으로 개량하는 검토가 이루어지고 있다.
나일론을 먹다
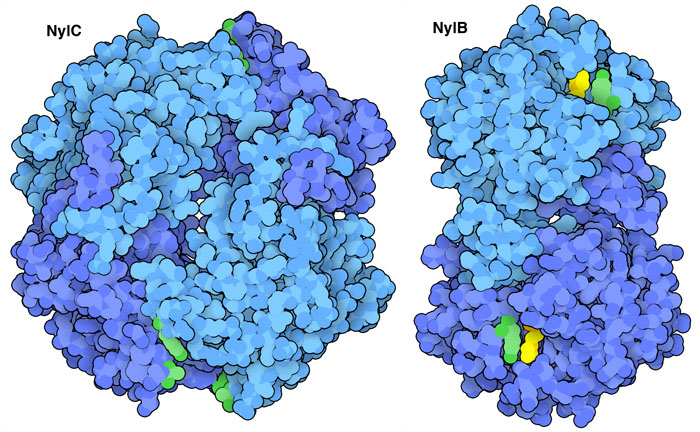
플라스틱을 먹는 효소 발견을 계기로 새로운 플라스틱을 먹는 생물 탐색이 시작되었다. 예를 들어 많은 세균을 선별한(조사하여 조건에 맞는 것을 골라낸) 결과, 플라스틱 일종인 나일론(nylon)을 분해하는 효소가 발견되었다. 왼쪽 그림에 나타낸 것은 그 구조이다. 나일론은 단백질을 구성하는 아미노산을 연결하는 것과 같은 아미드 결합(amide bond)으로 이루어져 있다. 나일론의 분해는 PET의 효소와 마찬가지로 NylC가 나일론 폴리머를 더 작은 단편으로 분해하고 NylB가 그 단편을 구성 성분으로 완전히 분해하는 2단계 공정을 거쳐 이루어진다.(역주: 이 효소들은 나일론 6 제조 과정에서 부산물로 생기는 나일론 올리고머를 분해하여 환경 부하를 억제하는 데 도움이 된다. 굳어진 나일론 수지는 효소가 접근하기 어려워 직접 분해하기 어렵다.) 여기에 나타낸 효소는 PDB 엔트리 5y0m과 2zm0의 구조로, 활성과 열 안정성을 높이기 위해 여러 아미노산이 개변되었다.
구조 보기
인터랙티브 조작이 가능한 페이지로 전환하려면 그림 아래의 버튼을 클릭하십시오. 읽기가 시작되지 않을 때는 그림을 클릭하십시오.
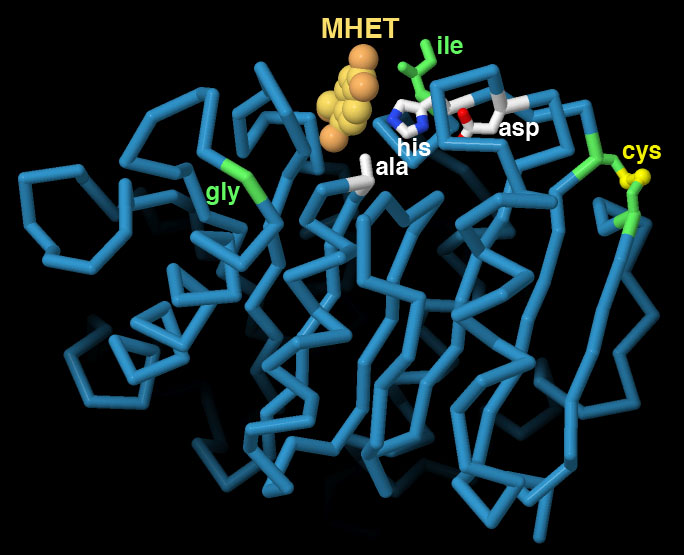
PDB 엔트리 7vve에는 플라스틱 분해 능력과 열 안정성을 향상시킨 PET 분해 효소가 포함되어 있다. 이 구조에는 활성 부위에 결합한 MHET가 포함되어 있으며, 몇 가지 변이에 의해 효소의 활성이 증강되는 것으로 밝혀졌다. 인접한 2개의 부위가 시스테인으로 변화하고 이들이 함께 어울려 디설피드 결합을 형성함으로써 효소의 열에 대한 안정성이 향상되었다. 다른 두 변이는 활성 부위를 사이에 둔 아미노산의 크기를 줄이는 것으로, 아마도 플라스틱 고분자 사슬이 더 접근하기 쉬워진 것으로 생각된다. 이 효소는 세린 프로테아제에서 전형적으로 볼 수 있는 세린-히스티딘-아스파라긴산의 촉매 삼잔기를 사용하고 있다. 또한 이 구조에서는 플라스틱 조각이 활성 부위에 결합한 구조를 결정하기 위해 세린은 알라닌으로 변경되었다. 이 구조를 보다 자세히 살펴보고 야생형의 큐티나아제 효소(cutinase, PDB 엔트리 4eb0)와 비교하기 위해 이미지 아래의 버튼을 클릭하여 인터랙티브 조작이 가능한 이미지로 전환해 보기 바란다.
이해를 높이기 위한 토픽
- 인공적으로 만들어진 플라스틱을 분해하는 효소의 구조가 PDB 아카이브에 다수 공개되어 있습니다. 이를 찾으려면 먼저 몇 가지 효소의 구조 페이지에 접속하여 PDBj의 경우 ‘상동단백질’ 탭을, RCSB PDB의 경우 ‘Find similar proteins by: Sequence’(각 구조 개요 페이지의 ‘Macromolecules’ 섹션에 있음)를 참조해 보십시오. 덧붙여 변화는 보통 미미하기 때문에 80%나 90%의 배열이 일치하는 것을 추출하면 유용한 리스트를 얻을 수 있을 것입니다.
- PDBj의 ASH 또는 RCSB PDB의 ‘Pairwise Structure Alignment’를 사용하면 인공적인 개변을 가한 구조와 야생형의 구조를 간단히 비교할 수 있습니다.
참고문헌
- 2022 Microbial enzymes will offer limited solutions to the global plastic pollution crisis. Microbial Biotechnology DOI:10.1111/1751-7915.14135
- 7vve 2022 Substrate-Binding Mode of a Thermophilic PET Hydrolase and Engineering the Enzyme to Enhance the Hydrolytic Efficacy. ACS Catal 12 3033-3040
- 6qga 2019 Structure of the plastic-degrading Ideonella sakaiensis MHETase bound to a substrate. Nat Commun 10 1717
- 5y0m 2018 Structural basis of the correct subunit assembly, aggregation, and intracellular degradation of nylon hydrolase. Sci Rep 8 9725-9725
- 5xh3 2017 Structural insight into catalytic mechanism of PET hydrolase. Nat Commun 8 2106-2106
- 2zm0 2009 Molecular design of a nylon-6 byproduct-degrading enzyme from a carboxylesterase with a beta-lactamase fold. FEBS J 276 2547-2556











