277: 吃塑料的酶(Plastic-eating Enzymes)
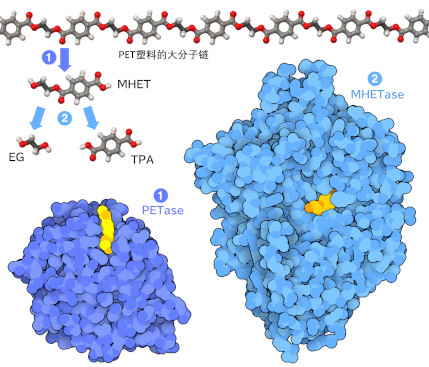
塑料(plastic)已经改变了我们生活的世界,成为制造消费产品的一种廉价和通用的材料。然而,使用塑料有着严重的弊端。大多数常用的塑料是高度稳定的化学聚合物,当被丢弃后,会在环境中保留几十年甚至几百年。塑料的广泛使用对全球环境和人类健康构成了日益严重的威胁。研究人员现在正在寻找回收和分解塑料的自然方法来解决这个问题。令人惊讶的是,一些细菌比我们先一步,已经进化出可以攻击某些种类塑料的酶。
吃塑料的酶
这里显示的两种酶(PDB条目5xh3和6qga)是在回收设施的塑料瓶上生长的细菌中发现的。这两种酶可以降解聚对苯二甲酸乙二醇酯(polyethylene terephthalate,PET)。这种塑料由两种成分组成,通过酯键(ester linkage)连接形成长聚合物链(polymer chain)。PET酶将聚合物链裂成可处理的碎片,MHET酶(monohydroxyethyl terephthalate hydrolase,单羟乙基对苯二甲酸酯水解酶)通过从片段中分离单个成分来完成任务。细菌中的另一种酶将这些成分作为生长的营养物质。
回收塑料
在工业规模上回收塑料有几个挑战。将塑料聚合物中的成分-通常是酯和酰胺-结合在一起的键具有稳定的化学性质。幸运的是,许多酶都善于裂解这种类型的键。这是因为这种类型的键在生物聚合物中也很常见。然而,塑料中许多聚合物纤维的致密结构使酶难以进入这些化学键。解决这个问题的一个办法是加热塑料以软化其结构。 出于这个原因,人们正在研究对这种天然酶进行人工修饰,使其更加稳定并能在更高温度下工作。
分解尼龙
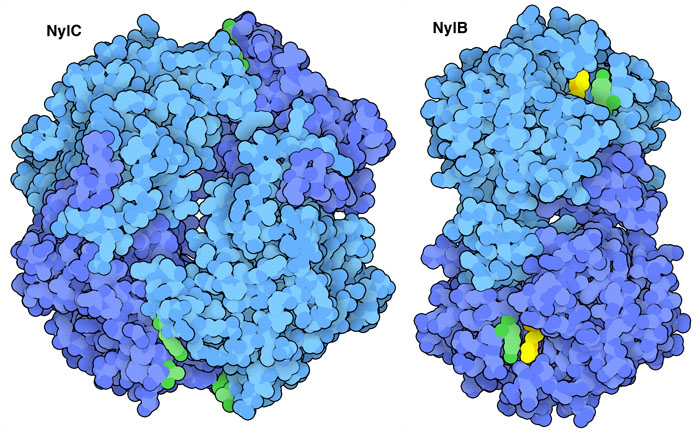
吃塑料的酶的发现引发了对新的食塑料生物的寻找。例如,在筛选(检查和选择那些符合标准的)许多细菌后,发现了一种能降解塑料尼龙(nylon)的酶。该酶的结构如左图所示。尼龙是由酰胺键(amide bond)组成的,与连接构成蛋白质的氨基酸的键相同。与PET酶一样,尼龙的降解分两步进行,NylC将尼龙聚合物分解成更小的碎片,NylB将碎片完全降解为其组成成分。(注:这些酶可分解尼龙6制造过程中的副产品尼龙低聚物,并有助于减少对环境的影响,固化的尼龙树脂很难被酶直接接触和分解)。这里显示的酶来自PDB ID 5y0m 和 2zm0,一些氨基酸被修改以增加活性和热稳定性。
探索结构
要切换到有互动控制的页面,请点击图表下方的按钮。如果加载没有开始,请尝试点击图表。
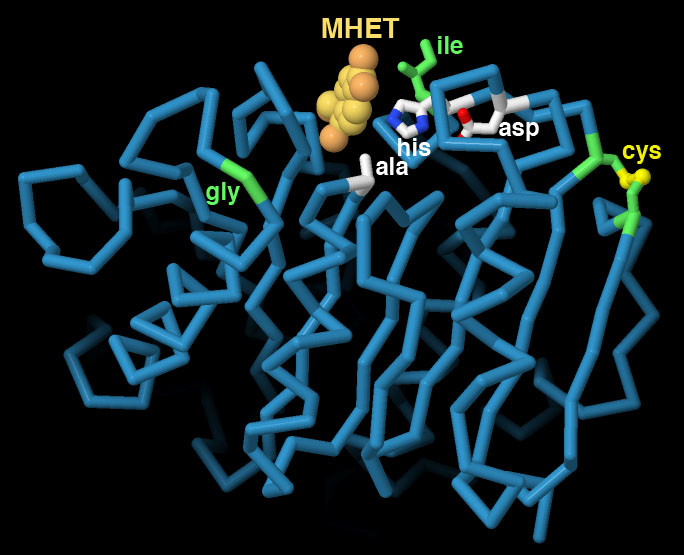
PDB ID 7vve 包含一种PET降解酶,具有增强的塑料消化能力和更高的热稳定性。该结构包括结合在活性位点的MHET,并揭示了几种突变如何增强酶的活性。两个相邻的位点被改变为半胱氨酸,它们一起形成一个二硫键,增加了酶的热稳定性。另外两个变化减小了活性位点侧翼氨基酸的大小,可能使其更容易被塑料聚合物链接近。该酶使用经典的丝氨酸-组氨酸-天冬氨酸催化三联体,就像在丝氨酸蛋白酶中看到的那样。请注意,在这个结构中,丝氨酸已被改为丙氨酸,以确定与活性部位结合的塑料片的结构。为了更详细地探索这个结构,并与野生型角蛋白酶(cutinase,PDB ID 4eb0)进行比较,请点击图片下面的按钮,请单击图像以获取交互式JSmol。
进一步的讨论议题
参考文献
- 2022 Microbial enzymes will offer limited solutions to the global plastic pollution crisis. Microbial Biotechnology DOI:10.1111/1751-7915.14135
- 7vve 2022 Substrate-Binding Mode of a Thermophilic PET Hydrolase and Engineering the Enzyme to Enhance the Hydrolytic Efficacy. ACS Catal 12 3033-3040
- 6qga 2019 Structure of the plastic-degrading Ideonella sakaiensis MHETase bound to a substrate. Nat Commun 10 1717
- 5y0m 2018 Structural basis of the correct subunit assembly, aggregation, and intracellular degradation of nylon hydrolase. Sci Rep 8 9725-9725
- 5xh3 2017 Structural insight into catalytic mechanism of PET hydrolase. Nat Commun 8 2106-2106
- 2zm0 2009 Molecular design of a nylon-6 byproduct-degrading enzyme from a carboxylesterase with a beta-lactamase fold. FEBS J 276 2547-2556
 生物大分子学习的教育门户
生物大分子学习的教育门户 









