277: プラスチックを食べる酵素(Plastic-eating Enzymes)
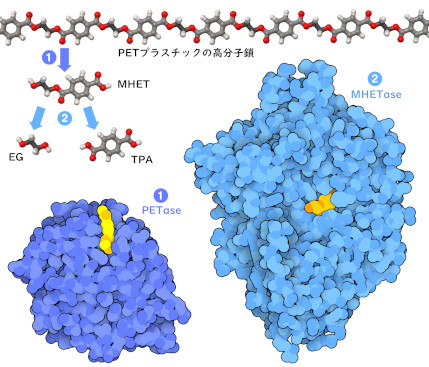
プラスチック(plastic)は、私たちの住む世界を大きく変え、消費者向け製品を作るための安価で汎用性のある方法になっている。しかし、プラスチックには深刻な欠点がある。一般的に使用されているプラスチックのほとんどは、非常に安定した化学高分子であり、廃棄されると、何十年、あるいは何世紀にもわたって環境中に残留する。プラスチックの普及は、地球環境と人々の健康にますます深刻な脅威を与えている。研究者たちは現在、この問題に立ち向かうため、プラスチックのリサイクルや分解を行う自然な方法を探しているが、驚くべきことに、一部の細菌は私たちよりも先に、ある種のプラスチックを攻撃する酵素をすでに進化させている。
プラスチックを食べる者たち
ここに示す2つの酵素(PDBエントリー5xh3、6qga)は、リサイクル施設のペットボトルで増殖する細菌から発見された。この2つの酵素は、ポリエチレンテレフタレート(polyethylene terephthalate、PET)を分解する。このプラスチックは、2種類の成分がエステル結合(ester linkage)でつながり長い高分子鎖(ポリマー鎖、polymer chain)になったものである。PETase酵素が高分子鎖を扱いやすい大きさに切断し、MHETase酵素(monohydroxyethyl terephthalate hydrolase)がその断片から個々の構成成分を切り分けて、作業を完了させる。そして、細菌が持つ別の酵素が、これらの成分を栄養分として利用し、生育に役立てる。
プラスチックをリサイクルする
プラスチックを産業規模でリサイクルするには、いくつかの課題がある。プラスチックポリマーの構成要素となっているエステルやアミド(amide)などの結合は、化学的に安定である。幸い、酵素の多くはこの種の結合を切断することを得意としている。なぜなら、この種の結合は生体高分子でも共通して使われる結合だからである。しかし、プラスチックは多くの高分子繊維が密集した構造をしているため、酵素がこの化学結合にアクセスすることが難しい。この問題に対する一つの解決策は、プラスチックを熱して構造を柔らかくすることである。 このため、この天然酵素をより安定させ、高温でも作用するように人工的に改良する検討が行われている。
ナイロンを食べる
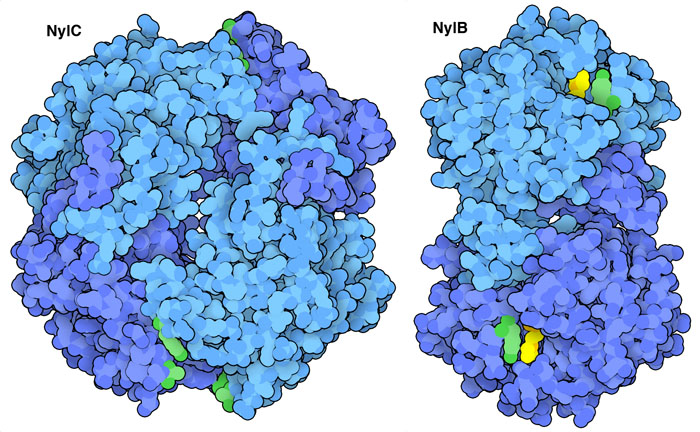
プラスチックを食べる酵素の発見をきっかけに、新たなプラスチックを食べる生物の探索が始まった。例えば、多くの細菌をスクリーニングした(調べて条件に合うものを選び出した)結果、プラスチック一種ナイロン(nylon)を分解する酵素が発見された。左図に示すのはその構造である。ナイロンは、タンパク質を構成するアミノ酸をつなぐのと同じアミド結合(amide bond)でできている。ナイロンの分解は、PETの酵素と同様に、NylCがナイロンポリマーをより小さな断片へと分解し、NylBがその断片を構成成分へと完全に分解するという2段階の工程を経て行われる。(訳注:これらの酵素はナイロン6製造過程で副産物としてできるナイロンオリゴマーを分解し、環境への負荷を抑えるのに役立つ。固められたナイロン樹脂は酵素がアクセスしづらく直接分解するのは難しい。)ここに示した酵素はPDBエントリー5y0mと2zm0の構造で、活性と熱安定性を高めるために、いくつかのアミノ酸が改変されている。
構造をみる
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
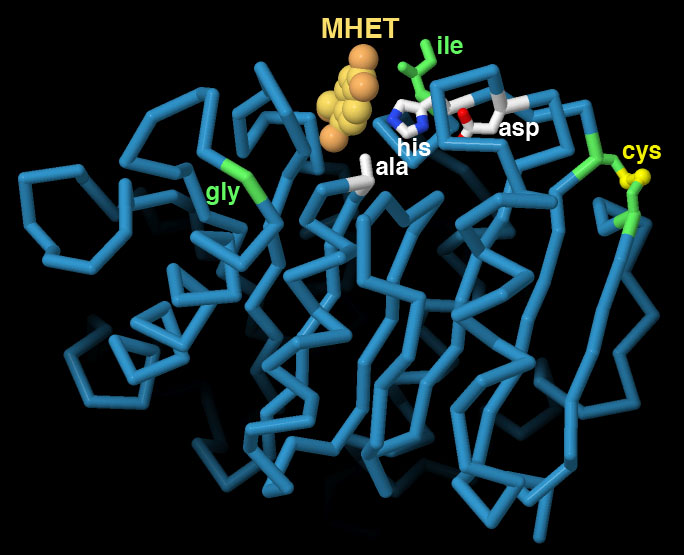
PDBエントリー7vveには、プラスチック分解能力と熱安定性を向上させたPET分解酵素が含まれている。この構造には、活性部位に結合したMHETが含まれており、いくつかの変異によって酵素の活性が増強されることが明らかになっている。隣接する2つの部位がシステインに変化し、それらが一緒になってジスルフィド結合を形成することで、酵素の熱に対する安定性が向上している。他の2つの変異は、活性部位を挟むアミノ酸のサイズを小さくするもので、おそらくプラスチック高分子鎖がよりアクセスしやすくなったものと考えられる。この酵素は、セリンプロテアーゼで典型的に見られるような、セリン-ヒスチジン-アスパラギン酸の触媒三残基を使っている。なおこの構造では、プラスチック片が活性部位に結合した構造を決定するために、セリンはアラニンに変更されている。この構造をより詳しく調べ、野生型のクチナーゼ酵素(cutinase、PDBエントリー4eb0)と比較するため、画像の下のボタンをクリックして対話的操作のできる画像に切り替えてみて欲しい。
理解を深めるためのトピックス
- 人工的に作られたプラスチックを分解する酵素の構造が、PDBアーカイブで多数公開されています。それらを見つけるには、まずいずれかの酵素の構造ページにアクセスし、PDBjの場合は「相同蛋白質」タブを、RCSB PDBの場合は「Find similar proteins by: Sequence」(各構造概要ページの「Macromolecules」セクションにあります)を参照してみてください。なお、変化は通常わずかなので、80%か90%の配列が一致しているものを抽出すれば、役に立つリストが得られるでしょう。
- PDBjのASHまたはRCSB PDBの「Pairwise Structure Alignment」を使うと、人工的な改変を加えた構造と野生型の構造とを簡単に比較することができます。
参考文献
- 2022 Microbial enzymes will offer limited solutions to the global plastic pollution crisis. Microbial Biotechnology DOI:10.1111/1751-7915.14135
- 7vve 2022 Substrate-Binding Mode of a Thermophilic PET Hydrolase and Engineering the Enzyme to Enhance the Hydrolytic Efficacy. ACS Catal 12 3033-3040
- 6qga 2019 Structure of the plastic-degrading Ideonella sakaiensis MHETase bound to a substrate. Nat Commun 10 1717
- 5y0m 2018 Structural basis of the correct subunit assembly, aggregation, and intracellular degradation of nylon hydrolase. Sci Rep 8 9725-9725
- 5xh3 2017 Structural insight into catalytic mechanism of PET hydrolase. Nat Commun 8 2106-2106
- 2zm0 2009 Molecular design of a nylon-6 byproduct-degrading enzyme from a carboxylesterase with a beta-lactamase fold. FEBS J 276 2547-2556
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









