261: DNA 배열 나노포어 (DNA-Sequencing Nanopores)
이 기사는 2021년 1월에 정량생물의학 럿거스연구소(Rutgers Institute for Quantitative Biomedicine) 주최로 개최된 1주일간의 집중 합숙 ‘생물의학에서의 과학 커뮤니케이션’(Science Communication in Biology and Medicine)의 일환으로 제니퍼 장(Jennifer Jiang), 캐서린 H. 박(Katherine H. Park), 키란마이 베뮤리(Kiranmayi Vemuri)에 의해 집필되고 그려진 것입니다.
DNA 시퀀싱(DNA sequencing)은 생물학 연구에 혁명을 가져오고 생명의 과정을 코딩한 정보를 밝혀냈다. 인간 게놈 프로젝트(Human Genome Project)에서는 기존의 생어 사슬종료법(Sanger chain-termination method)이 사용되었고, 모두 13년의 세월과 약 30억 달러의 비용이 들었다. 유전체학에 대한 관심이 높아지면서 보다 빠르고 고성능이며 비용 대비 효과가 높은 배열 결정 기술을 요구하는 목소리도 점점 높아지고 있다. 나노포어 시퀀싱(nanopore sequencing)은 이러한 요구를 충족시키기 위해 급속히 발전하고 있는 툴이며, 디바이스 가격이 1,000달러를 밑도는 경우도 드물지 않다. 오늘날 이 기술은 남극부터 국제우주정거장까지 어디서나 사용할 수 있다!
포켓 크기 배열 판독기
나노포어 시퀀싱에서는 막에 매립된 작은 구멍을 DNA 사슬이 통과할 때 배열을 해독한다. 기존 DNA 배열결정법과 달리 DNA의 합성이나 증폭에 의존하지 않고 뉴클레오타이드를 직접 읽어내고 있다. 이 기술은 긴 DNA 단편 배열 해독에도 사용할 수 있어 생물의 전체 게놈을 빠르고 확실하게 단편화하여 읽을 수 있다. 하나의 디바이스(배열 판독 장치, 시퀀서)에는 수천 개의 나노포어(nm 나노미터=10억분의 1미터 정도의 크기를 가지는 구멍)가 있지만 그래도 심플하고 저렴하게 주머니에 넣을 수조차 있다. 현재 에러율 저감이나 1회당의 수율 향상 등의 문제는 최소한으로 억제되고 있다.
나노포어가 작용하는 모습
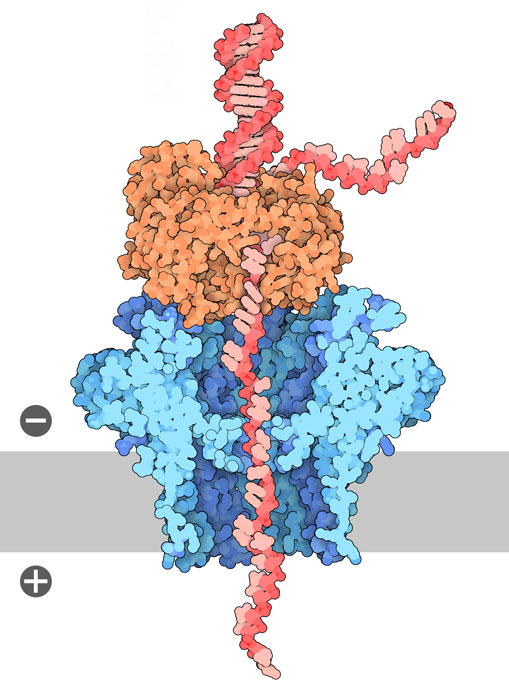
나노포어 시퀀서에는 염용액을 2개의 구획으로 구분하는 막이 있고, 그 막에 나노포어가 매립되어 있다. 막에 전압을 가하면 이온의 흐름이 발생하여 그것을 측정한다. DNA 사슬이 구멍을 지날 때 이온의 흐름이 부분적으로 방해를 받아 측정되는 전류값이 떨어진다. 4종류의 핵산 염기에 대응하는 이온 흐름의 변화량은 각기 다르기 때문에 식별할 수 있다. 상업적으로 입수 가능한 시퀀서는 현재 세균의 단백질 CsgG를 나노포어에 상당하는 것으로 이용한 파생품을 사용하고 있다. 여기에 나타낸 CsgG(PDB 엔트리 4uv3)는 직경 약 1nm로 개폐되지 않는 비선택적인 외막 단백질로, 단일가닥 DNA가 쉽게 통과할 수 있다. DNA 판독 성능을 향상시키기 위해 CsgG에는 개량이 진행되어 아래와 같이 나노포어의 형태가 바뀌었다. 그 결과, 수율과 배열 판독의 정확성이 향상되었다.
DNA의 구멍 통과를 제어하다
DNA 사슬이 나노포어를 지날 때 배열을 효율적으로 읽을 수 있는 속도를 유지하려면 DNA 사슬을 정확하게 제어할 필요가 있다. 유리한 DNA 사슬이 나노포어를 통과하는 속도가 너무 빠르면 각 염기를 명확하게 읽을 수 없게 된다. 연구자들은 중합효소(polymerase)나 헬리카제(helicase) 같은 DNA 결합성 효소를 나노포어 구조에 부가함으로써 속도를 조절할 수 있음을 발견했다. 여기에 나타낸 것은 박테리오파지 φ29의 DNA 중합효소(DNA polymerase, PDB 엔트리 1xhz)이다. 이것은 DNA를 감속시키는 스테핑 모터(stepper motor)로 작용한다. DNA는 φ29 DNA 중합효소와 결합하여 1염기의 분해능으로 CsgG의 구멍을 통해 이동한다.
천연 구멍
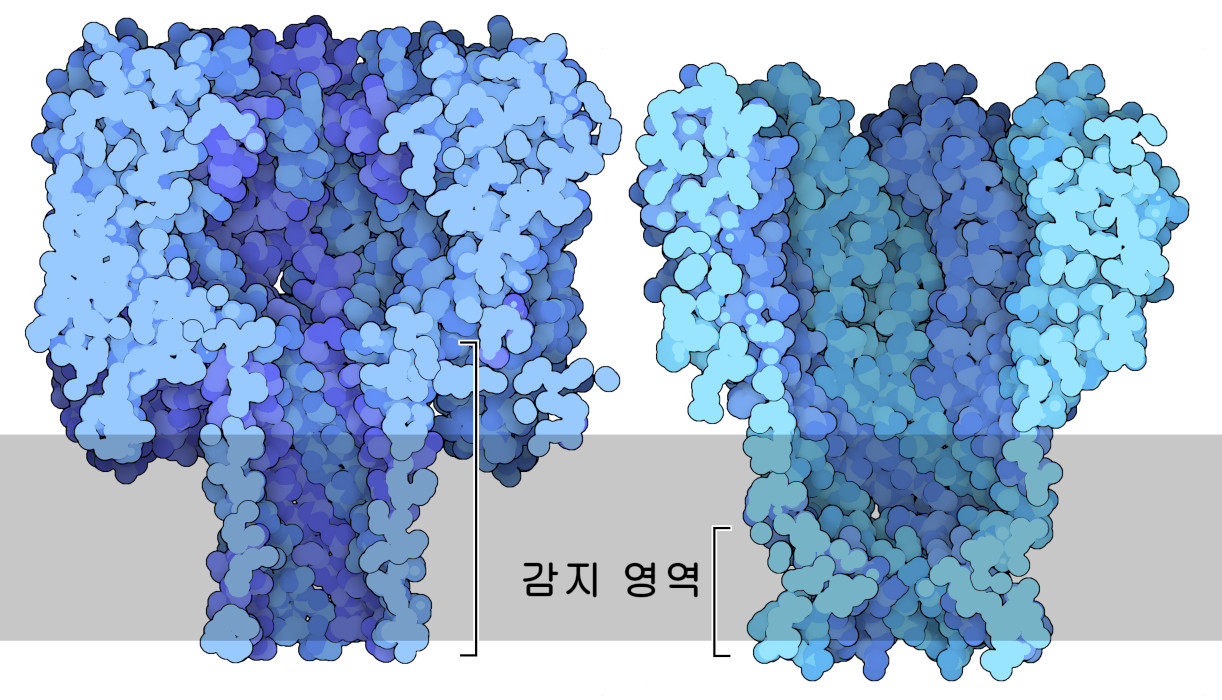
구멍을 만드는 천연의 막 단백질은 그 밖에도 많이 존재한다. α 용혈소(α-hemolysin, PDB 엔트리 7ahl)는 세균이 만드는 독소로, DNA 배열 결정에 이용된 최초의 생물에서 유래한 나노포어이기도 하다. 구멍은 1.5~2.5nm의 직경을 갖는 긴 채널로, 개개의 핵산 염기를 식별하는 데 최적이라고는 할 수 없다. 마이코박테리아에서 유래한 단백질 MspA(PDB 엔트리 1uun)는 이보다 짧은 감지 영역을 가진 천연 나노포어다. 이 특징은 이점이 된다. 왜냐하면 감지 영역이 짧은 나노포어에서는 기록되는 전류의 특성에 영향을 주는 뉴클레오타이드가 적어지고, 그 결과, 보다 정확하게 배열을 읽을 수 있기 때문이다. MspA는 직경 약 1.2nm로 보다 짧은 감지 영역을 가지기 때문에 α 용혈소에 비해 좋은 시퀀서라 할 수 있다.
구조 보기
인터랙티브 조작이 가능한 페이지로 전환하려면 그림 아래의 버튼을 클릭하십시오. 읽기가 시작되지 않을 때는 그림을 클릭하십시오.
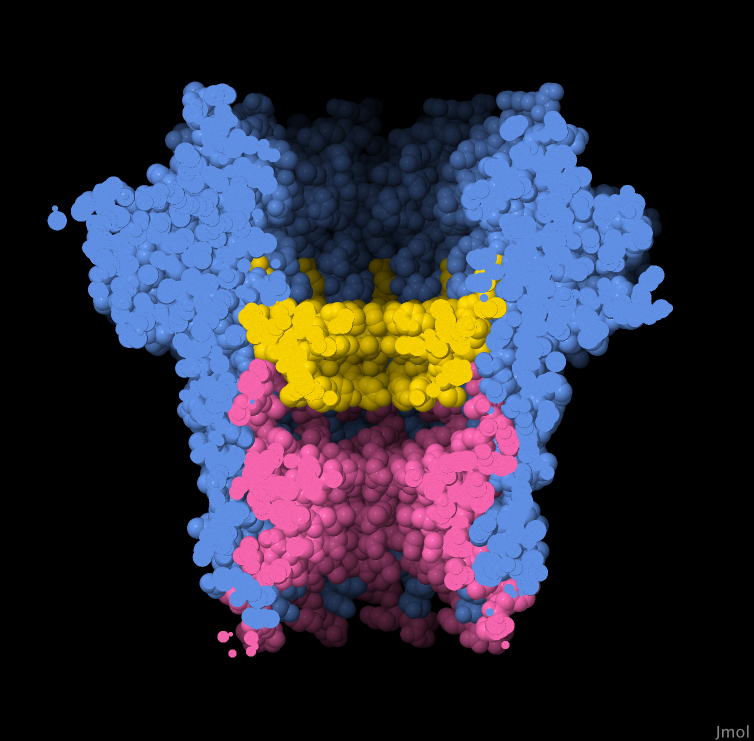
구멍의 안정성을 향상시켜 최적의 기능을 갖도록 개량한 나노포어가 만들어졌다. 예를 들어 이중협착 나노포어(dual constriction nanopor)에서는 CsgG에 보조 단백질을 부가해 DNA를 감지하는 2번째의 좁아진 부분을 도입했다. 여기에 나타낸 CsgG-CsgF 복합체(PDB 엔트리 6si7)에서는 CsgG 분자를 파란색, 구멍을 구성하고 있는 영역은 노란색, CsgF 분자는 분홍색으로 강조하고 있다. DNA가 지나는 경로 상에서 CsgG가 첫 번째로 좁아진 곳을 만들고 CsgF가 두 번째로 좁아진 곳을 만들고 있다. 2개의 좁아진 곳으로부터의 신호를 통합하면 염기의 판독 정밀도가 향상된다. 특히 같은 염기가 연속되는 곳이 여러 곳 존재하는 DNA 영역에서 그렇다. 그림 아래의 버튼을 클릭하여 인터랙티브로 조작할 수 있는 이미지로 전환하여 보다 자세하게 이 구조를 살펴보기 바란다.
이해를 높이기 위한 토픽
참고문헌
- 6si7 2020 A dual-constriction biological nanopore resolves homonucleotide sequences with high fidelity. Nat Biotechnol 38 1415-1420
- 2017 Robust long-read native DNA sequencing using the ONT CsgG Nanopore system. Wellcome Open Res 2 23
- 2017 Nanopore DNA sequencing and genome assembly on the International Space Station. Sci Rep 7 18022
- 2017 Real-time DNA sequencing in the Antarctic dry valleys using the Oxford Nanopore sequencer. J Biomol Tech 28 2-7
- 2016 Three decades of nanopore sequencing. Nat Biotechnol 34 518-524
- 4uv3 2014 Structural and mechanistic insights into the bacterial amyloid secretion channel CsgG. Nature 516 250-253
- 2010 Processive replication of single DNA molecules in a nanopore catalyzed by phi29 DNA polymerase. J Am Chem Soc 132 17961–17972
- 1uun 2004 The structure of mycobacterial outer-membrane channel. Science 303 1189-1192
- 1xhz 2004 Insights into strand displacement and processivity from the crystal structure of the protein-primed DNA polymerase of bacteriophage phi29. Mol Cell 16 609-618.
- 7ahl 1996 Structure of staphylococcal alpha-hemolysin, a heptameric transmembrane pore. Science 274 1859-1866
 의 생체 고분자 학습 포털 사이트
의 생체 고분자 학습 포털 사이트 









