300: 鞭毛马达(Flagellar Motor)
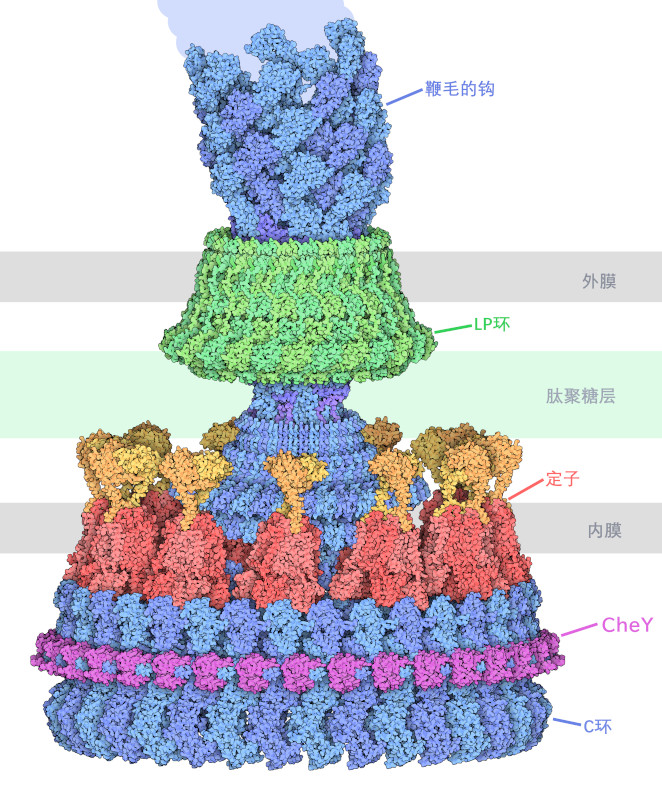
在本月的第 300 个 "当月的分子 "中,我们将探索生物分子世界奇迹之一的最新原子细节。鞭毛马达(flagellar motor) 几十年来一直吸引着生物学家。它是一个由大约 20 种不同蛋白质组成的巨大分子组合体,这些蛋白质共同组成了一个旋转马达。这种马达足以旋转长长的螺旋状鞭毛,其长度是细胞长度的好几倍。图中所示的沙门氏菌(Salmonella)马达每分钟旋转约 18,000 圈。有些细菌的马达旋转速度甚至比这还快。
改变路线
值得注意的是,细菌通过扭转鞭毛马达的旋转来控制其游动方向。沙门氏菌和大肠杆菌(E. coli)的细胞表面散布着大约十根鞭毛。当马达逆时针旋转时,所有鞭毛都会聚集在一起,为细胞朝一个方向前进提供动力。然而,如果细胞认为它没有朝着正确的方向前进,它就会将马达切换到顺时针方向。这会导致所有鞭毛分离,细胞就会在原地随意伸展。如果再将马达转回逆时针方向,鞭毛就会再次捆绑在一起,细胞就会朝着新的,可能更好的方向游去。
活动部件
鞭毛马达有许多活动部件,人们曾利用 X 射线晶体学和低温电子显微镜对这些部件分别进行了研究。这张图片结合了 PDB 中的几个条目(8ucs,8upl,7cgo,2zvy,1f4v),展示了马达的顺时针结构。顶部的钩子将马达与长鞭毛连接起来。LP环环绕中心轴,减少了与外膜和肽聚糖层的摩擦。动力由大约 11 个称为定子(stator)的旋转马达产生。每个马达都是由氢离子流过内膜驱动的旋转电机。这些较小的马达共同带动大型C环旋转,进而带动中轴和鞭毛旋转。然后,CheY蛋白决定旋转方向。
切换方向
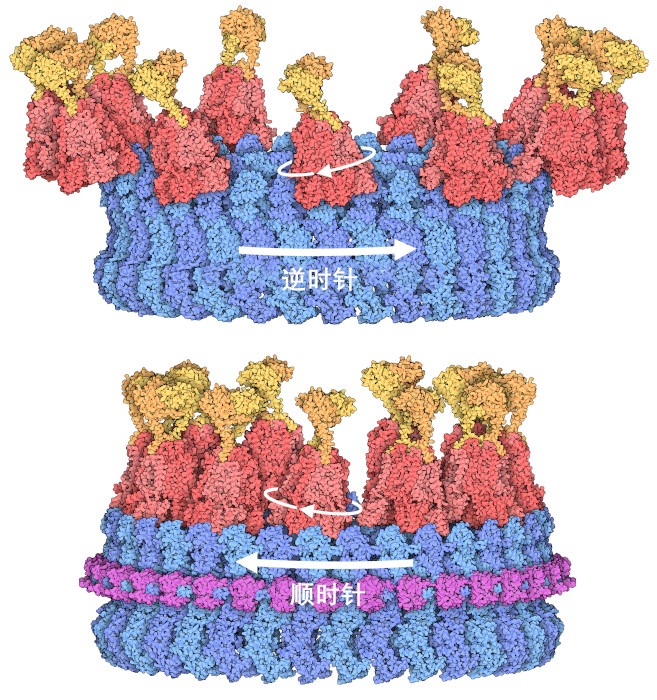
马达以直接方式切换旋转方向,就像我们在宏观世界中看到的机器一样:C环就像一个大齿轮,定子就像一个小齿轮。图的上半部分显示的是逆时针状态下的结构,由 PDB ID 8uox和 8ucs得出。定子位于C环的外部,当齿轮啮合时,C环的转动方向与定子的转动方向相反;当 CheY 加入时,C环的形状略有改变,定子的啮合位置变为环的内部(PDB IDs 8upl,8ucs和 1f4v)。在这种布局下,C环的旋转方向与定子的旋转方向相同。
探索结构
要切换到有互动控制的页面,请点击图表下面的按钮。如果加载没有开始,请尝试点击图表。
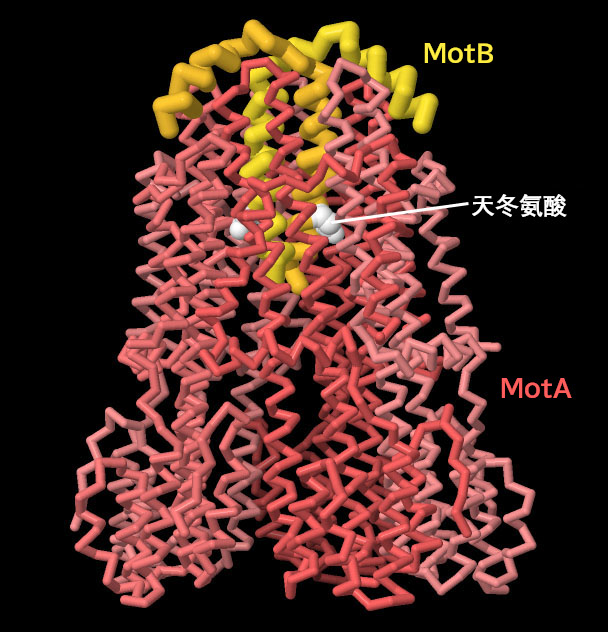
定子的原子结构证实了它是一个小型旋转马达的假设。定子由五个围绕一对 MotB 亚基旋转的 MotA 亚基组成,如 PDB ID 6ykm所示;MotB 上的一个重要氨基酸天冬氨酸(本结构中为 D22,大肠杆菌中为 D32)被认为可以管理为旋转提供动力的氢离子。MotB 还有一些结构域未包含在本结构中,这些结构域可到达细菌细胞壁中的肽聚糖层并与之结合,从而将定子固定在整个 鞭毛马达组合体中。点击图下按钮,切换到交互式可操作图像,更详细地了解这一结构。
参考文献
- 8ucs, 8uox, 8upl 2024 Structural basis of directional switching by the bacterial flagellum. Nat Microbiol 9 1282-1292
- 7cgo 2021 Structural basis of assembly and torque transmission of the bacterial flagellar motor. Cell 184 2665-2679.e19
- 6ykm 2020 Structure and function of stator units of the bacterial flagellar motor. Cell 183 244-257.e16
- 2zvy 2009 Snapshot of a Michaelis complex in a sulfuryl transfer reaction: Crystal structure of a mouse sulfotransferase, mSULT1D1, complexed with donor substrate and acceptor substrate. Biochem Biophys Res Commun 383 83-87
- 2006 The maximum number of torque-generating units in the flagellar motor of Escherichia coli is at least 11. Proc Natl Acad Sci USA 103 8066-8071
- 1f4v 2001 Crystal structure of an activated response regulator bound to its target. Nat Struct Biol 8 52-56
- 1998 The turn of the screw: the bacterial flagellar motor. Cell 93 17-20
 生物大分子学习的教育门户
生物大分子学习的教育门户 









