295: 核糖体多样性(Ribosome Diversity)
2000年首次确定的核糖体(ribosome)原子结构揭示了生物蛋白质合成的细节。核糖体的小亚基负责管理信使RNA,将其与适当的 转运RNA(transfer RNA)分子配对,并使密码子与反密码子相匹配。核糖体的大亚基将 转运RNA携带的氨基酸连接成新的蛋白质链。在这一发现之后的25年中,又有数千个核糖体结构被成功解析,许多蛋白质合成的步骤也被阐明。这些结构也使得比较不同生物的核糖体结构成为可能。
不断演变的复杂性
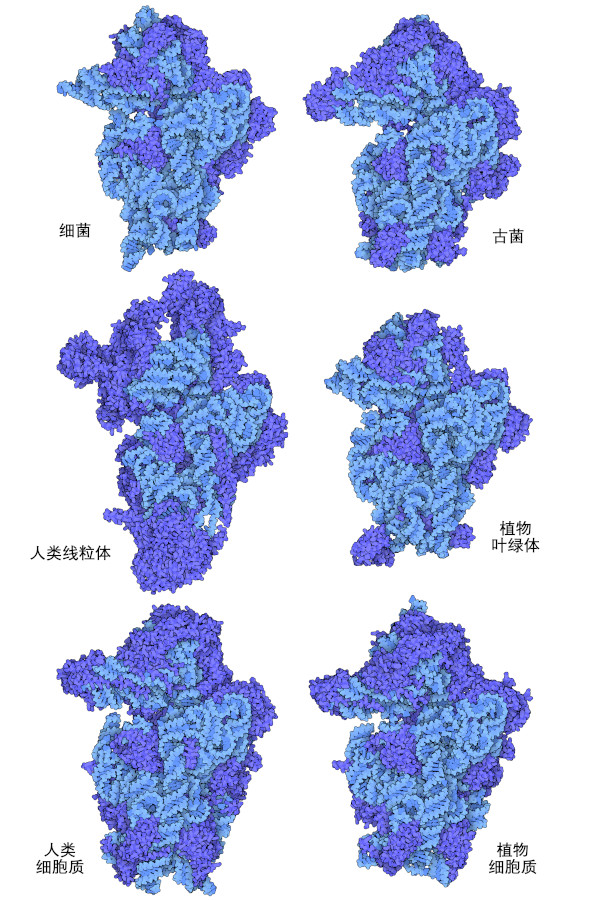
由于功能性核糖体对所有生物都是必需的,因此核糖体的结构对研究分子进化非常有用,并且可以对所有生物的核糖体进行对比。这里的图片展示了几种不同生物的核糖体小型亚基。细菌的核糖体结构相对简单,小亚基(PDB ID 1fjg)包含一条长度为 1522 个核苷酸的 RNA 链和 20 个蛋白质。同样,古细菌小亚基(PDB ID 6tmf)也有一条长度为 1485 个核苷酸的小 RNA 链和 29 个蛋白质。相比之下,人类和植物中的小亚基(PDB ID 5a2q,8auv)的结构更为复杂,其 RNA 链长度超过 1800 个核苷酸,蛋白质超过 30 种。
携手合作
线粒体(mitochondria)和叶绿体(chloroplast)制造自己的核糖体,与植物、动物和真菌细胞质中的核糖体不同。这两种细胞器都被认为是从细菌进化而来的,这些细菌在现代真核细胞祖先的细胞质中共生。叶绿体核糖体的结构是支持这种内共生假说的证据之一。叶绿体核糖体的小型亚基(PDB ID 5mmj)与细菌核糖体的小亚基相似,由长度为 1491 个核苷酸的短 RNA 和 25 个蛋白质组成。线粒体核糖体的小亚基(PDB ID 6rw4)的结构表明,线粒体核糖体与与其假定的细菌前身有显著差异,与细菌核糖体相比,线粒体核糖体的 RNA 链更短,只有 955 个核苷酸,蛋白质含量比典型的细菌核糖体更多。
启动的复杂性
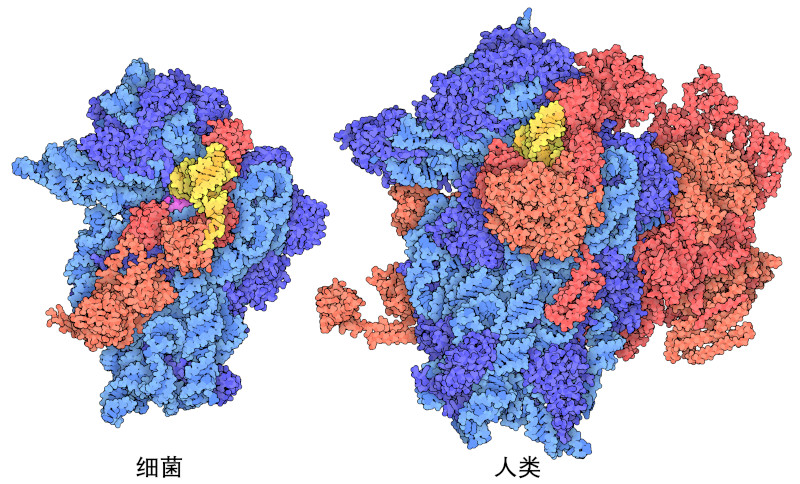
真核生物的复杂性还体现在协助核糖体进行蛋白质合成的许多蛋白质上。例如,这里显示的两个结构(PDB ID 5lmv, 8oz0)代表了协调蛋白质合成启动的蛋白质。它们一起帮助捕获信使RNA,并将起始 转运RNA 与翻译起始密码子对齐。细菌细胞只需要三个起始因子蛋白就能完成这项任务,而真核生物则需要大约 20 个。
探索结构
要切换到有互动控制的页面,请点击图表下面的按钮。如果加载没有开始,请尝试点击图表。
此交互视图比较了来自细菌和人类小亚基(PDB ID 1fjg, 5a2q)的核糖体 RNA。请注意,这两种结构都有一个相似的核心,它的基本任务是将信使 RNA 和 转运RNA结合在一起。就人类 RNA 而言,围绕这一核心有许多延伸部分,从而形成更大、更复杂的核糖体亚基。点击图片下方的按钮,切换到互动模式,更详细地了解这些结构。
进一步的讨论议题
参考文献
- 8oz0 2024 The structure of a human translation initiation complex reveals two independent roles for the helicase eIF4A. Nat Struct Mol Biol 31 455-464
- 8auv 2023 Structure of the actively translating plant 80S ribosome at 2.2 angstrom resolution. Nat Plants 9 987-1000
- 6rw4 2020 Distinct pre-initiation steps in human mitochondrial translation. Nat Commun 11 2932-2932
- 6tmf 2020 Molecular analysis of the ribosome recycling factor ABCE1 bound to the 30S post-splitting complex. EMBO J 39 e103788
- 5mmj 2017 The complete structure of the chloroplast 70S ribosome in complex with translation factor pY. EMBO J 36 475-486
- 5lmv 2016 Large-scale movements of IF3 and tRNA during bacterial translation initiation. Cell 167 133-144.e13
- 5a2q 2015 Cryo-EM structure of hepatitis C virus Ires bound to the human ribosome at 3.9 Angstrom Resolution. Nat Commun 6 7646
- 1fjg 2000 Functional insights from the structure of the 30S ribosomal subunit and its interactions with antibiotics. Nature 407 340-348
 生物大分子学习的教育门户
生物大分子学习的教育门户 









