295: リボソームの多様性(Ribosome Diversity)
2000年に決定されたリボソーム(ribosome)の最初の原子構造から、生物学的なタンパク質合成の詳細が明らかになった。リボソームの小サブユニットは伝令RNA(messenger RNA)を管理していて、然るべき転移RNA(transfer RNA)分子を持ってきて対合させ、コドンとそれに合うアンチコドンとを突き合わせる。リボソームの大サブユニットは、転移RNAが運んできたアミノ酸を新しいタンパク質鎖につなげる。この発見以降25年の間に、リボソームの構造がさらに何千も決定され、タンパク質の合成段階の多くが解明された。これらの構造によって、異なる生物が持つリボソームの構造を比較することもできるようになった。
進化する複雑性
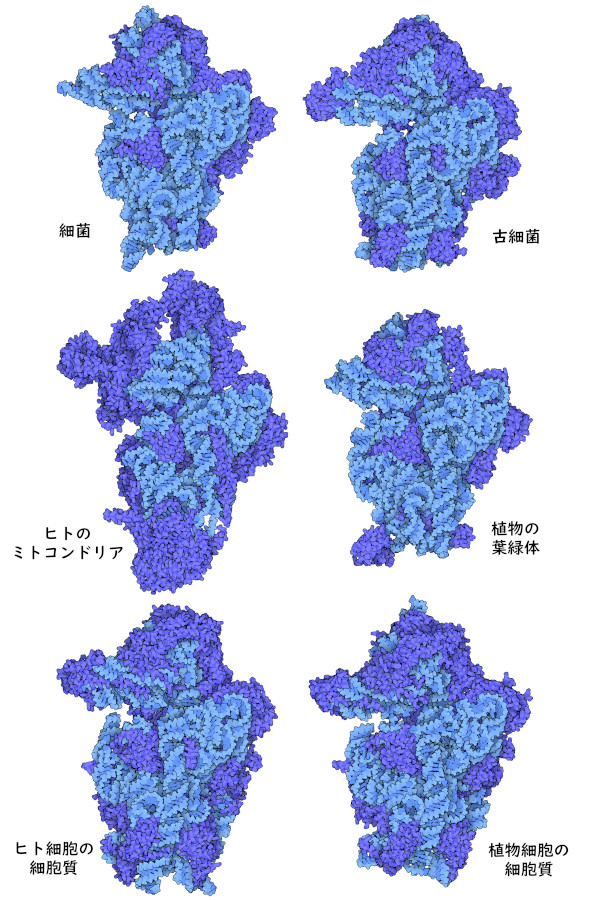
すべての生物にとって機能するリボソームは必要であるため、リボソームの構造は分子進化の研究にとって有用であり、あらゆる生物の間でリボソームを比較対照することができる。ここに示すのは、いくつかの生物から得られたリボソームの小サブユニットの図である。細菌のものは比較的単純な構造をしていて、小サブユニット(PDB ID 1fjg)には、長さが1522ヌクレオチドのRNA鎖が1本とタンパク質20個が含まれている。同様に、古細菌の小サブユニット(PDB ID 6tmf)は、1485ヌクレオチドの小さなRNA鎖と29個のタンパク質を持つ。一方、ヒトや植物の小サブユニット(PDB ID 5a2q、8auv)は、より複雑な構造へと進化させており、長さが1800ヌクレオチド以上のRNA鎖と30個以上のタンパク質を持つ。
共に働く
ミトコンドリア(mitochondria)と葉緑体(chloroplast)は独自のリボソームを作り、植物、動物、菌類の細胞質にあるリボソームとは異なっている。どちらの細胞小器官も、現代の真核細胞の祖先の細胞質に共生するようになった細菌から進化したと考えられている。葉緑体リボソームの構造は、この内部共生仮説を支持する証拠のひとつになっている。葉緑体リボソームの小サブユニット(PDB ID 5mmj)は、細菌のものとよく似ており、長さ1491ヌクレオチドの短いRNAと25個のタンパク質でできている。ミトコンドリアリボソーム小サブユニット(PDB ID 6rw4)の構造は、ミトコンドリアリボソームが祖先と推定される細菌のリボソームから大きく分岐したことを示していて、細菌のリボソームよりもさらに短い955ヌクレオチドのRNA鎖と、より多くのタンパク質を持っている。
イニシエーションの複雑さ
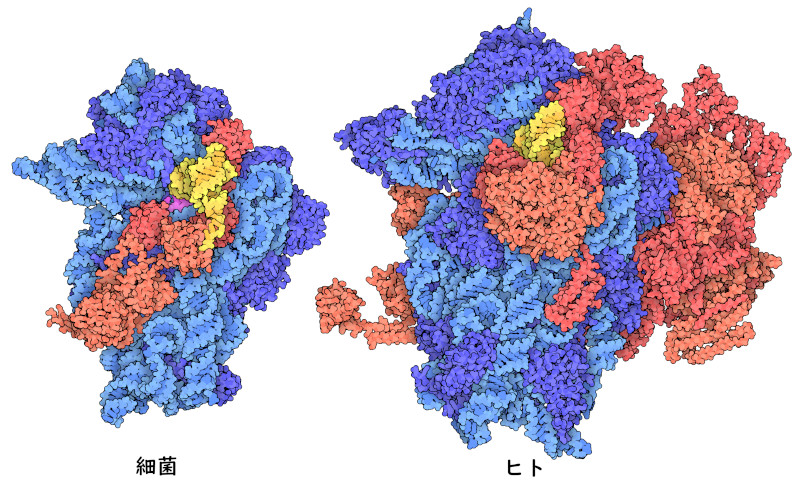
真核生物ではより複雑になっているという現象は、タンパク質合成の過程でリボソームを補助する多くのタンパク質でも見られる。例えば、ここに示す2つの構造(PDB ID 5lmv、8oz0)は、タンパク質合成の開始を指揮するタンパク質を示している。これらは共に、伝令RNAを捉え、翻訳開始コドンと開始転移RNAを並べるのを助ける。細菌細胞はこのタスクを成し遂げるのに3つの開始因子タンパク質しか必要としないが、真核生物は20個程度を必要とする。
構造をみる
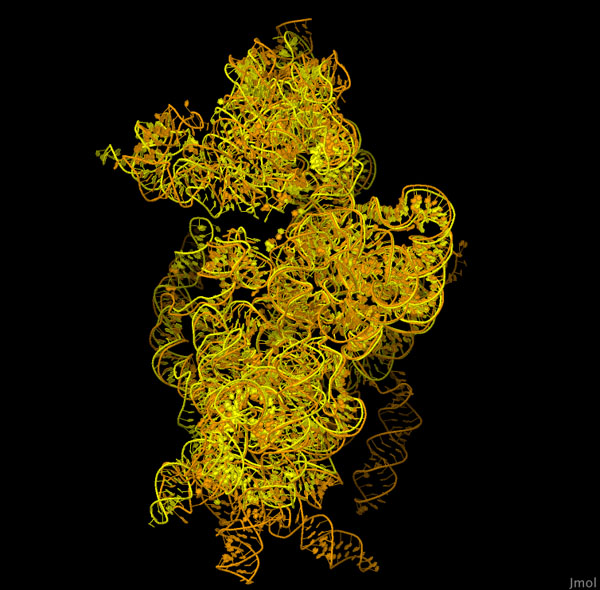
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
この対話的操作のできる表示では、細菌とヒトの小サブユニット(PDB ID 1fjg、5a2q)に由来するリボソームRNAを比較できるようにしている。どちらの構造にも、伝令RNAと転移RNAをまとめるという基本的な仕事を行う、同じような中核部分があることに注目してほしい。ヒトのRNAの場合、この中核部分の周囲に多くの拡張領域があり、より大きく複雑なリボソームサブユニットを作り出している。画像の下のボタンをクリックして対話的操作のできるモードに切り替え、これらの構造をさらに詳しく調べてみて欲しい。
理解を深めるためのトピックス
参考文献
- 8oz0 2024 The structure of a human translation initiation complex reveals two independent roles for the helicase eIF4A. Nat Struct Mol Biol 31 455-464
- 8auv 2023 Structure of the actively translating plant 80S ribosome at 2.2 angstrom resolution. Nat Plants 9 987-1000
- 6rw4 2020 Distinct pre-initiation steps in human mitochondrial translation. Nat Commun 11 2932-2932
- 6tmf 2020 Molecular analysis of the ribosome recycling factor ABCE1 bound to the 30S post-splitting complex. EMBO J 39 e103788
- 5mmj 2017 The complete structure of the chloroplast 70S ribosome in complex with translation factor pY. EMBO J 36 475-486
- 5lmv 2016 Large-scale movements of IF3 and tRNA during bacterial translation initiation. Cell 167 133-144.e13
- 5a2q 2015 Cryo-EM structure of hepatitis C virus Ires bound to the human ribosome at 3.9 Angstrom Resolution. Nat Commun 6 7646
- 1fjg 2000 Functional insights from the structure of the 30S ribosomal subunit and its interactions with antibiotics. Nature 407 340-348
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









