281: 纤维素酶和生物能源(Cellulases and Bioenergy)
石油衍生的燃料正在减少,并且是气候变化的主要促成因素。因此,生物能源研究人员正在转向自然界,为人类寻找更清洁、可再生的燃料。 有人提出,乙醇(ethanol)被认为是一种有吸引力的解决方案,可以作为燃料或作为生产可持续航空燃料的中间物。自史前时代以来,酒精饮料一直是通过糖的发酵来生产的,目前同样的技术也被用来生产作为燃料的乙醇。目前,大多数糖是用玉米和甘蔗生产的,但这并不理想,因为它占用了适合粮食生产的土地,而且这些作物的生产需要大量能源。因此,生物能源研究人员正在寻找将柳枝稷、杨树和玉米秆等耐寒非粮食作物中的纤维素(cellulose)转化为乙醇的方法。
纤维素的降解
纤维素是由葡萄糖(glucose)分子组成的,这些葡萄糖分子彼此紧密结合,形成稳定的聚合物。纤维素是由纤维素合酶(cellulose synthase)产生的,它将许多纤维素链层叠成更坚韧的纤维使其更牢固。因此,工业流程需要分离这些纤维束并将其切割成糖,然后才能发酵成乙醇。通常,通常情况下,植物材料会经过化学药品和 "蒸汽爆炸 "等侵蚀性工艺的预处理,这些工艺会破坏植物细胞壁,并提供获取纤维素纤维的途径。然后,纤维素酶混合物将纤维素链分解成单个糖。纤维素酶是由一种叫做Trichoderma reesei(也被称为 Hypocrea jecorina)的真菌制成。这是第二次世界大战期间在所罗门群岛发现的一个物种,生长在棉织物上。如今,它广泛用于生物乙醇生产以及其他任务,例如软化石洗牛仔裤中的牛仔布。
纤维素酶科学
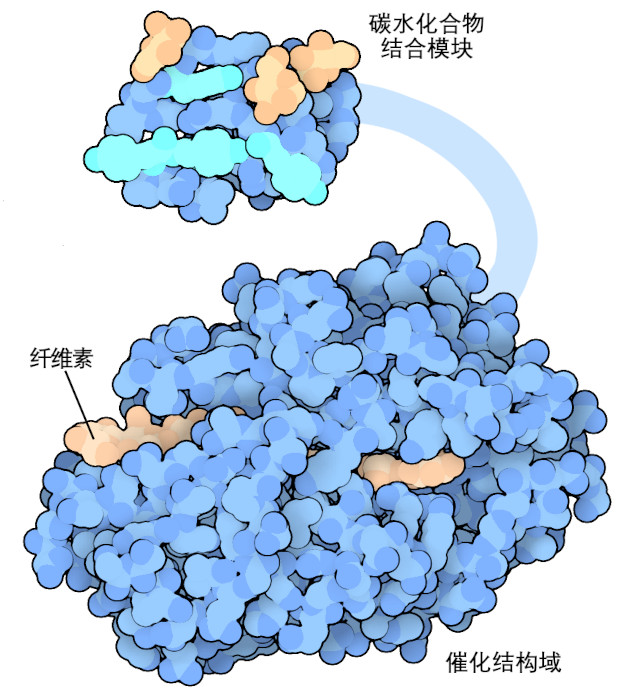
一些纤维素酶含有两个结构域用于分解坚固的纤维素纤维(这里显示的是PDB条目7cel和2mwk的结构)。其中一个结构域有一个长凹槽,形状适合与纤维素链结合,拉紧一个葡萄糖-葡萄糖键并将其定位在催化机制上进行裂解。另一个结构域与纤维素结合,确保酶始终处于正确的位置,使其能够快速连续地进行许多裂解反应。。
纤维素酶鸡尾酒
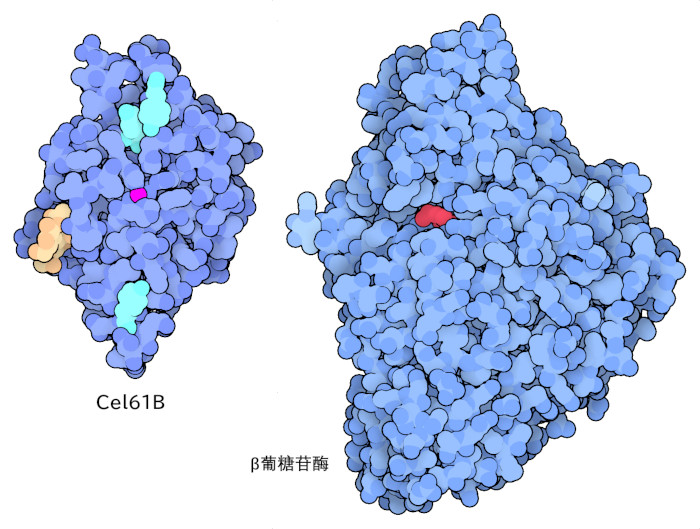
像Trichoderma reesei这样的真菌,会分泌一种复杂的纤维素降解酶混合物,这些酶共同作用来攻击纤维素纤维。这里包含两个示例,并且在PDB存档中提供了许多其他示例的结构。Cel61B,被称为内切葡聚糖酶7(endoglucanase 7),利用金属离子产生活性氧,以攻击性的方式攻击纤维素(PDB条目2vtc)。与Cel7A的纤维素结合结构域类似,Cel61B它具有多个酪氨酸和糖基化,有助于将其粘合到纤维素纤维上。β-葡萄糖苷酶(beta-glucosidase,PDB条目3zyz)在此过程的后期发挥作用,将纤维素短片段剪切成单个葡萄糖。
探索结构
要切换到有互动控制的页面,请点击图表下面的按钮。如果加载没有开始,请尝试点击图表。
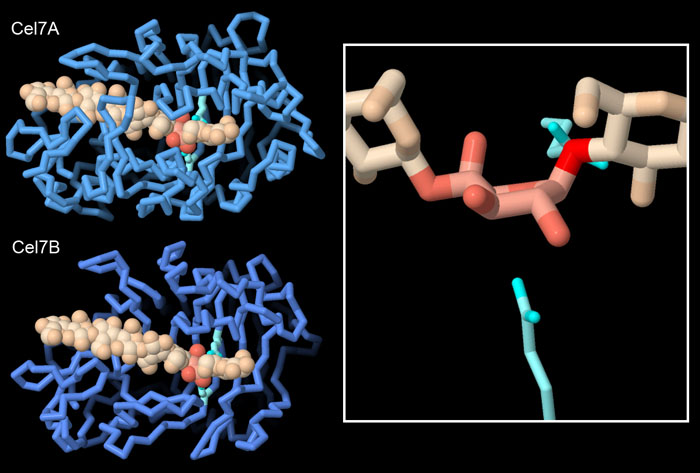
纤维素酶使用一些分子技巧来使其功能更加有效。如上所述,纤维素酶通常具有与纤维素纤维表面结合的结构域或表面。与纤维素结合的部位的形状也是量身定做的,以实现专门的功能:在Cel7A(PDB条目4c4c)中,几个环状物围绕着纤维素聚合物,与一端的活性位点形成一个隧道。因此,Cel7A 能从纤维素纤维的末端裂解出小的双糖片段。另一方面,Cel7B(内切酶EG-1,PDB条目1eg1)缺少这些环,它有一个开放的沟槽,可以与纤维素长链上的任何部位结合,使其能够在纤维素链的中间裂解纤维素。图中右侧的放大图显示了另一种分子技巧。这些酶经常将一个糖环(这里显示为浅红褐色)扭曲成一个不太稳定的排列。这种扭曲的作用是激活氧(红色),从而在两个谷氨酸(浅蓝色)催化的裂解反应中将单糖连接在一起。点击图下方的按钮,切换到互动图像,以更详细地查看这些结构。请注意,PDB条目4c4c的纤维素链与两种蛋白质一起显示。
进一步的讨论议题
- 你可以使用PDBj搜索或RCSB PDB注释浏览器找到其他纤维素酶结构:3.2.1.4(纤维素酶纤维素酶: PDBj / RCSB PDB)或3.2.1.91(纤维素1,4-β-纤维素酶PDBj / RCSB PDB)。
- 要看到连接Cel7A两个结构域的灵活环路,请看由AlphaFold2预测的结构AF_AFP62694F1。注意连接体的末端有一些用于灵活的脯氨酸和一些可能被糖基化的苏氨酸和丝氨酸。
- 关于纤维素酶的科学的一点说明:有许多不同类型的纤维素酶,科学家喜欢对事物进行分类和命名。这就是为什么同一种酶往往有几个不同的名字。例如,Cel7A这个短名称是基于纤维素酶家族中的广泛分类,其中包括1,4-beta-D-葡聚糖纤溶酶I(1,4-beta-D-glucan cellobiohydrolase I)和纤维素1,4-beta纤溶酶(cellulose 1,4-beta cellobiosidase)和外切葡聚糖酶I(exoglucanase I)等长名称,是指其从纤维素链末端裂解二糖单位的作用。
参考文献
- 2020 Current state-of-the-art in ethanol production from lignocellulosic feedstocks. Microbiol Res 240 126534
- 2mwk 2015 O-glycosylation effects on family 1 carbohydrate-binding module solution structures. FEBS J 282 4341-4356
- 2015 Fungal cellulases. Chem Rev 115 1308-1448
- 3zyz 2014 Biochemical Characterization and Crystal Structures of a Fungal Family 3 Beta-Glucosidase, Cel3A from Hypocrea Jecorina. J Biol Chem 289 31624-31637
- 4c4c 2014 The Mechanism of Cellulose Hydrolysis by a Two-Step, Retaining Cellobiohydrolase Elucidated by Structural and Transition Path Sampling Studies. J Am Chem Soc 136 321-329
- 2vtc 2008 The First Structure of a Glycoside Hydrolase Family 61 Member, Cel61B from the Hypocrea Jecorina, at 1.6 A Resolution. J Mol Biol 383 144-154
- 1998 High-resolution crystal structures reveal how a cellulose chain is bound in the 50 A long tunnel of cellobiohydrolase I from Trichoderma reesei. J Mol Biol 275 309-325
- 1eg1 1997 The crystal structure of the catalytic core domain of endoglucanase I from Trichoderma reesei at 3.6 A resolution, and a comparison with related enzymes. J Mol Biol 272 383-397
 生物大分子学习的教育门户
生物大分子学习的教育门户 









