246: SARSコロナウイルス2型 スパイク(SARS-CoV-2 Spike)
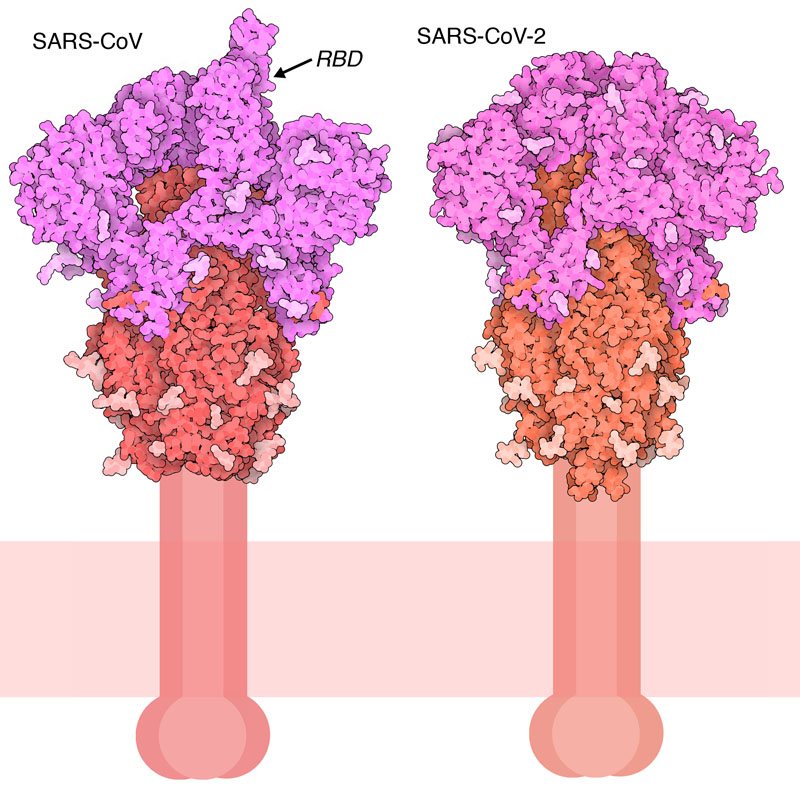
以前流行したSARSコロナウイルス(SARS-CoV)に関する長年の研究に基づき、現在流行している新型コロナウイルス(SARS-CoV-2)への対策が早急に進められている。このウイルスのスパイクタンパク質(spike protein)は、ウイルスに対抗する免疫の元となる抗体の主な標的となるため、感染症対策においてまず第一に研究対象となるタンパク質である。コロナウイルスは、表面がこのスパイクで覆われているため、電子顕微鏡で見た外観は独特な王冠状になっている。スパイクは受容体と結合し、細胞膜と融合してウイルスゲノムを細胞の中に放出することで一連の感染過程が始まる。他のエンベロープを持つウイルスも多くがこれに似たスパイク様のタンパク質を使って細胞に感染する。例えば、インフルエンザウイルスの赤血球凝集素(influenza hemagglutinin)、そしてエイズウイルス-1(HIV-1)やエボラウイルス(Ebolavirus)のエンベロープ糖タンパク質が挙げられる。
適当なサイズにカット
スパイクタンパク質は3つの同じ鎖で構成され、これらが集まってウイルス内部にある小さなドメイン、膜につながる部分、そしてウイルスの外に突き出した大きな外部ドメインをつくっている。さらに、スパイクは糖タンパク質であり、外部ドメインはウイルスを免疫系から隠すのに役立つ糖鎖で覆われている。SARSコロナウイルスと新型コロナウイルス(SARS-CoV-2)から得られたスパイクタンパク質の外部ドメインだけの構造(6crz、6vxx)をここに示す。ご覧の通り両者は非常によく似ている。各鎖はまずつながった1本の鎖として合成され、その後細胞が持つプロテアーゼによって切断されて2つの機能単位に分かれる。外側のS1領域(赤紫)は細胞の受容体と結合し、S2領域(赤)はウイルスと細胞の融合を指示する。どちらの構造も糖鎖についてはスパイクタンパク質を覆う部分しか含まれていない。なぜなら糖は柔軟性が高く観察が難しいからである。
柔軟であるという特徴
最近解明された構造から、SARS-CoVとSARS-CoV-2のスパイクはどちらも非常に柔軟であることが分かった。これら構造を見ると、受容体結合ドメインの配置が異なっている。ドメインが一つだけが上に出ているいうことはよくあるが、すべてがひっこんでいたり複数のドメインが外に出ていたりすることもある。外に出た配置は受容体と結合するのに必要なので、この柔軟さはウイルスにとって非常に有利である。最近の変異型SARSは受容体結合ドメインの柔軟性が特に高いので悪性が高く、より広まっていて一般的な風邪を引き起こすコロナウイルスは柔軟性が低いので危険度はまだましであるという仮説が立てられている。
受容体との結合
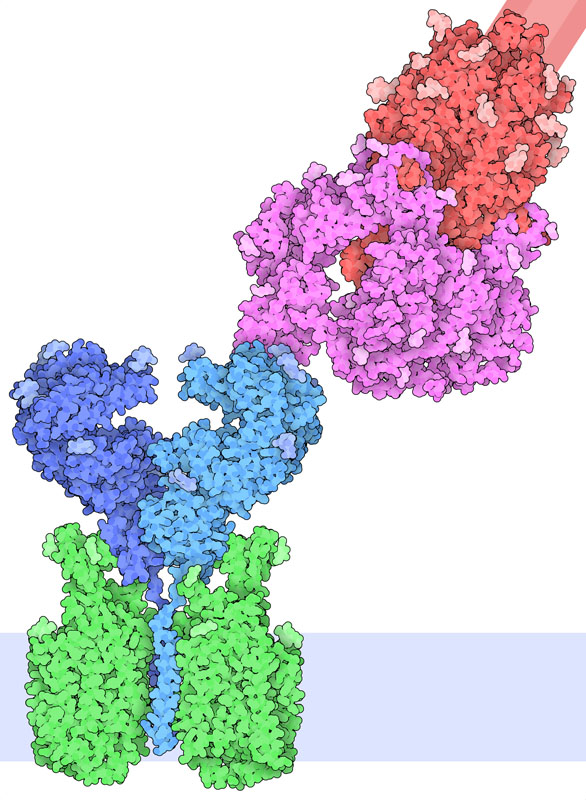
スパイクタンパク質は細胞表面にあるACE2(アンギオテンシン変換酵素2、angiotensin-converting enzyme 2)に結合する。ACE2は血圧の調節に関係するペプチドホルモンの一つアンギオテンシンを活性化する酵素である。ACE2は肺、心臓、腎臓、腸の細胞で見られ、これらの細胞はウイルスによる感染の対象となる。PDBエントリー6m17の構造によって、ACE2にSARS-CoV-2スパイクタンパク質の受容体結合ドメインが結合した複合体の構造が明らかになった。複合体の中で、ACE2はアミノ酸輸送体B0AT1にも結合している。ここに示すイラストはこの複合体の構造に、PDBエントリー6vsbから得られたスパイクタンパク質の構造を重ね合わせて描いたものである。
構造をみる
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
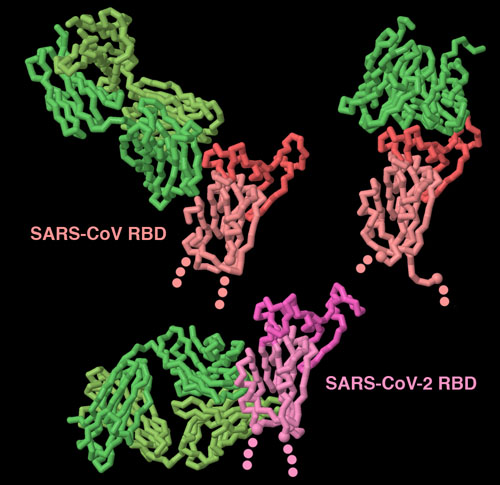
私たちがコロナウイルスに感染すると、私たちの免疫系はこれに対抗する。スパイクはウイルス表面に露出しているため、この対抗措置における主な対象となる。これら3つの構造(PDBエントリー3bgf、2ghw、6w41)は抗体(緑)がさまざまな方法でスパイクタンパク質を認識できることを示している。このうち2つはドメインの受容体結合部位(濃い色で示す部分)を阻害している。そしてもう1つの抗体はドメインの基部に隠れた部分を対象としている。この部分は抗体が結合したときにしか外に露出しない。これらの構造をより詳しくみるため、図の下のボタンをクリックして対話的操作のできる画像に切り替えてみて欲しい。
理解を深めるためのトピックス
- 新型コロナウイルス(SARS-CoV-2)および新型コロナウイルスによる感染症(COVID-19)について、より詳しい情報がメインのRCSB PDBサイト、RCSB PDBの教育サイトPDB-101およびPDBjにある。
参考文献
- 6vxx 2020 Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein. Cell 181 281-292
- 6w41 2020 A highly conserved cryptic epitope in the receptor-binding domains of SARS-CoV-2 and SARS-CoV. Science 10.1126/science.abb7269
- 6vsb 2020 Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science 367 1260-1263
- 6m17 2020 Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2. Science 367 1444-1448
- 6crz 2018 Stabilized coronavirus spikes are resistant to conformational changes induced by receptor recognition or proteolysis. Sci Rep 8 15701-15701
- 3bgf 2009 Structural insights into immune recognition of the severe acute respiratory syndrome coronavirus S protein receptor binding domain. J Mol Biol 388 815-823
- 2ghw 2006 Structural basis of neutralization by a human anti-severe acute respiratory syndrome spike protein antibody, 80R. J Biol Chem 281 34610-34616
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









