259: 人工設計タンパク質と市民科学(Designed Proteins and Citizen Science)
この記事は、2021年1月に定量生物医学ラトガース研究所(Rutgers Institute for Quantitative Biomedicine)の主催で開催された1週間の集中合宿「生物医学における科学コミュニケーション」(Science Communication in Biology and Medicine)の一環で、チャンペン・ルー(Changpeng Lu)、ナタリー・ロサダ(Natalie Losada)、ニティッシュ・セルバラジュ(Nithish Selvaraj)により執筆され、描かれたものです。
デノボ設計を一目見る
蛋白質構造データバンク(Protein Data Bank、PDB)は1971年に発足して以来、生体分子構造の中心的アーカイブとなっている。タンパク質や核酸およびこれらとの複合体の構造は、生物学における分子構造と機能の関係を理解し、ポリペプチド鎖がどのようにして機能するタンパク質へと折りたたまれていくのかを学ぶのに役立っている。最近この知見を使って個別調整タンパク質をデノボ設計(de novo design、一から設計)し、プラスチックの分解や特定の薬を効率的につくるなど21世紀の問題を解決することを目指す試みが行われている。AlphaFold(アルファフォールド)、やRosetta(ロゼッタ)のようなタンパク質の折りたたみと設計を行うプログラムは、人工的に設計したタンパク質の構造を予測することができる。しかし、これらのタンパク質は機能を確認するために実際合成したとき必ずしも安定であるとは限らない。科学者たちはこのような場面において、タンパク質の設計を改良する手助けをゲーマーや市民科学者に求めている。
一つの解決策: Foldit
Foldit(フォールドイット) は、タンパク質の配列を設計してその構造を予測することができる対話的なコンピュータゲームである。プレイヤーは、その使いやすいインタフェースを使って空間的な直観に基づき構造を操作しながら、Rosettaプログラムが設定した水素結合や疎水的相互作用などの相互作用を安定化するためのルールにもしたがって、ゲームの最高得点を目指す。科学者はこの挑戦を始めるにあたり、既に折りたたまれた構造を修正しただけのものから、完全に一から設計されたタンパク質の構造予測まで、さまざまな難易度の折りたたみパズルをアップロードした。プレイヤーは三次元の問題解決能力を駆使し、純粋な計算的手法では見逃されてしまうようなユニークな構造を探し出す。こうして設計されたタンパク質はその後科学者によって実際に合成され、意図した特性や機能を持っていることが実験室で確認されれば、ユーザは予測された構造を実際のタンパク質として見ることができる。
デノボ設計の影響
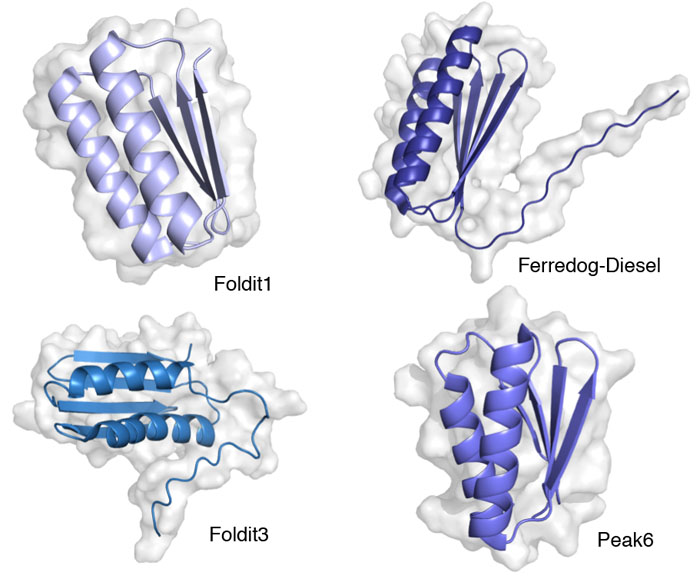
新たなタンパク質折りたたみ様式は、新たな酵素やその他の機能タンパク質などをつくる、デノボ設計において重要なものである。科学者は、Rosettaのようなアルゴリズムを使って、Top7(1qys、ここには図示していない)のような新しいタンパク質を一から設計している。これらの新たな折りたたみ様式は、ウイルスの感染防止に役立つタンパク質(PDBエントリー7jzlと3r2x、ここには図示していない)のような新酵素をつくる基礎となったり、細胞内で特定の化学物質を追跡できる分子をつくることにつながったりする。Folditプレイヤーは56種類のタンパク質を一から作り上げた。そのうちの一つは新たに発見されたタンパク質の折りたたみ様式を持っていた! それらの新たなタンパク質の中から、実験でつくられた4つの構造をここに示す(PDBエントリー6mrr、6nuk、6msp、6mrs)。 新たな計算設計ツールのおかげで、一から設計したタンパク質の可能性がこれまで以上に広がっている。
タンパク質の構造予測と設計
デノボ設計を行うために開発されたツールは、構造予測と最適化にとっても非常に効果的である。ここでは3つの事例を示す。1つ目は、構造最適化を使って、ポリエステルのプラスチックを分解するという困難な化学的作業を効率的に行うPETデポリメラーゼ(PET depolymerase、PDBエントリー6tht、ここには図示せず)を設計した例である。2つ目は、10年間の試行錯誤の末、Folditプレイヤーの助けも借りてついにメイソン-ファイザー モンキーウイルス(Mason-Pfizer monkey virus、M-PMV)が持つプロテアーゼの構造(PDBエントリー3sqf、ここには図示していない)を決定した例で、この構造は標的薬の発見に活用できる。そして最後は、Folditプレイヤーが酵素活性を10倍以上に高め、既存タンパク質の機能を改良することができた例である。この出発点は、特殊なディールス・アルダー反応(Diels-Alder reaction)を行う、人工タンパク質(PDBエントリー3i1c)であった。Folditプレイヤーは科学者の案内にしたがって酵素の「ふた」をつくり、基質をより強く酵素に固定して触媒がより効率的に働くようにした(PDBエントリー3u0s)。
構造をみる
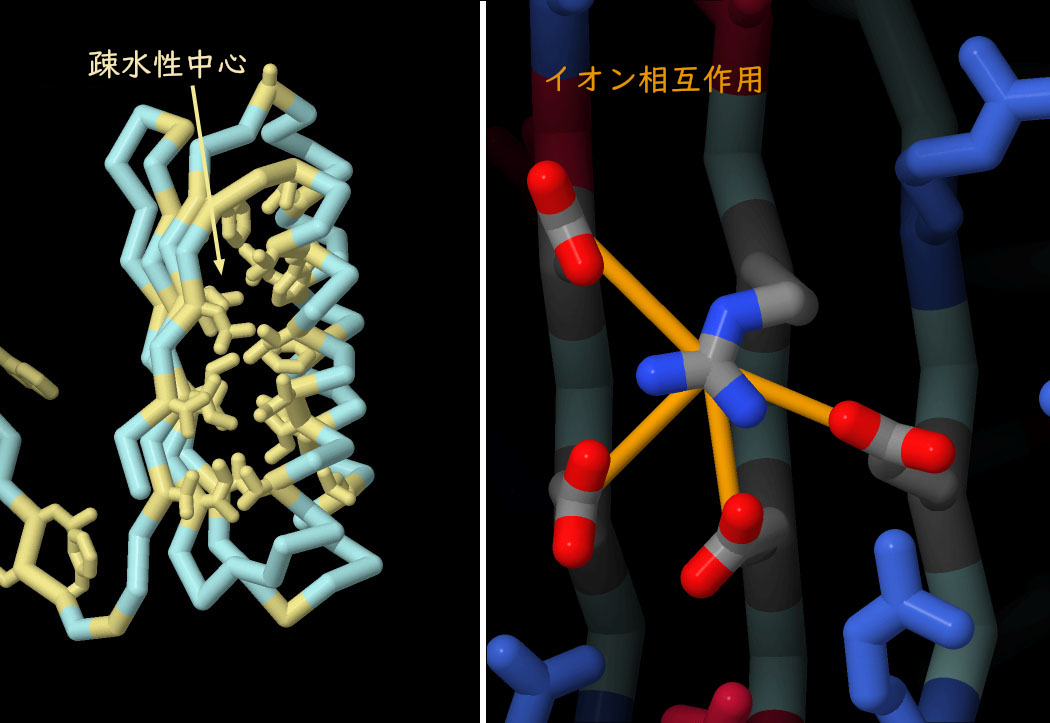
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
タンパク質を安定な三次元構造を取るため、さまざまな種類の相互作用が個々のアミノ酸間で行われる。炭素が豊富な側鎖は酵素の内側に集まって疎水性中心(hydrophobic core)をつくる。一方電荷を帯びていたり極性があったりする側鎖はタンパク質の表面に並んで周囲の水と相互作用することが多い。イオン相互作用(ionic interaction)や水素結合(hydrogen bond)など特定の相互作用がさらにタンパク質を安定化させ、ポリペプチド鎖の局所的な細かい部分が折りたたまれるよう導く。図の下のボタンをクリックし、対話的操作のできる図に切り替えると、Folditで設計されたタンパク質(PDBエントリー6nuk)におけるこれらの相互作用を見ることができる。
理解を深めるためのトピックス
参考文献
- 2020 Improved protein structure prediction using potentials from deep learning. Nature 577 706–710
- 2020 An engineered PET depolymerase to break down and recycle plastic bottles. Nature 580 216–219
- 6mrs、6mrr、6msp、6nuk 2019 De novo protein design by citizen scientists. Nature 570 390–394
- 2015 A General Strategy to Construct Small Molecule Biosensors in Eukaryotes. ELife 4
- 3u0s 2012 Increased Diels-Alderase activity through backbone remodeling guided by Foldit players. Nature Biotechnology 30(2) 190-192
- 2010 Predicting protein structures with a multiplayer online game. Nature 466 756–760
- 3i1c 2010 Computational design of an enzyme catalyst for a stereoselective bimolecular Diels-Alder reaction. Science 329 309-313
- 1999 Ab initio protein structure prediction of CASP III targets using ROSETTA. Proteins 37 171-176
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









