259: 인공합성단백질과 시민 과학 (Designed Proteins and Citizen Science)
이 기사는 2021년 1월에 정량생물의학 럿거스연구소(Rutgers Institute for Quantitative Biomedicine) 주최로 개최된 1주일간의 집중 합숙 ‘생물의학에서의 과학 커뮤니케이션’(Science Communication in Biology and Medicine)의 일환으로 창펭 루(Changpeng Lu), 나탈리 로사다(Natalie Losada), 니티시 세르바라주(Nithish Selvaraj)에 의해 집필되고 그려진 것입니다.
드 노보 디자인을 한눈에 보다
단백질 정보 은행(Protein Data Bank, PDB)는 1971년 출범한 이래 생체분자 구조의 중심적 아카이브가 되었다. 단백질과 핵산 및 이들과의 복합체 구조는 생물학에서 분자 구조와 기능의 관계를 이해하고 폴리펩타이드 사슬이 어떻게 기능하는 단백질로 접혀져 가는지 배우는 데 도움을 준다. 최근 이 식견을 사용해 개별 조정 단백질을 드 노보 디자인(de novo design, 처음부터 설계)을 하고, 플라스틱 분해나 특정 약을 효율적으로 만드는 등 21세기의 문제를 해결하는 것을 목표로 하는 시도가 이루어지고 있다. AlphaFold(알파폴드)나 Rosetta(로제타) 같은 단백질의 접기와 설계를 수행하는 프로그램은 인공적으로 설계한 단백질의 구조를 예측할 수 있다. 그러나 이 단백질들은 기능을 확인하기 위해 실제 합성했을 때 반드시 안정적이지는 않다. 과학자들은 이런 상황에서 게이머나 시민과학자에게 단백질 설계를 개량하는 데 도움을 줄 것을 요구하고 있다.
한 가지 해결책: Foldit
Foldit(폴딧)은 단백질 배열을 설계하여 그 구조를 예측할 수 있는 인터랙티브 컴퓨터 게임이다. 플레이어는 사용하기 쉬운 인터페이스를 사용하여 공간적 직관에 기초하여 구조를 조작하면서 Rosetta 프로그램이 설정한 수소 결합이나 소수적 상호 작용 등의 상호 작용을 안정화하기 위한 규칙에도 따라 게임의 최고 점수를 목표로 한다. 과학자는 이 도전을 시작하면서 이미 접힌 구조를 수정만 한 것부터 완전히 처음부터 설계된 단백질의 구조 예측까지 다양한 난이도의 접이식 퍼즐을 업로드했다. 플레이어는 3차원의 문제 해결 능력을 구사하여 순수한 계산적 수법으로는 간과되어버리는 독특한 구조를 찾아낸다. 이렇게 설계된 단백질은 이후 과학자에 의해 실제로 합성되어 의도한 특성이나 기능을 가지고 있음이 실험실에서 확인되면 사용자는 예측된 구조를 실제 단백질로 볼 수 있다.
드 노보 디자인의 영향
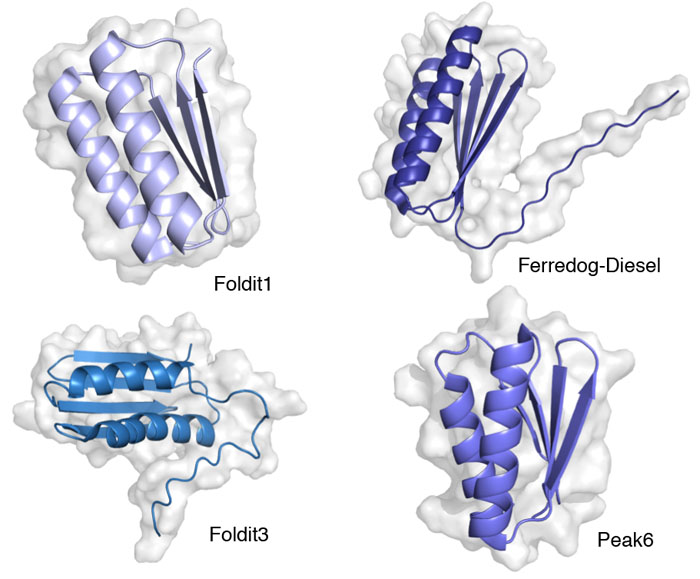
새로운 단백질 접이식 양식은 새로운 효소나 기타 기능 단백질 등을 만드는 드 노보 디자인에 있어서 중요한 것이다. 과학자는 Rosetta와 같은 알고리즘을 사용하여 Top7(1qys, 여기에는 도시하지 않음)과 같은 새로운 단백질을 처음부터 설계하고 있다. 이러한 새로운 접이식 양식은 바이러스의 감염 방지에 도움이 되는 단백질(PDB 엔트리 7jzl과 3r2x, 여기에는 도시하지 않음)과 같은 새로운 효소를 만드는 기초가 되기도 하고 세포 내에서 특정 화학물질을 추적할 수 있는 분자를 만드는 것으로 이어지기도 한다. Foldit 플레이어는 56종류의 단백질을 처음부터 만들었다. 그중 하나는 새로 발견된 단백질 접이식 양식을 가지고 있었다! 그러한 새로운 단백질 중에서 실험으로 만들어진 4가지 구조를 여기에 나타낸다(PDB 엔트리 6mrr, 6nuk, 6msp, 6mrs). 새로운 계산 설계 도구 덕분에 처음부터 설계한 단백질의 가능성이 그 어느 때보다 넓어지고 있다.
단백질의 구조 예측과 설계
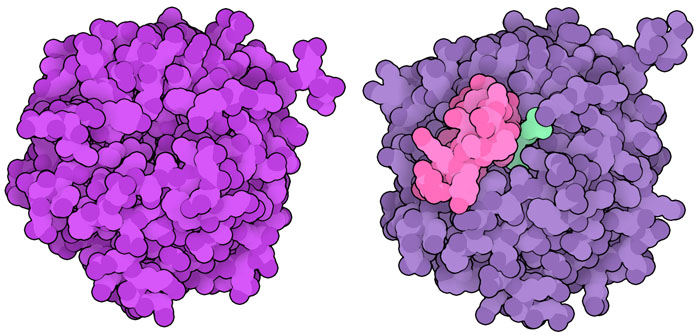
드 노보 디자인을 수행하기 위해 개발된 툴은 구조 예측과 최적화에도 매우 효과적이다. 여기서는 3가지 사례를 나타낸다. 첫 번째는 구조 최적화를 사용하여 폴리에스테르 플라스틱을 분해하는 어려운 화학적 작업을 효율적으로 수행하는 PET 분해효소(PET depolymerase, PDB 엔트리 6tht, 여기에는 도시하지 않음)를 설계한 예이다. 두 번째는 10년간의 시행착오 끝에 Foldit 플레이어의 도움으로 마침내 메이슨-화이자 원숭이 바이러스(Mason-Pfizer monkey virus, M-PMV)가 가지는 프로테아제의 구조(PDB 엔트리 3sqf, 여기에는 도시하지 않음)를 결정한 예로, 이 구조는 표적약의 발견에 활용할 수 있다. 그리고 마지막은 Foldit 플레이어가 효소 활성을 10배 이상 높여 기존 단백질의 기능을 개량할 수 있었던 예이다. 이 출발점은 특수한 딜스-알더반응(Diels-Alder reaction)을 수행하는 인공 단백질(PDB 엔트리 3i1c)이었다. Foldit 플레이어는 과학자의 안내에 따라 효소의 ‘뚜껑’을 만들어 기질을 보다 강하게 효소에 고정시켜 촉매가 보다 효율적으로 작용하도록 했다(PDB 엔트리 3u0s).
구조 보기
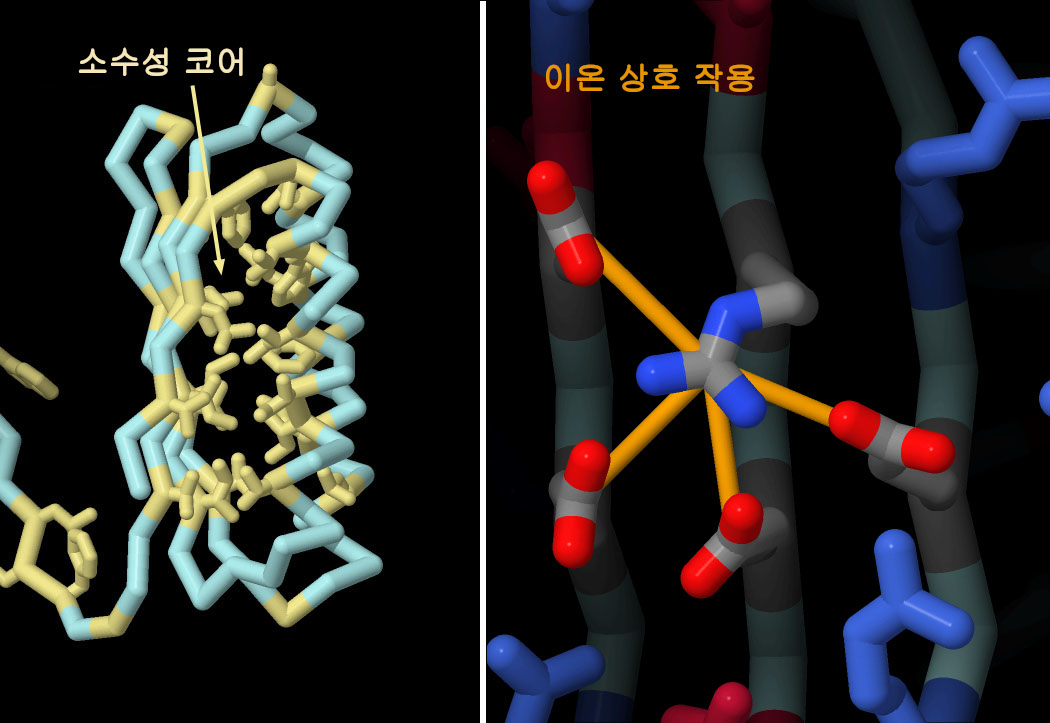
인터랙티브 조작이 가능한 페이지로 전환하려면 그림 아래의 버튼을 클릭하십시오. 읽기가 시작되지 않을 때는 그림을 클릭하십시오.
단백질을 안정적인 3차원 구조를 취하기 위해 다양한 종류의 상호 작용이 개별 아미노산 사이에서 이루어진다. 탄소가 풍부한 곁사슬은 효소의 안쪽에 모여 소수성 코어(hydrophobic core)를 만든다. 한편 전하를 띠거나 극성이 있는 곁사슬은 단백질 표면에 나란히 있어서 주위의 물과 상호 작용하는 경우가 많다. 이온 상호 작용(ionic interaction)이나 수소결합(hydrogen bond) 등 특정 상호 작용이 더욱 단백질을 안정화시켜 폴리펩타이드 사슬의 국소적인 미세한 부분이 접히도록 돕는다. 그림 아래의 버튼을 클릭하여 인터랙티브 조작이 가능한 그림으로 전환하면 Foldit으로 설계된 단백질(PDB 엔트리 6nuk)에서의 이러한 상호 작용을 볼 수 있다.
이해를 높이기 위한 토픽
참고문헌
- 2020 Improved protein structure prediction using potentials from deep learning. Nature 577 706–710
- 2020 An engineered PET depolymerase to break down and recycle plastic bottles. Nature 580 216–219
- 6mrs、6mrr、6msp、6nuk 2019 De novo protein design by citizen scientists. Nature 570 390–394
- 2015 A General Strategy to Construct Small Molecule Biosensors in Eukaryotes. ELife 4
- 3u0s 2012 Increased Diels-Alderase activity through backbone remodeling guided by Foldit players. Nature Biotechnology 30(2) 190-192
- 2010 Predicting protein structures with a multiplayer online game. Nature 466 756–760
- 3i1c 2010 Computational design of an enzyme catalyst for a stereoselective bimolecular Diels-Alder reaction. Science 329 309-313
- 1999 Ab initio protein structure prediction of CASP III targets using ROSETTA. Proteins 37 171-176
 의 생체 고분자 학습 포털 사이트
의 생체 고분자 학습 포털 사이트 









