245: 스플라이소좀 (Spliceosomes)
생물학에서 이 정도로 단순한 것은 없다! 유전정보의 흐름에 관한 기본적인 구조는 세균에서 발견된 것으로, 그것은 먼저 DNA에서 전령 RNA(messanger RNA, mRNA)로 전사(transcript)되고 그 다음에 그것이 단백질로 번역(translate)되는 것을 나타내고 있었다. 그러나 더 복잡한 생물로 알아보려니 이야기가 복잡해졌다. 우리의 DNA에 코딩되어 있는 유전자는 일반적으로 단백질로 번역되는 전령 RNA 사슬보다도 4~10배는 길다. 우리의 게놈을 더 자세히 보면 대부분의 유전자 단백질의 코드 부분은 엑손(exon)이라고 불리는 구간으로 나누어져 있고, 그것들은 인트론(intron)이라고 불리는 긴 비(非)코드 배열로 구분되어 있다. 인트론의 수는 단백질에 따라 다양하여, 예를 들어 우리의 헤모글로빈(hemoglobin) 유전자는 3개로, 거대한 티틴(titin)의 경우 365개의 구간으로 분할되어 있다. 분할하여 유전자를 갖고 있으면 세포는 여러 가지 방법으로 유전자의 스플라이싱(연결)을 할 수 있다는 큰 이점이 있다. 동일한 DNA 배열을 사용해 조금씩 기능이 다른 다양한 단백질을 만들어낼 수 있는 것이다.
스플라이싱 반응
스플라이소좀(spliceosome)은 유전자로부터 만들어진 긴 전령 RNA 전구체 분자를 잘라 다시 연결하여 단백질을 만드는 데 사용되는 보다 짧은 전령 RNA 분자를 만들어낸다. 이는 많은 부분으로 이루어진 복잡한 분자 기계로, 단백질 사슬과 몇 개의 작은 RNA 사슬이 포함되어 있다. 복합체는 단계적인 방법으로 만들어져 있고, 스플라이싱의 각 반응 단계마다 서브유닛의 부가나 제거가 이루어진다. 먼저, 연결하는 두 부위가 인식되어 가까이 모아진다. 다음으로, 한쪽 스플라이스 부위가 절단되고 말단이 인트론 내에 있는 특별한 아데닌 뉴클레오타이드에 연결되어 루프 구조가 생긴다. 마지막으로 다른 쪽 스플라이스 부위가 절단되어 인트론 루프가 방출되고 엑손의 말단끼리 연결된다.
동적인 구조
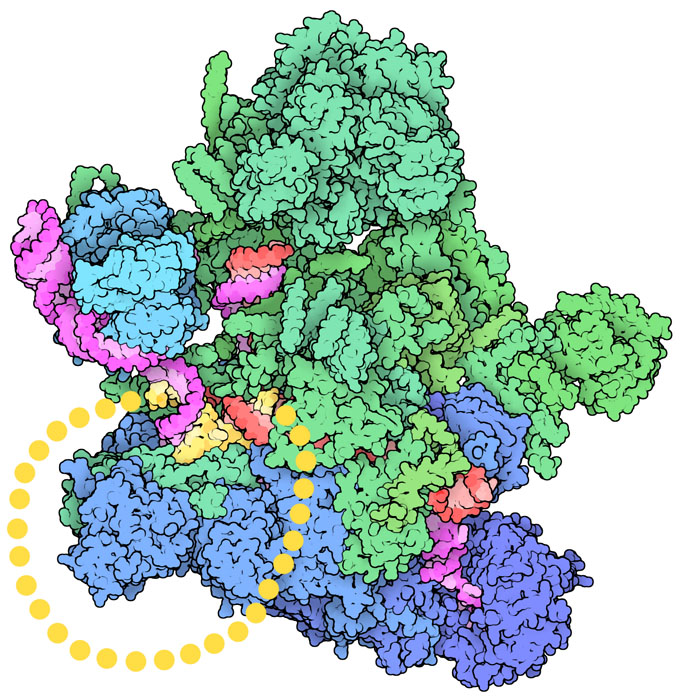
스플라이소좀이 수행하는 반응의 다양한 단계를 조사하는 데 저온전자현미경(cryoelectron microscopy)이 사용되고 있다. 스플라이소좀은 매우 동적인 기계이므로 복합체의 안정적인 부분밖에 구조가 밝혀지지 않았다. 그렇기 때문에 지금 보다시피 동적이고 관찰할 수 없는 부분의 구조는 상상으로 보완할 필요가 있다. 여기에 나타낸 구조는 PDB 엔트리 3jb9로부터 얻어진 것으로 스플라이싱의 최종 단계에 있는 스플라이소좀의 모습을 포착하고 있다. 연결된 엑손이 풀려나고 루프형 광유도 분자 올가미(lariat)이라 불리는 인트론이 아직 연결된 상태다.
RNA의 역할
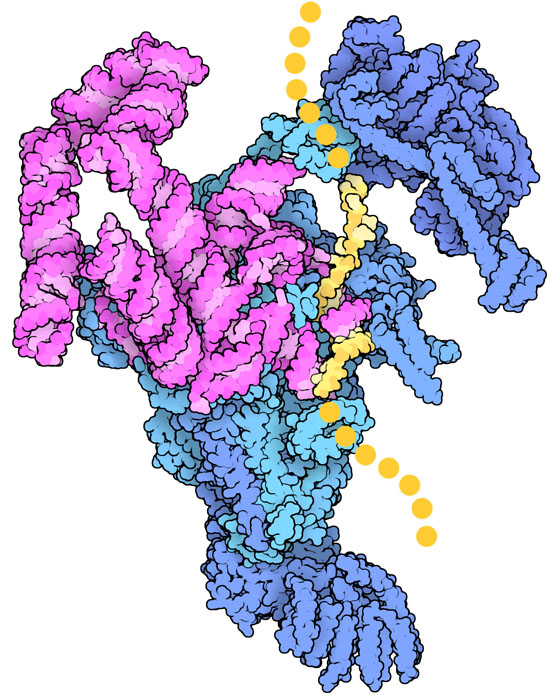
스플라이소좀이 실행하는 상세한 작업의 대부분은 작은 RNA 사슬에 의해 실행되고 있다. 이 구조(PDB 엔트리 6n7p)는 극히 초기의 반응 단계를 포착한 것으로, 스플라이싱 부위 1개가 인식된 시점이다. 스플라이소좀의 RNA는 전령 RNA 전구체와 대조하여 연결하는 부분의 배열을 읽는다. 그리고 또 다른 단백질과 RNA의 서브유닛이 이러한 복합체 주위에 모여 스플라이소좀 전체가 만들어진다. 이들 RNA 사슬은 2개의 마그네슘 이온의 도움을 받아 전령 RNA를 자르고 붙이는 반응도 수행한다. 놀랍게도 자가스플라이싱 RNA(self-splicing RNA) 중에는 이와 비슷한 구조를 사용하여 스플라이소좀의 도움 없이 자기 스스로 자르고 붙일 수 있는 것이 있다.
암의 연결고리
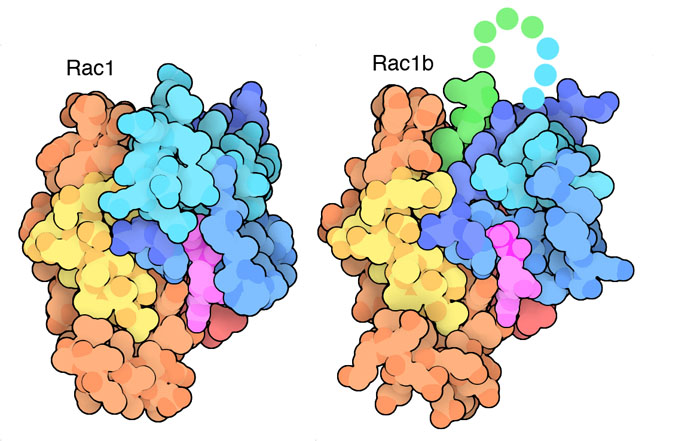
전령 RNA의 스플라이싱에 문제가 있으면 다양한 질병 발생으로 이어진다. 예를 들어 잘리고 붙여진 구간이 다른 신호 단백질 Rac1은 어떤 형태의 암에 대해 암 발병에 어떤 역할을 한다. Ras 단백질(Ras protein)과 마찬가지로 Rac1도 GTP와 GDP를 사용하여 ‘ON’과 ‘OFF’를 전환하는 분자 스위치로 작용하여 세포의 성장과 증식을 제어하는 신호를 전달하고 있다. Rac1은 일반적으로 6개의 엑손으로 구성되어 있다(여기에 나타낸 것은 PDB 엔트리 1mh1). 한편 사슬 중간에 여분의 엑손이 추가된 다른 형태(PDB 엔트리 1ryf)에는 단백질을 보다 지속적으로 ‘ON’ 상태로 만드는 효과가 있다. 암세포에서 Rac1b가 과도하게 발현되면 일반적인 제어를 당하지 않고 증식하는 데 도움이 된다
구조 보기
인터랙티브 조작이 가능한 페이지로 전환하려면 그림 아래의 버튼을 클릭하십시오. 읽기가 시작되지 않을 때는 그림을 클릭하십시오.
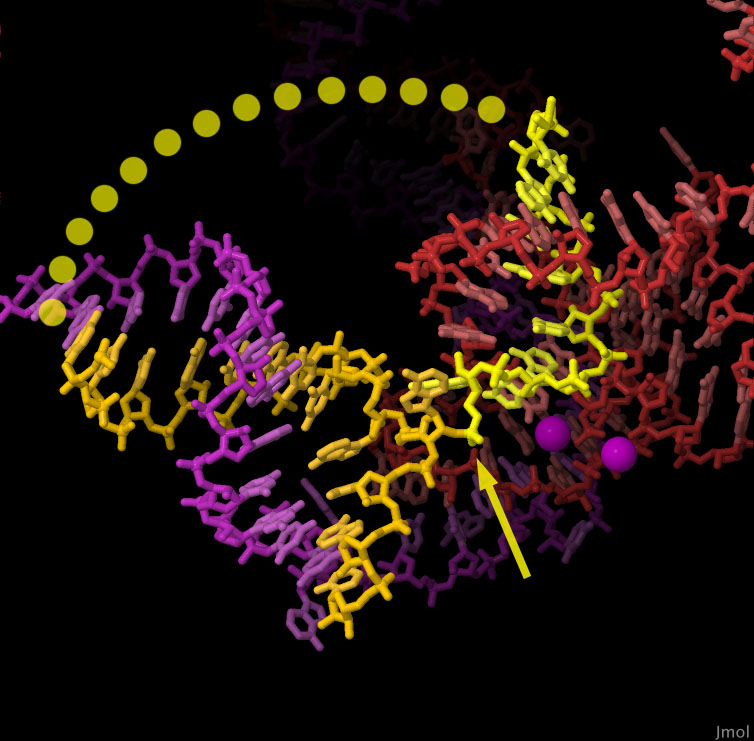
여기에 나타낸 스플라이소좀의 활성 부위를 확대한 그림은 인트론의 ‘광유도 분자올가미’(노란색)의 말단이 어떻게 스플라이소좀 안에 있는 2개의 작은 RNA 분자(자홍색과 빨간색)에 잡혀 있는지를 나타내고 있다. 인트론은 고리를 만들고, 그 말단은 인트론의 도중으로 되돌아가 RNA의 2’ 위치에 있는 하이드록실기(화살표로 표시된 부분)에 부가되어 있다. 도시된 이전의 반응 단계에서 촉매로서 작용하는 2개의 마그네슘 이온은 RNA 분자 근처에 자홍색 구로 표시된다. 이 구조를 더 자세히 보려면 그림 아래의 버튼을 클릭하여 인터랙티브 조작이 가능한 이미지로 전환해 보기 바란다.
이해를 높이기 위한 토픽
- 예를 들어 GenBank 등의 데이터베이스를 사용하여 티틴 유전자에서는 어떻게 엑손과 인트론이 배치되어 있는지 볼 수 있습니다.
참고문헌
- 6n7p 2019 A unified mechanism for intron and exon definition and back-splicing. Nature 573 375-380
- 2019 RNA splicing by the spliceosome. Ann. Rev. Biochem. 89 1.1-1.30
- 2017 Cryo-electron microscopy snapshots of the spliceosome: structural insights into a dynamic ribonucleoprotein machine. Nat. Struct.Mol. Biol. 10 791-799
- 3jb9 2015 Structure of a yeast spliceosome at 3.6-angstrom resolution. Science 349 1182-1191
- 1ryf 2004 Alternative Splicing of Rac1 Generates Rac1b, a Self-activating GTPase. J.Biol.Chem. 279 4743-4749
- 1mh1 1997 The crystal structure of human rac1, a member of the rho-family complexed with a GTP analogue. Nat.Struct.Mol.Biol. 4 147-152
- 2010 The pathobiology of splicing. J. Pathol. 220 152-163
- 1994 Nobel Lecture: Split genes and RNA splicing. Cell 77 805-815
 의 생체 고분자 학습 포털 사이트
의 생체 고분자 학습 포털 사이트 









