229: 蛍光RNAアプタマー(Fluorescent RNA Aptamers)
著者: David S. Goodsell
翻訳: 工藤 高裕(PDBj)
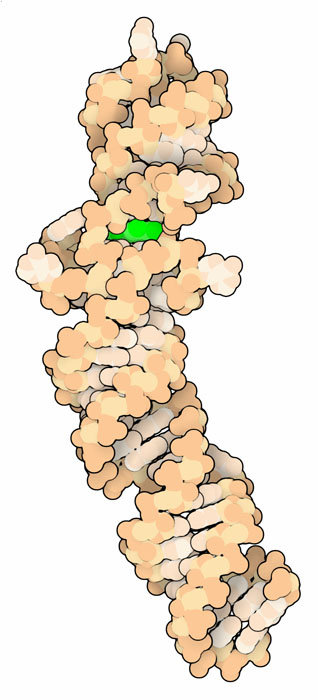
蛍光アプタマー「ホウレンソウ」。RNAは明るいオレンジ色で、蛍光体は緑色で示す。
細胞の中をより詳しく調べる新たな道具を探す試みは常に続けられている。緑色蛍光タンパク質(green fluorescent protein、GFP)は全く新たな分野を切り開いた道具の一例である。これを使えばある特定のタンパク質に印をつけることができ、生きた細胞の中でそのタンパク質が何をしているのかをみることができる。最近、これと似た方法でRNAをみる新たな道具の開発が進められている。RNA自体は蛍光を発しないので、蛍光体(fluorophore、小さな蛍光分子)に結合してその蛍光を強める短いRNAををつくるという手法が用いられた。このRNAはリボソーム(ribosome)のような天然のRNAに組み込むことができる。蛍光体を細胞に加えると、改変型リボソームに結合し、それを私たちは観察できるようになる。
アプタマーの獲得
SELEX(Systematic Evolution of Ligands by EXponential enrichment、急速増幅法によるリガンドの計画的進化) はこのような役に立つ蛍光体結合性RNA分子を見つけるのに使われてきた。この手法では、まずある蛍光体にさまざまなRNA配列を混ぜ、次にそれと結合するものを単離する。そしてランダムに修飾を加え、その中から最もよいものを選び出す。その後、修飾と選別を何度も繰り返し、蛍光体に結合して蛍光を増強するアプタマー(aptamer)を見つけ出す。ここに示すのはホウレンソウ(spinach)と呼ばれているアプタマーで、緑色蛍光タンパク質の中にあるものと似た蛍光体を利用しこの過程を使って発見された。
構造解析への挑戦
ホウレンソウアプタマーは長いヘアピン型の構造をもち、中心には複雑に折りたたまれた部分がある。これが蛍光体を取り囲んでいる。RNAの結晶化には常に困難がつきまとうので、構造決定には2つの手法が使われた。PDBエントリー4kzd(ここに図示する構造)では、末端のループ部分に抗体が結合できるようにする改変が加えられており、これが安定な結晶格子の形成を促している。PDBエントリー4ts2(以下に対話的操作のできる画像で示している構造)では、端から端まで分子を詰め込んだ結晶をつくりやすくなるようループ部分が切り離されている。
いろんな色のアプタマー
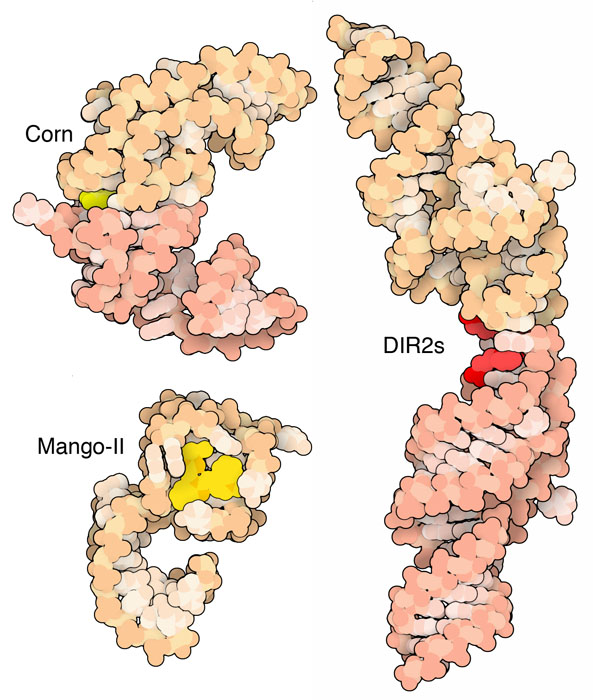
3つの蛍光アプタマー。RNAは薄いオレンジ色やピンク色で、蛍光体は濃い黄色やピンク色で示す。
他の蛍光アプタマーも見つかっていて虹の各色がそろっているが、これは非常に役に立つ。なぜなら、一つの細胞にある異なる種類のRNA分子をそれぞれ別々の色を発する蛍光アプタマーで印をつけておくと、それらを色の違いで識別できるからである。また、FRET(Fluorescence Resonance Energy Transfer、蛍光共鳴エネルギー移動)のような技術を使うと、生きた細胞の中にある異なる蛍光体間の距離を追跡できる。ここに3つの例、黄色のコーンアプタマー(Corn aptamer、PDBエントリー5bjp)、オレンジ色のマンゴー-IIアプタマー(PDBエントリー6c63)、赤色のDIR2sアプタマー(PDBエントリー6db8)を示す。
構造をみる
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
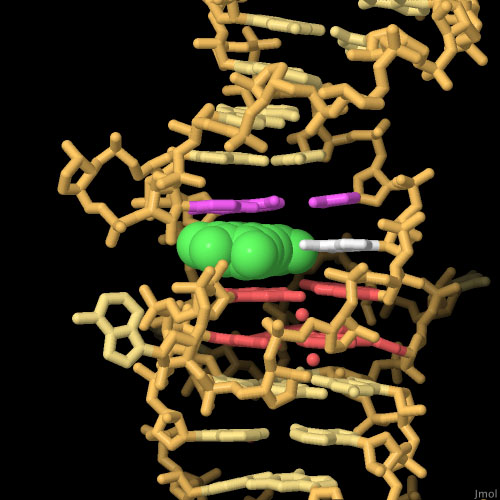
ホウレンソウアプタマーは蛍光体を取り囲み、分子の蛍光を強める丈夫なくぼみをつくる。蛍光体の一方の側はグアニン四重鎖(G-quadruplex、ピンク色の部分)に対して詰め込まれ、もう一方の側は三つ組のヌクレオチド塩基(赤紫色の部分)でおおわれている。さらにグアニン(白色の部分)は蛍光体の端と相互作用し、くぼみの中に配置されている。
理解を深めるためのトピックス
-
ホウレンソウアプタマーが結晶格子に詰め込まれている様子をみるには、RCSB PDBサイトの構造メインページにアクセスし、Assembly(集合体)メニューにある「Unit Cell」または「Supercell」を選んでみてください。
-
構造をみるとお分かりのように、ほとんどのアプタマーは蛍光体に対しグアニン四量体が詰め込まれています。どのようにRNA鎖が折りたたまれ4つのグアニンが正しい位置に配置されるのかを追ってみてください。
参考文献
-
Trachman, R. J., Truong, L., Ferré-D’Amaré, A. R.
2017
Structural principles of fluorescent RNA aptamers.
Trends Pharmacol. Sci.
38
928-939
-
6db8
Shelke, S.A., Shao, Y., Laski, A., Koirala, D., Weissman, B.P., Fuller, J.R., Tan, X., Constantin, T.P., Waggoner, A.S., Bruchez, M.P., Armitage, B.A., Piccirilli, J.A.
2018
Structural basis for activation of fluorogenic dyes by an RNA aptamer lacking a G-quadruplex motif.
Nat. Commun
9
4542-4542
-
6c63
Trachman 3rd., R.J., Abdolahzadeh, A., Andreoni, A., Cojocaru, R., Knutson, J.R., Ryckelynck, M., Unrau, P.J., Ferré-D'Amaré, A.R.
2018
Crystal structures of the Mango-II RNA aptamer reveal heterogeneous fluorophore binding and guide engineering of variants with improved selectivity and brightness.
Biochemistry
57
3544-3548
-
5bjp
Warner, K.D., Sjekloca, L., Song, W., Filonov, G.S., Jaffrey, S.R., Ferré-D'Amaré, A.R.
2017
A homodimer interface without base pairs in an RNA mimic of red fluorescent protein.
Nat. Chem. Biol.
13
1195-1201
-
4kzd
Huang, H., Suslov, N.B., Li, N.S., Shelke, S.A., Evans, M.E., Koldobskaya, Y., Rice, P.A., Piccirilli, J.A.
2014
A G-quadruplex-containing RNA activates fluorescence in a GFP-like fluorophore.
Nat. Chem. Biol.
10
686-691
-
4ts2
Warner, K.D., Chen, M.C., Song, W., Strack, R.L., Thorn, A., Jaffrey, S.R., Ferré-D'Amaré, A.R.
2014
Structural basis for activity of highly efficient RNA mimics of green fluorescent protein.
Nat. Struct. Mol. Biol.
21
658-663

 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト