207: 光活動性黄色タンパク質(Photoactive Yellow Protein)
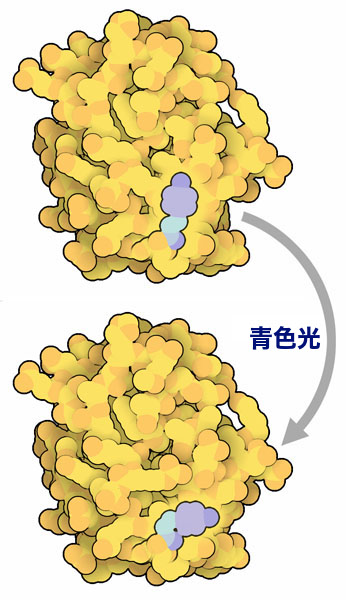
タンパク質をみるとき、その動きの大半はきわめて小さく動作時間も短い。これは光に関係する分子反応のとき特に言えることである。この小さくて速い過程をみるためのより感度の良い実験方法が発明されてきた。光活動性黄色タンパク質(photoactive yellow protein、PYP)はそのような研究の対象とされてきたタンパク質の一つである。光活動性黄色タンパク質は紅色硫黄細菌にみられる小さな水溶性タンパク質で、青色光を感知する。大きくてよく回折する安定な結晶をつくるので研究対象として完璧であり、これを使うと原子レベルで何が起こっているのかを知ることができる。
光の検知
フェムト秒※1レベルの非常に速い速度で始まり約1秒で終わる光活動性黄色タンパク質の光サイクルには、分かれた数多くの連続ステップが関わっている。分子中の発色団が青色光を吸収し、分子はほぼまっすぐな配置(トランス型)から曲がった配置(シス型)へと変化する。この変化には1ピコ秒※1もかからない。この配向変化によって、細菌の泳ぐ方向を制御する他の光感知タンパク質が活性化すると考えられている。最後に、発色団は約0.5秒で元のまっすぐな型に戻り、次の青色光子を感知する準備が整う。
感知器をみる
光によって活性化された状態は1秒も続かないので、結晶構造解析で観察するのは難しくラウエ回折(Laue diffraction)のようなうまい方法を使う必要がある。通常、結晶構造解析では単一波長のX線が使われ、結晶を回転してデータを集め、多くの回折パターンを観察する。一方ラウエ法では、静止した結晶に対しシンクロトロンから出た多波長で構成されるX線を一度に照射する。これによりはるかに複雑な回折パターンが生成される。これには露光中得られたすべての回折斑点がこのパターンの中に含まれていて、一連のデータをより素早く得ることができる。ここに示す光活動性黄色タンパク質の構造(PDBエントリー2pyp)は、レーザーからの青色光を結晶に照射し、数ミリ秒後に複数波長のX線を短時間一気に照射してデータを集め決定されたものである。こうして、発色団とそれを取り囲むタンパク質が光を吸収した後どのように動くのかをみることができるようになった。
※1:この種の分子遷移に関することを議論するとき、より短い時間を表現するメートル法の用語がよく使われる。
- ミリ秒(millisecond)=1000分の1秒
- マイクロ秒(microsecond)=100万分の1秒
- ナノ秒(nanosecond)=10億分の1秒
- ピコ秒(picosecond)=1兆分の1秒
- フェムト秒(femtosecond)=1000兆分の1秒
水素をみる
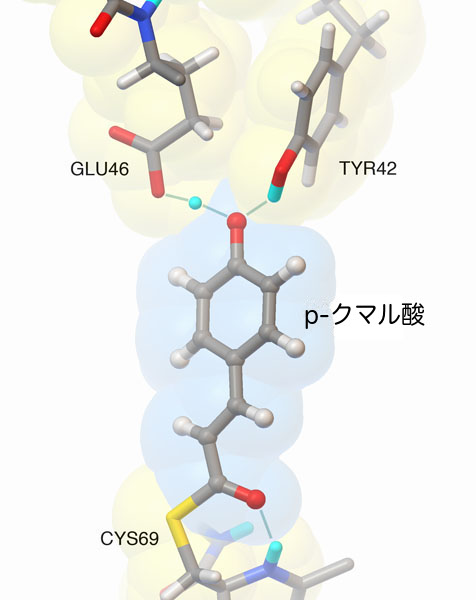
光活動性黄色タンパク質のX線結晶解析構造から、発色団(p-クマル酸、4'-ヒドロキシケイ皮酸とも呼ばれる)がタンパク質分子の中にしっかりとらえられていることが分かった。一端はシステインと共有結合し、もう一方の端では2つのアミノ酸との間に通常より短い水素結合を形成している。この水素結合をより詳しくみるため、視点の異なる2つの手法〜X線回折と中性子回折〜が使われた。X線結晶解析では分子構造の中で電子のある位置が見えるが、これは炭素、窒素、酸素などの重い原子の位置決定に役立つ。これは水素結合の距離測定にも有用で、光活動性黄色タンパク質における水素結合はPDBに登録されている構造の中で最も短いことがわかった。しかしほとんどの場合、X線結晶解析で水素は直接観察できない。一方、中性子は原子の核により回折するので、水素原子の位置情報も得られる。光活動性黄色タンパク質の中性子回折によって得られた構造(PDBエントリー2zoi)から、水素原子が発色団とグルタミン酸で共有された非常にまれな配置になっていることが明らかになった。この珍しい相互作用は、発色団の中にある電荷を持った酸素原子を安定化させるのに役立っているという説が出されている。
構造をみる
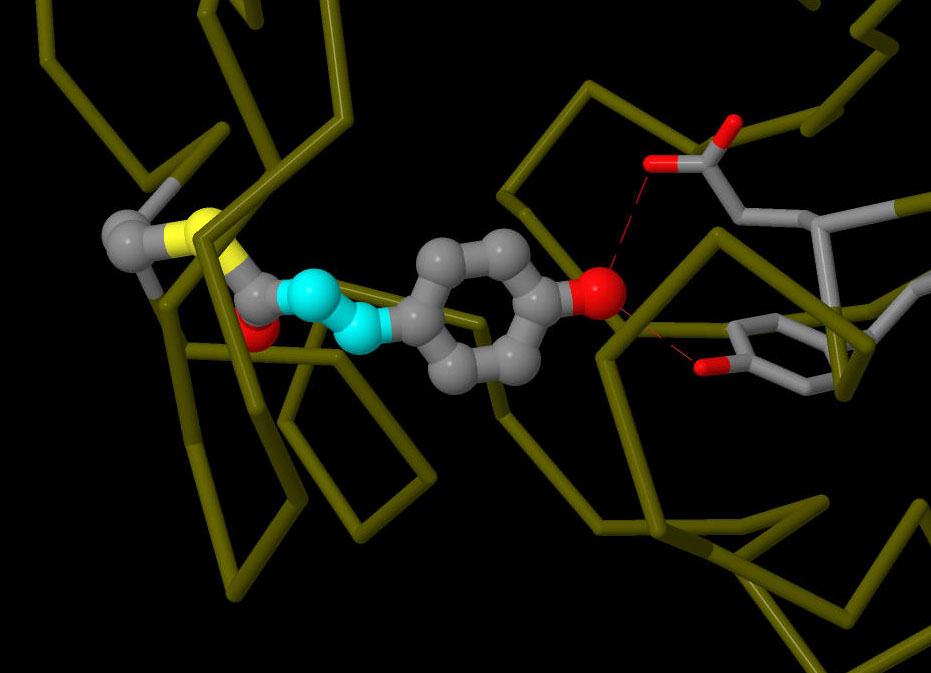
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
タンパク質の分子構造をより速くみる方法の開発が進められている。今のところ最も速いのは連続フェムト秒結晶構造解析(serial femtosecond crystallography、SFX)という方法である。X線自由電子レーザー(X-ray Free Electron Laser: XFEL)から出た非常に強力なX線の閃光をタンパク質の小さな結晶に当てると、その瞬間の回折パターンが生成され、この過程で結晶は燃え尽きてしまう。この方法を使って、発色団が光を吸収した後にトランス型からシス型に変化する瞬間がとらえられた。ここに示す構造は、照射後100〜400フェムト秒後の状態をとらえたものである。画像の下のボタンをクリックすると、この驚くべき構造の動画をみることができる。
理解を深めるためのトピックス
- 「連続フェムト秒結晶構造解析」(serial femtosecond crystallography)で検索してみると、同じ手法で研究された他の吸光タンパク質をみることができる。この中にはバクテリオロドプシン(bacteriorhodopsin)や光化学系 I・II(photosystem I・II)などが含まれる。
- この他の感覚性タンパク質の多くが持っているドメインは光活動性黄色タンパク質が持つドメインと似ていて、これらをまとめてPASドメインと呼んでいる。例えば、光活動性黄色タンパク質とフォトトロピン(phototropin)の光感知ドメイン(PDBエントリー2z6c)との構造を比較してみて欲しい(RCSB PDB)。
参考文献
- 5hd3、5hdc、5hdd、5hds、5hd5 2016 Femtosecond structural dynamics drives the trans/cis isomerization in photoactive yellow protein. Science 352 725-729 DOI:10.1126/science.aad5081 PMID:27151871 PMC:PMC5291079
- 4b9o 2012 Watching a signaling protein function in real time via 100-ps time-resolved Laue crystallography. Proceedings of the National Academy of Science USA 109 19256-19261 DOI:10.1073/pnas.1210938109 PMID:23132943 PMC:PMC3511082
- 2zoi 2009 Low-barrier hydrogen bond in photoactive yellow protein. Proceedings of the National Academy of Science USA 106 440-444 DOI:10.1073/pnas.0811882106 PMID:19122140 PMC:PMC2626721
- 1ts0 2005 Visualizing reaction pathways in photoactive yellow protein from nanoseconds to seconds. Proceedings of the National Academy of Science USA 102 7145-7150 DOI:10.1073/pnas.0409035102 PMID:15870207 PMC:PMC1088170
- 2004 Short hydrogen bonds in photoactive yellow protein. Acta Crystallographica D60 1008-1016 DOI:10.1107/S090744490400616X PMID:15159559
- 2pyp 1997 Structure of a protein photocycle intermediate by millisecond time-resolved crystallography. Science 275 1471-1475 DOI:10.1126/science.275.5305.1471 PMID:9045611
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









