177: アポトソーム(Apoptosomes)
「生きるべきか死ぬべきか」この質問は、私たちの各細胞で常に問いかけられている。細胞は指令があると死ぬようプログラムされている。この仕組みは私たちのように大きな生物の発生において不可欠なものである。細胞は協調して成長したり、死んだりして完全な体を作っていく。大人にとっても、損傷したり病気に感染したりがんになったりした細胞を取り除く上でこの仕組みは必要である。細胞死に関わる機械は常に細胞内にあって静かに待機しているが、死が選択されるとすぐに発動することができる。
門番
プログラムされた細胞死の過程はアポトーシス(apoptosis)とも呼ばれており、高度に制御され、死ぬべきかどうかの決断は多くの分子が連携することにより行われている。この過程のうち内因性経路(intrinsic pathway)と呼ばれる主要過程の一つでは、アポトソーム(apoptosome)が門番の役割を果たしている。アポトソームは問題を検知する分子と、死ぬべきという選択がなされた細胞を解体する分子との間にある。通常、構成する多くのサブユニットは分離され、不活性な状態で何かを傷つけることなく細胞内を動き回っている。しかし問題が起こると、集まって星形の複合体を形成し、タンパク質を切断するカスパーゼ(caspase)を活性化してアポトーシスを開始する。
シトクロムcによる誘発
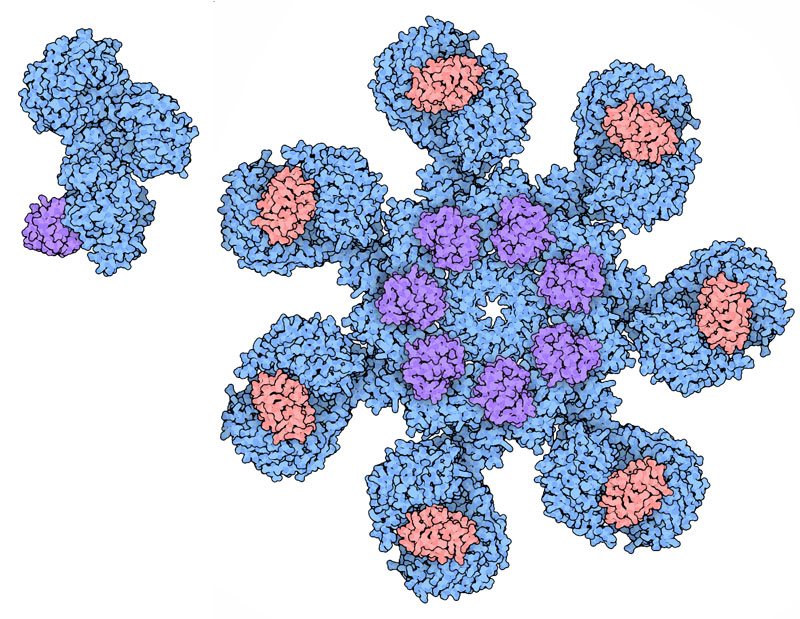
私たちの細胞内において、アポトソームを活性化する信号となるのは、ミトコンドリアからのシトクロムc(cytochrome c)放出である。シトクロムc(赤)が別のタンパク質APAF-1(青・紫)と結合し、集まって7回対称の環状構造を作る。そして、シトクロムcはアポトソームのCARDドメイン(紫)と結合して活性化される。なおここに示すアポトソーム(PDBエントリー 3j2t、上図右)の構造は、EMデータバンクのエントリー[[EMDB:5186]]に収められている電子顕微鏡のマップ情報を利用して解かれたものである。ヒトのアポトソームの場合、CARDドメインは柔軟なリンカー部分を介してタンパク質の残りの部分とつながっているが、このPDBエントリーにCARDドメイン部分は含まれていない。ここではCARDドメイン(PDBエントリー 2p1h)の結晶構造を用いておおよその位置を示している。また上図左に示すのは、不活性なApaf-1単量体(PDBエントリー 1z6t、3sfz)の構造である。
アポトソーム
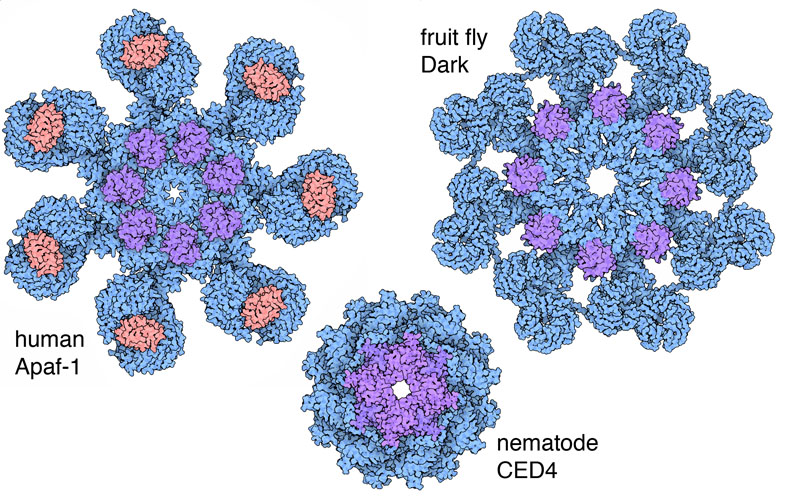
他の生物が持つアポトソームと見比べてみると、構造やどのようにして働くかについて大きな違いがあることが分かる。ヒトのアポトソームは7つのサブユニットで構成されており、シトクロムcによって活性化される。DARKと呼ばれているショウジョウバエ由来のもの(PDBエントリー 3iz8)は、8つのサブユニットで構成されていて、活性化するのにシトクロムcが必要かどうかは分かっていない。CED-4と呼ばれている線虫由来のもの(PDBエントリー 3lqq)は、上記のものよりずっと小さくシトクロムcと結合する部位を欠いている。カスパーゼはCARDドメイン側(図の手前側)ではなくその裏側に結合すると考えられている。
構造をみる
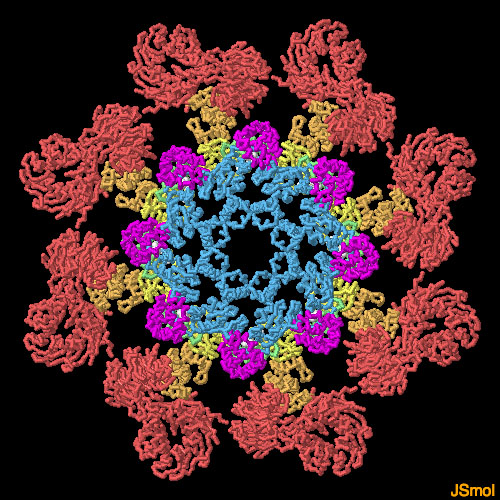
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
アポトソームはいくつかの機能部位で構成されていて、それぞれ異なる機能を担っている。ここに示すのはショウジョウバエのアポトソームで、各ドメインを異なる色で表現している。鎖の一端にはカスパーゼとの相互作用に関係するCARDドメイン(赤紫)と、複合体が集合するのを助けるヌクレオチド結合ドメイン(青)がある。もう一方の端にはシトクロムcをつかみ、複合体形成を開始させるβプロペラドメイン(赤)が2つある。これらの間には両端のドメインが行う活動を連携させるいくつかの小さなドメイン(緑、黄色)がある。図の下にあるボタンをクリックして対話的操作のできる画像に切り替え、詳しく見てみて欲しい。
理解を深めるためのトピックス
- 線虫の不活性型アポトソームは、2つのサブユニット(CED-4)に制御タンパク質(CED-9)が結合してできています。この構造をPDBエントリー 2a5y で見ることができます。
- カスパーゼの構造が多数PDBに登録されています。まずは今月の分子 - カスパーゼで扱っているエントリーから見てみてください。
参考文献
- 2013 Apoptosome structure, assembly, and procaspase activation. Structure 21 501-515 10.1016/j.str.2013.02.024
- 3j2t 2013 Changes in Apaf-1 conformation that drive apoptosome assembly. Biochemistry 52 2319-2327 10.1021/bi301721g
- 3iz8 2011 Structure of the Drosophila apoptosome at 6.9 A resolution. Structure 19 128-140 10.1016/j.str.2010.10.009
- 3sfz 2011 Crystal structure of full- length Apaf-1: how the death signal is relayed in the mitochondrial pathway of apoptosis. Structure 19 1074-1083 10.1016/j.str.2011.05.013
- 3lqq 2010 Crystal structure of the Caenorhabditis elegans apoptosome reveals an octameric assembly of CED-4. Cell 141 446-457 10.1016/j.cell.2010.03.017
- 2p1h 2007 Rapid folding and unfolding of Apaf-1 CARD. Journal of Molecular Biology 369 290-304 10.1016/j.jmb.2007.02.105
- 1z6t 2005 Structure of the apoptotic protease-activating factor 1 bound to ADP. Nature 434 926-933 10.1038/nature03465
代表的な構造
- 3j2t: ヒトのアポトソーム
- アポトソームはプログラム細胞死(アポトーシス)の過程でカスパーゼを活性化する。この構造は、APAF-1サブユニットが7つ集まったものにシトクロムcが結合してできた活性型アポトソームで、これが細胞にアポトーシスを開始させる信号となる。
- 3iz8: ショウジョウバエのアポトソーム
- アポトソームはプログラム細胞死(アポトーシス)の過程でカスパーゼを活性化する。この構造は、DARKと呼ばれるタンパク質のサブユニットが8つ集まってできたショウジョウバエのアポトソームの構造である。
- 3lqq: 線虫のアポトソーム
- アポトソームはプログラム細胞死(アポトーシス)の過程でカスパーゼを活性化する。この構造は、CED-4と呼ばれるタンパク質のサブユニットが8つ集まってできた線虫のアポトソームの構造である。
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









