187: ニューデリー メタロβラクタマーゼ(New Delhi Metallo-β-Lactamase)
ペニシリン(penicillin)などの抗生物質を発見したことは、人類の歴史にとって画期的な出来事であった。しかし、この発見から数年のうちに細菌は反撃を開始し、抗生物質に抵抗性を示す細菌種が出現し始めた。その後何十年か経過した現在も、これらの抵抗性細菌に対抗する新たな抗生物質を探すための医学研究が続けられている。この努力は、天然物の探索と化合物の人工設計の両方を使って進められている。
超強力細菌
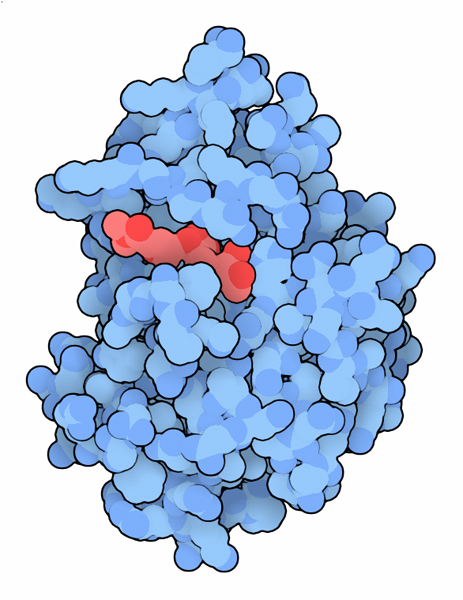
近年、私たちは投与した薬剤を全て破壊してしまう高抵抗性細菌に直面している。最も危険な菌株が南アジアで出現し、最近ロサンゼルス地域医療センターで致命的な感染拡大が発生した。これらの細菌はNDM-1(new delhi metallo-β-lactamase、ニューデリー メタロβラクタマーゼ)という酵素を使って、幅広い種類のペニシリン様抗生物質を不活性化する。ここに示す構造(PDBエントリー 4eyl)は、高度先進型のカルバペネム系抗生物質(carbapenem antibiotic)を分解した直後の酵素である。
情報の共有
多剤耐性菌の薬剤分解酵素を他の細菌と共有できてしまうことが、事態をより危険なものにしている。これら酵素の設計指示書は、プラスミド(plasmid)と呼ばれる二重鎖の小さな環状DNAで運ばれていることがよくあり、これを使って細胞から細胞へと簡単に伝えることができる。例えば、ある新たな細菌がNDM-1の遺伝子を含むプラスミドを取り込むと、広い範囲の抗生物質に対する耐性を獲得することになる。このぞっとする現実により、新たな別のものを対象とする抗生物質の探索を強いられている。なぜなら、この超強力細菌は私たちが用意した最強のペニシリン様βラクタムでも止めることはできないからである。
様々な攻撃手法
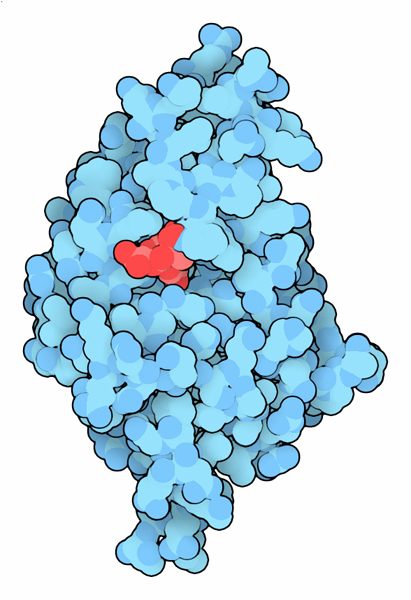
抗生物質抵抗性細菌はこのような抗生物質を破壊するのに様々な手法を用いる。NDM-1は、反応の際2つの亜鉛(zinc)原子を使う金属酵素である。最近、肺炎桿菌(Klebsiella pneumoniae)の系統でカルバペネム系抗生物質に対しても強い抵抗性を持つものが出てきている。この細菌は全く異なる方法を使っていて、特に反応性の高いセリンアミノ酸で抗生物質を攻撃する。だが私たちも反撃を仕掛けているところである。ここに示す構造(PDBエントリー 3rxw)には、このセリンに結合して細菌の酵素を不活性化する薬の試作品が含まれている。
構造をみる
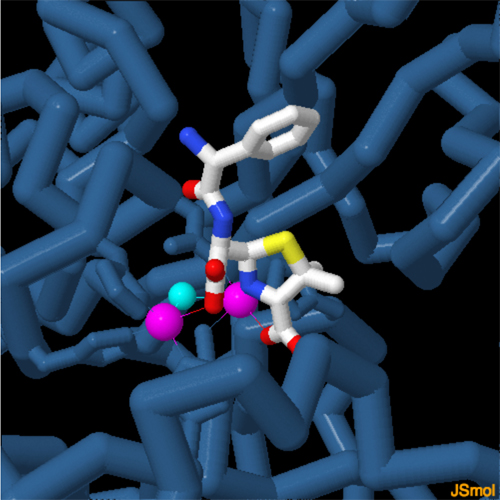
対話的操作のできるページに切り替えるには図の下のボタンをクリックしてください。読み込みが始まらない時は図をクリックしてみてください。
NDM-1は通常の酵素のように対象基質が特異的ではないので、特に効果的である。ほとんどの酵素は基質を取り囲み、特定の対象分子だけを認識する。一方NDM-1の場合、2つの亜鉛イオンを使って抗生物質の重要な活性部位だけを認識し残りの大部分は無視する。こうしてほとんど全てのβラクタム系抗生物質(β-lactam antibiotics)を無効化することができる。上図は酵素が抗生物質のアンピシリン(ampicillin)に結合して切断した後の様子を示したもの(PDBエントリー 3q6x)である。図の下のボタンをクリックし、対話的操作のできる画像に切り替えると、この酵素が別の抗生物質(ペニシリン penicillin、セファロスポリン cephalosporin、カルバペネム carbapenem)と結合している様子も見ることができる。
理解を深めるためのトピックス
参考文献
- 4rl0、4rl2 2014 Structural and mechanistic insights into NDM-1 catalyzed hydrolysis of cephalosporins. Journal of the American Chemical Society 136 14694-14697 DOI:10.1021/ja508388e
- 3rxw 2012 Crystal structures of KPC-2 β-lactamase in complex with 3-nitrophenyl boronic acid and the penam sulfone PSR-3-226. Antimicrobial Agents and Chemotherapy 56 2713-2718 DOI:10.1128/AAC.06099-11
- 4exs, 4ey2, 4eyf, 4eyl, 4eyb 2012 New Delhi metallo-β-lactamase: structural insights into β-lactam recognition and inhibition. Journal of the American Chemical Society 134 11362-11365 DOI:10.1021/ja303579d
- 3se7 2011 Antibiotic resistance is ancient. Nature 477 457-461 DOI:10.1038/nature10388
- 3q6x 2011 Crystal structure of NDM-1 reveals a common β-lactam hydrolysis mechanism. The FASEB Journal 25 2574-2582 DOI:10.1096/fj.11-184036
- 1e4e 2000 The molecular basis of vancomycin resistance in clinically relevant Enterococci: crystal structure of D-alanyl-D-lactate lyase (VanA). Proceedings of the National Academy of Science USA 97 8921-8925 DOI:10.1073/pnas.150116497
 の生体高分子学習ポータルサイト
の生体高分子学習ポータルサイト 









